基因家族鉴定流程
以NLR基因家族流程鉴定为例
- 资料下载
首先在interpro网站(https://www.ebi.ac.uk/interpro/),Member DB中搜索NLR,并下载它全部的蛋白质
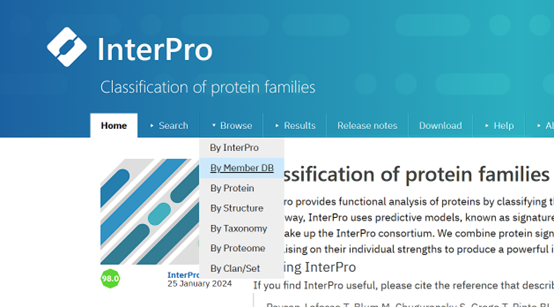
Fig 1 interPro html home
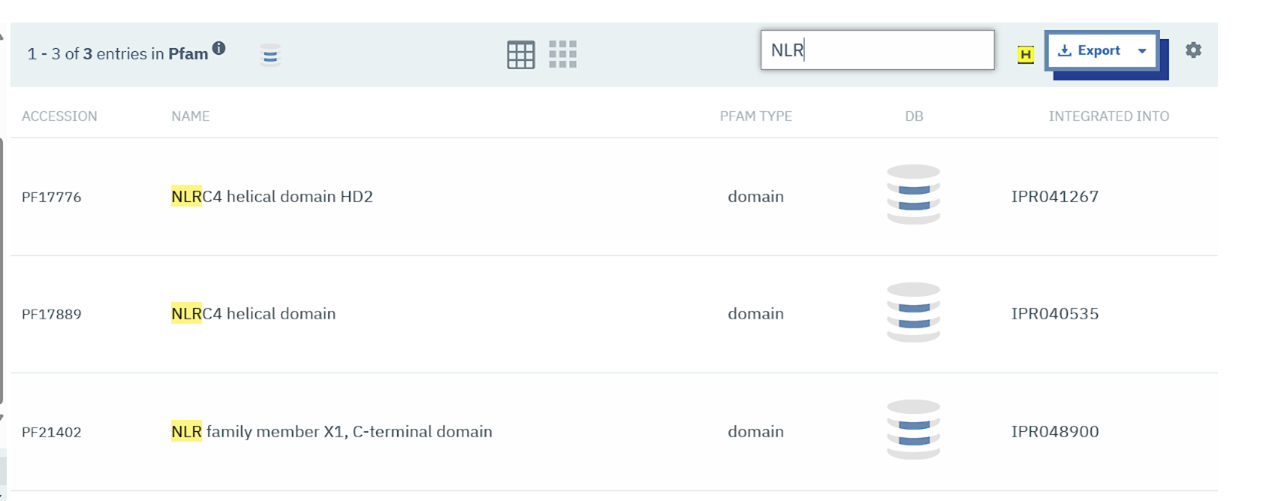
Fig2 NLR搜索结果

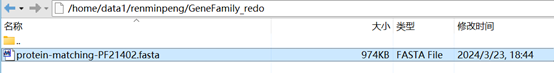
Fig3 下载fasta文件
下载完成后会得到一个名为protein-matching-PF21402.fasta的文件,将其上传至服务器指定文件夹内(自己分析文件处),本次示例存放在/home/data1/renminpeng/GeneFamily_redo路径下。
至此,资料下载部分介绍完毕。
2.
2.1使用hmmbuild构建.hmm文件
打开SecureCRT软件,切换路径:cd /home/data1/renminpeng/GeneFamily_redo/
然后运行以下代码:
mafft –auto –clustalout /home/data1/renminpeng/GeneFamily_redo/protein-matching-PF21402.fasta > /home/data1/renminpeng/GeneFamily_redo/Pfam21402.FBNs.align.clustal
应得到以下文件:

2.2文件格式转换
将上述生成文件上传至:https://sequenceconversion.bugaco.com/converter/biology/sequences/fasta_to_phylip.php
网站中,根据以下选项进行设置

应得到以下文件,将该文件上传至服务器指定文件夹内:

2.3hmmbuild构建文件
在SecureCRT软件中,输入以下命令:
hmmbuild Pfam21402.FBNs.hmm sample.stockholm
得到以下文件:

2.4 hmmsearch
将参考蛋白质序列,即NCBI下载的protein.fa文件上传至服务器内
输入以下命令:
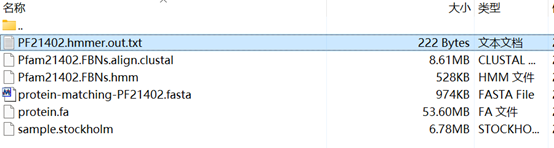
hmmsearch /home/data1/renminpeng/GeneFamily_redo/Pfam21402.FBNs.hmm /home/data1/renminpeng/GeneFamily_redo/protein.fa > /home/data1/renminpeng/GeneFamily_redo/PF21402.hmmer.out.txt
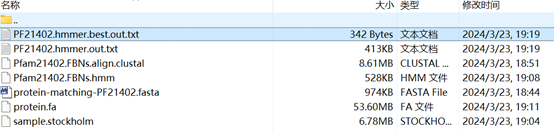
应得到以下文件:
输入筛选命令:
cat PF21402.hmmer.out.txt |grep -v “#” | awk ‘{if($4 < 1e-5 && $5 > 90) print $9}’ | sort | uniq | grep -v “+” > PF21402.hmmer.best.out.txt
应得到以下文件:
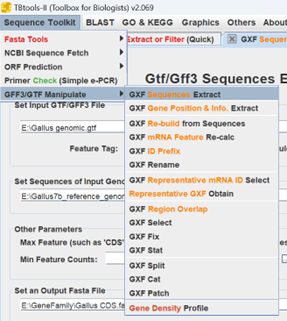
2.5TBtools提取参考基因组CDS序列
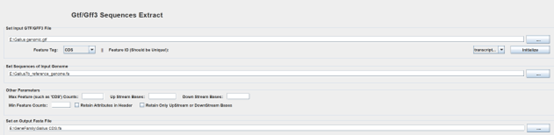
注意配套使用,即应用NCBI的参考基因组.fa和.gtf或者ensembl的.fa或.gft文件,不能交叉混用。
2.6翻译CDS序列
TBtools中Batch Translate CDS to Protein插件可以实现,输入上步得到的Gallus CDS.fa文件,输出Gallus CDS to protein.fa文件
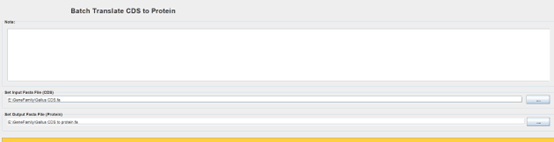
应得到以下文件:
2.7提取目的蛋白序列
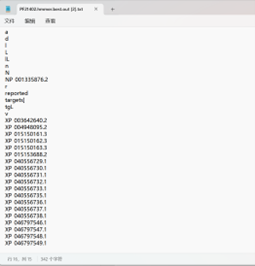
将2.4得到的目的基因蛋白ID,通过参考protein.fa进行提取,注意输出文件为.fa文件
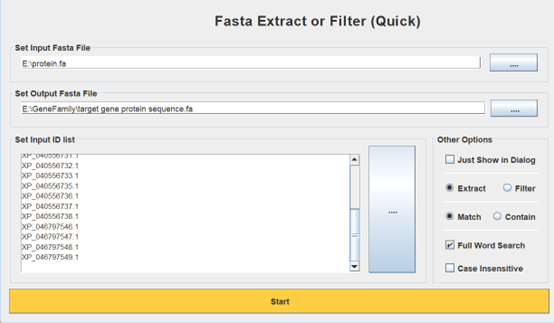
应得到以下文件:

2.8多序列比对
TBtools中 Blast Several Sequnences to a Big Database, 输入目标基因蛋白序列,和提取到目标物种的蛋白序列,进行比对,注意输出为Table制符表格式,以便后续Excel操作。
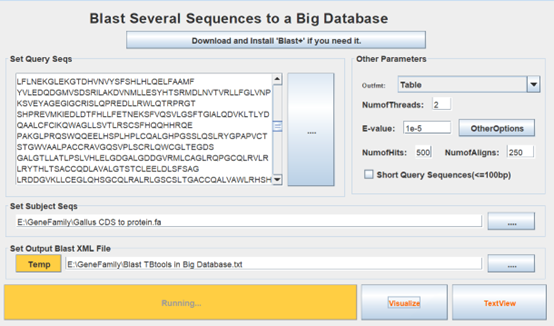
应得到以下文件:
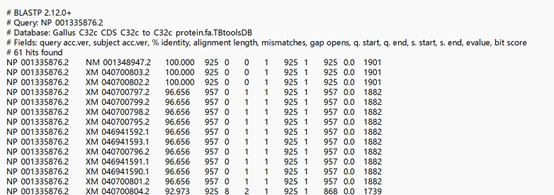
红色矩形圈出的为比对出的基因ID,可视情况进行筛选,将以上内容复制到excel中,提取所圈出列的内容。
2.9提取目标物种的NLR基因家族所有蛋白序列
在excel中,将重复的单元格进行删除。
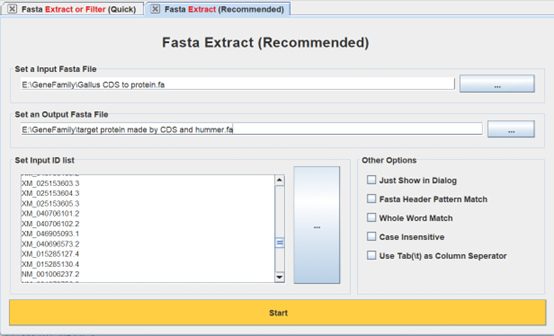
在TBtools中Fasta Extract,输入目标物种所有蛋白fasta文件(CDS to protein)和excel筛选好的基因名,设置输出结果。
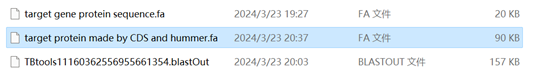
应得到以下文件:
3.
3.1基因家族Motif分析
首先打开MEME网页进行在线分析,将上步的得到的基因家族蛋白序列提交,motif设置为10进行分析。
MEME网站:https://meme-suite.org/meme/tools/meme

稍等片刻后,在结果页面点击MAST_XML_output, Ctrl+S进行保存

Tbtools打开Visualize MEME/MAST Motif Pattern工具,将保存好的mast文件拖入,设置好基因ID,点击start即可得到motif图片。
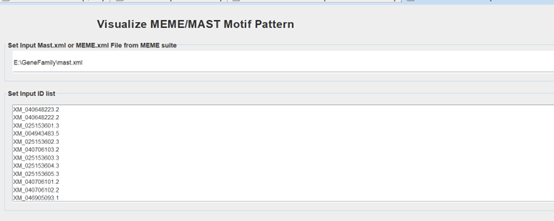
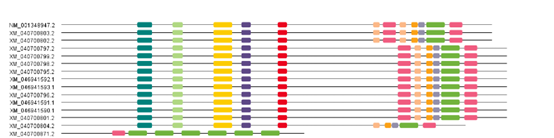
- 基因家族保守结构域分析
首先打开NCBI的Batch CDD 网页进行在线分析,将2.9得到的基因家族蛋白序列上传进行分析。
NCBI CDD网站:https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi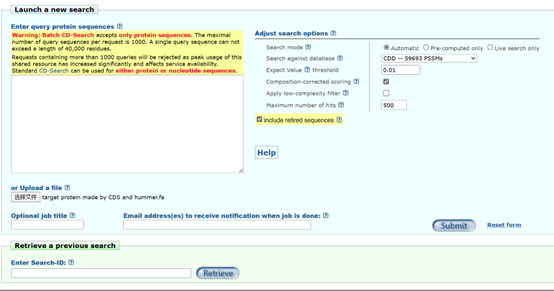 下载结果
下载结果
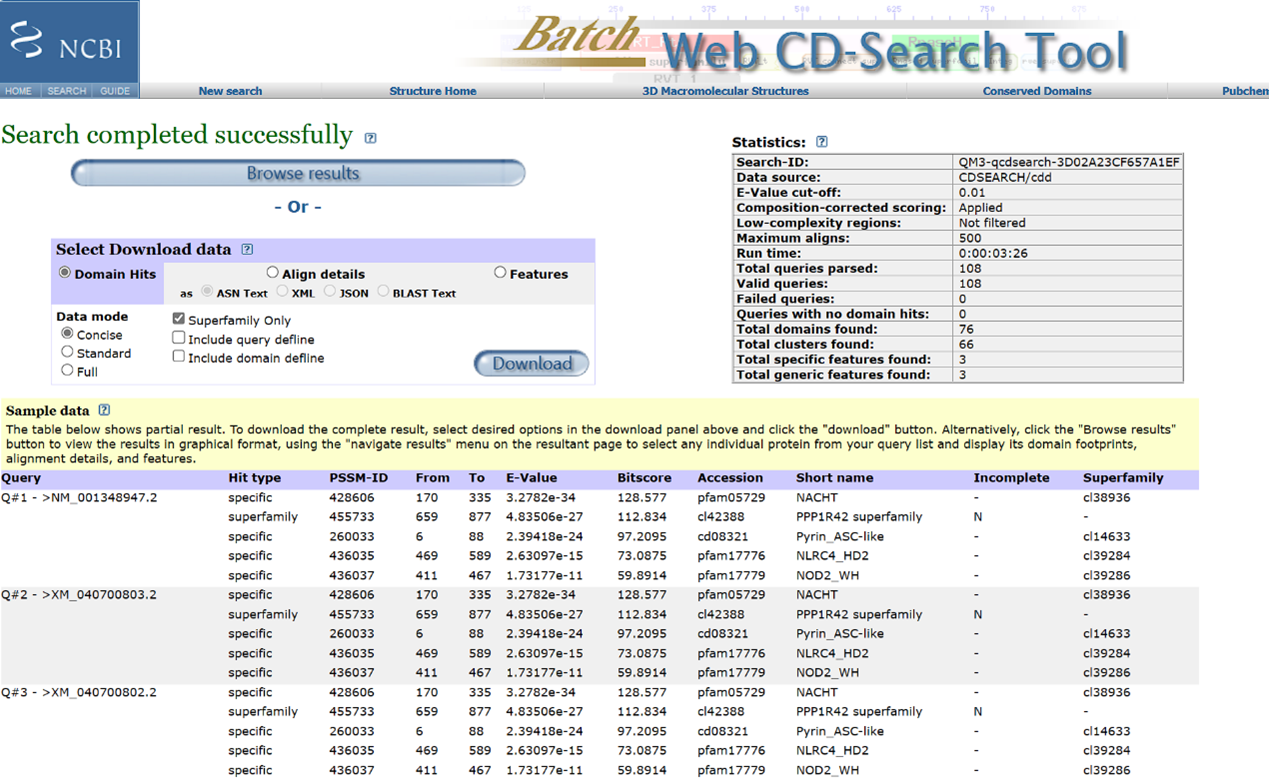
TBtools中打开Visualize NCBI CDD Domain Pattern工具,将刚下好的.txt文件和.fa文件拖入,点击start即可获得图片。
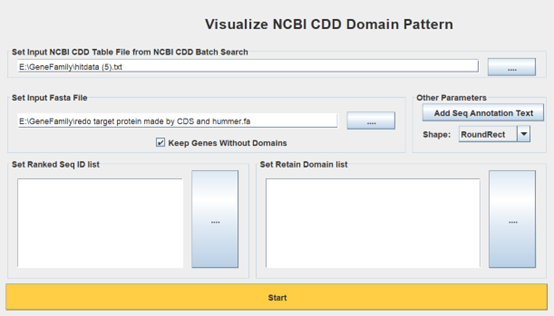
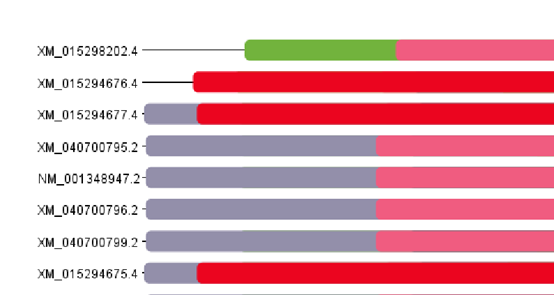
- 基因结构分析
TBtools中 Visualize Gene Structure完成,需要键入基因组注释gff文件和P450基因名。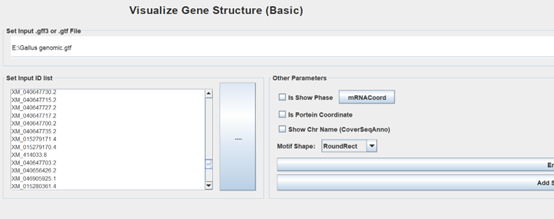 得到以下图片
得到以下图片
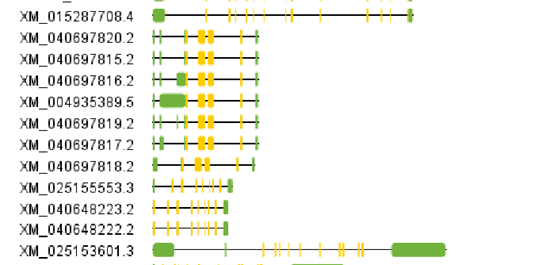
- 基因家族进化分析
通过MEGA构建一个简单的基因树,输入数据为目标物种P450基因家族的蛋白质序列。 - 1打开MEGA界面,上传序列数据
上传文件应为target protein made by CDS and hummer.fa,将序列全部选中后点击Align by MUSCLE进行比对,然后将文件保存为.mas文件
点击PHYLOGENY,选择第一个建树方法,输入文件为保存的.mas文件,使用默认参数(可后期调整)。
进程完成后,进行适当调整,输出.newick文件并保存。
- 展示所有结果
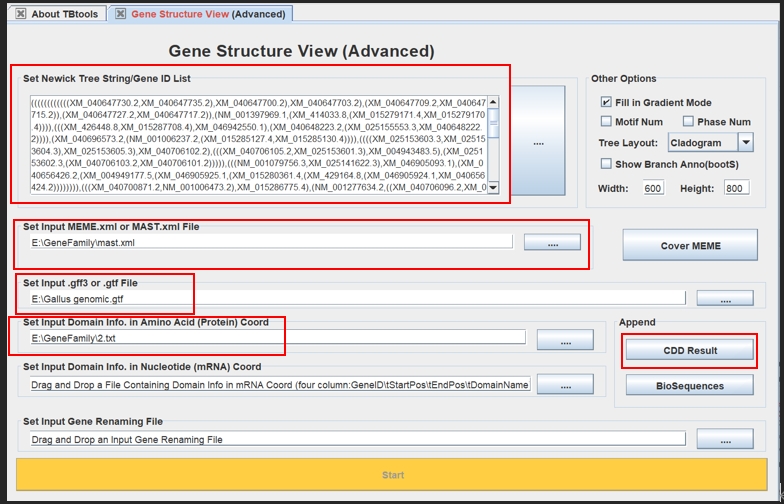
分别将生成的.nwk文件、mast.xml、基因组注释文件、CDD结果以及Domain结果输入
注意,Domain文件需要自己制作,方法如下:
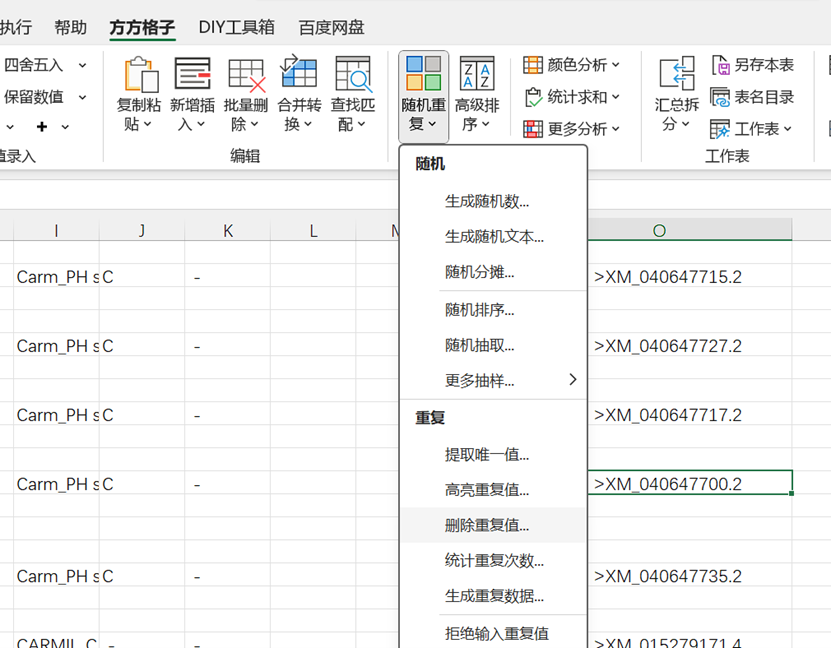
NCBI中CDD结果下载hitdata.txt文件,将整个txt内容复制到excel表中,全部选中,点击方方格子—随机重复—删除重复值—整行对比进行删除,完成后将query列、from列、to列、Short name列移动到一旁,这四列内容应进一步存放在新建txt文本中,在Set Input Doamin info. In amino Acid(protein)功能中使用。
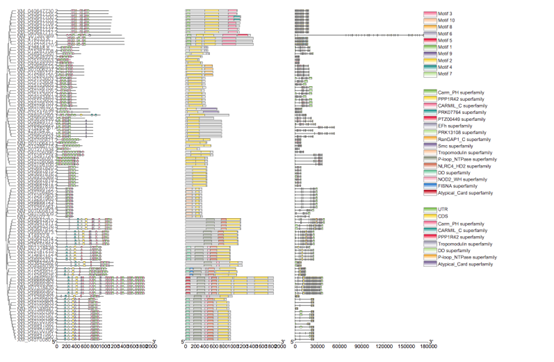
得到以下示例图:
动物的基因家族的生信分析,我想学习一下,但是很多知识点,我不清楚,你能告诉我哪些知识点我应该知道?
在学习动物的基因家族的生物信息学分析时,有几个关键知识点是必须掌握的。通过分析文档【3†source】中关于鸡GATA基因家族的生物信息学分析,我们可以总结出以下几个重要的知识点,这些知识点不仅适用于鸡GATA基因家族的分析,也适用于其他动物基因家族的生物信息学研究:
基因家族成员的鉴定:利用生物信息学方法鉴定基因家族成员,包括使用Ensembl数据库下载基因组序列和基因组结构注释文件,以及使用NCBI BLAST搜索和Pfam数据库比对来获得特定基因家族的成员。
基因和蛋白质的理化性质分析:包括分子质量、等电点、疏水性、不稳定性等参数的预测,以及基因的染色体定位。
保守结构域分析:利用SMART等工具分析基因或蛋白质的保守结构域,这对理解其功能和分类有重要意义。
基因结构分析:包括外显子和内含子的组成及其排列顺序,这有助于理解基因的功能和调控机制。
进化关系分析:构建系统进化树,分析基因家族成员之间以及与其他物种基因家族成员之间的进化关系,通常使用MEGA-X、Clustal W等软件。
蛋白质结构预测:包括二级和三级结构的预测,了解蛋白质的结构特征,为进一步的功能研究提供基础。
蛋白质互作分析:使用STRING等在线平台构建蛋白质相互作用网络,分析基因家族成员如何通过与其他蛋白质的相互作用参与生物过程。
掌握上述知识点是进行动物基因家族生物信息学分析的基础。此外,熟悉相关的生物信息学工具和数据库也非常重要,包括但不限于Ensembl、NCBI、Pfam、SMART、MEGA-X、Clustal W、ProtParam、MEME、TBtools、SOPMA、SWISS-MODEL和STRING等。通过系统学习和实践这些知识点和工具,你将能够有效地进行动物基因家族的生物信息学分析。
1,什么是基因家族?
基因家族是指在一个物种的基因组中,由于基因复制事件而产生的一组具有明显序列相似性的基因。这些基因通常编码功能相似或相关的蛋白质,它们可能参与相同或相似的生物学过程,具有共同的结构域或活性位点。基因家族的成员在进化过程中可能经历了功能的分化,使得它们可以在不同的生物学路径或过程中发挥作用。
基因家族的形成通常归因于两种主要机制:
- 基因复制:这是基因家族形成的最主要机制,包括染色体段复制、基因组整体复制(多倍体化),以及基因的串联复制。复制事件可以提供原始的遗传材料,随后通过进化过程中的变异和选择作用,复制的基因可能获得新的功能或对原有功能的改进。
- 逆转录和重组:逆转录产生的RNA模板通过逆转录酶逆向合成为cDNA,然后可能通过重组等方式插入基因组的其他位置,这也能导致基因家族成员数量的增加。
基因家族的研究对于理解生物复杂性、物种适应性和进化过程非常重要。通过比较不同基因家族成员的功能和表达模式,科学家可以揭示基因如何调控复杂的生物过程,如发育、代谢和应对环境压力等。此外,基因家族中的某些成员在疾病的发生发展中可能起着关键作用,因此对基因家族的研究也对疾病的预防、诊断和治疗具有重要意义。
2,为什么要研究它?
研究基因家族对于生物学和医学领域有着重要的意义,原因包括但不限于以下几点:
理解基因的功能和调控机制:基因家族成员往往参与相似或相关的生物学过程,通过研究这些基因如何互相作用以及它们在不同生物学过程中的作用,我们可以深入理解基因的功能和调控机制。这有助于揭示生命活动的基本规律。
揭示进化过程:基因家族的形成和分化通常伴随着物种的进化。通过比较不同物种间基因家族的差异,可以揭示基因如何在进化过程中适应环境的变化,这对于理解物种多样性和进化历史具有重要意义。
疾病机制研究和治疗靶点发现:基因家族中的某些成员可能在疾病的发生、发展中起着关键作用。通过研究这些基因的功能和相互作用,可以帮助我们理解疾病的分子机制,并可能发现新的治疗靶点或治疗方法。
药物开发:基因家族中的一些成员可能作为药物的靶点,通过特异性调控这些基因的活性,可以开发出新的治疗药物。例如,某些基因家族成员可能参与调节炎症反应、肿瘤生长等过程,成为治疗这些疾病的潜在药物靶点。
农业生物技术:在农业生物技术中,通过研究植物基因家族的功能,可以培育出具有优良性状的作物品种,如增强抗逆性、提高产量和改善品质等。基因家族成员的功能研究为转基因技术和分子育种提供了理论基础和技术支持。
环境保护和生态学研究:基因家族在生物适应环境变化中发挥作用,通过研究这些基因如何响应环境压力,可以帮助我们理解生态系统中生物多样性的维持机制,对于生态保护和环境管理具有指导意义。
综上所述,研究基因家族不仅有助于深入理解基因在生物体内的功能和调控机制,还对疾病的预防、诊断和治疗,以及农业和环境保护等领域具有重要的应用价值。
3,基因家族成员的鉴定:利用生物信息学方法鉴定基因家族成员,包括使用Ensembl数据库下载基因组序列和基因组结构注释文件,以及使用NCBI BLAST搜索和Pfam数据库比对来获得特定基因家族的成员。
比如我想研究鸡的节律或羽色,或别的性状,这一步要怎么做?
研究鸡的节律、羽色或其他性状的基因家族成员时,你可以遵循以下步骤来鉴定相关的基因家族成员:
1. 确定研究目标
首先明确你感兴趣的特定性状或生物学过程,如节律、羽色等。这将帮助你聚焦于相关的基因或基因家族。
2. 文献回顾
进行文献回顾,查找已经被报道与你感兴趣的性状相关的基因或基因家族。这可以通过阅读科学文章、基因数据库的描述等方式完成。了解这些基因的名称、功能以及它们在鸡中的同源基因。
3. 使用Ensembl数据库
- 访问Ensembl数据库:前往 Ensembl鸡基因组浏览器。
- 搜索基因:使用已知的相关基因名称或关键词进行搜索,找到感兴趣的基因及其在鸡基因组中的位置。
4. NCBI BLAST搜索
- 收集序列:从Ensembl或文献中获得已知相关基因的序列。
- BLAST搜索:使用NCBI BLAST工具进行同源性搜索。在NCBI BLAST中,选择“nucleotide BLAST”或“protein BLAST”,根据你收集的是DNA序列还是蛋白质序列来决定。将序列粘贴到查询框中,设置目标数据库为“Reference genomic sequences (refseq_genomic)”或特定于鸡的数据库,然后进行搜索。
5. Pfam数据库比对
- 访问Pfam数据库:前往 Pfam数据库。
- 搜索保守结构域:使用已知相关蛋白质的保守结构域进行搜索,找到与这些保守结构域相匹配的其他蛋白质。这有助于识别可能属于同一基因家族的其他成员。
6. 集成数据和进一步分析
- 数据整合:将从Ensembl、NCBI BLAST和Pfam获得的信息整合起来,识别出可能与你感兴趣的性状相关的基因家族成员。
- 基因注释和进一步的生物信息学分析:对鉴定出的基因进行注释,包括它们的表达模式、功能、进化关系等。可以使用如Gene Ontology (GO)、KEGG Pathway等工具进行功能和路径分析。
通过上述步骤,你可以鉴定并分析与鸡的特定性状相关的基因家族成员。这个过程可能需要多次迭代和详细的分析来精确地确定相关基因家族和成员。
你能帮我搜索一下吗?
要研究鸡的生物节律相关基因,可以关注核心组成部分和调控机制作为基础。在哺乳动物中,生物节律网络由转录因子如CLOCK和BMAL1控制,它们激活如Per1、Per2、Cry1和Cry2等基因。这些基因产生的蛋白质通过反馈循环抑制自身的转录,从而在维持生物节律周期中发挥关键作用。此外,这个网络包括节律性组蛋白修饰、RNA聚合酶II的招募、以及翻译后修饰等调节层面【16†source】。https://genomemedicine.biomedcentral.com/articles/10.1186/s13073-019-0704-0
对于人类的生物节律障碍,特定的生物钟基因突变与诸如家族性睡眠相位提前综合征等状况有关,其中PER2基因的突变是一个显著的例子。这些关于人类生物节律遗传学的见解强调了这些核心生物节律基因的重要性及其在鸡中的潜在相关性【17†source】。
https://www.nature.com/articles/nrg.2016.150
将这些信息转化为研究鸡时,可以开始通过使用Ensembl或NCBI等数据库,识别这些核心生物节律基因(例如,CLOCK、BMAL1、PER、CRY)在鸡基因组中的同源基因。对这些基因在鸡或亲缘关系接近的物种中的文献回顾也能提供宝贵的见解。为了从更广阔的视角理解生物节律及其对包括睡眠、代谢和免疫反应在内的各种生理系统的影响,所引用的综述和研究提供了丰富的知识,可以指导你的研究方向并帮助构建你的假设【16†source】【17†source】。https://genomemedicine.biomedcentral.com/articles/10.1186/s13073-019-0704-0
https://www.nature.com/articles/nrg.2016.150
鉴于生物节律的复杂性及其对生理和行为的广泛影响,专注于这些核心组成部分及其调控机制可以为理解鸡的生物节律提供坚实的基础。
我找到了一些关于基因家族分析和生物节律的资料,特别是与哺乳动物和斑马鱼相关的研究,这些研究可以提供一些对鸡进行类似研究的启示。
在哺乳动物中,一项研究构建了一个基于体外荧光素酶报告基因分析的小规模基因调控网络,涵盖16个基因和3个顺式调控元件。这项研究识别了多达9,995个在至少一个组织中显示生物节律振荡的已知基因,并发现41个常见的生物节律基因在多个组织中表现出生物节律振荡。这些结果提高了我们对关键生物节律基因如何驱动生物节律控制基因的理解,并为哺乳动物生物节律的更详细定量建模铺平了道路【25†source】。
https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1000193
在斑马鱼的研究中,通过全基因组转录组分析,识别出2,856个在长日照和恒暗条件下均显示生物节律振荡的基因。这项研究还发现,斑马鱼中几乎所有已知的哺乳动物核心生物节律基因的同源基因都代表在这些生物节律振荡基因中,表明基本的生物节律回路在斑马鱼和哺乳动物之间高度保守。此外,这项研究还揭示了光照对生物节律基因表达的影响,指出生物节律活动的振幅在恒暗条件下明显降低,表明生物节律活动在很大程度上依赖于光照的调节【26†source】。
https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1002940
这些研究显示,生物节律基因的识别和分析可以为理解生物节律的调控机制提供重要信息。虽然这些研究主要集中在哺乳动物和斑马鱼上,但它们的方法和发现可以为研究其他物种,包括鸡的生物节律提供参考。通过综合应用转录组数据分析、基因调控网络构建和生物节律基因的功能注释,我们可以更好地理解生物节律在不同生物体中的共性和特异性。
研究基因家族,,为什么需要转录组数据
研究基因家族时使用转录组数据有多个原因,以下是一些主要的理由:
基因表达水平的了解:转录组数据能提供在特定条件下,所有基因的表达水平信息。这对于了解基因家族中不同基因的功能及其在不同生物学过程中的角色至关重要。
识别差异表达基因:通过比较不同样本(如疾病状态与正常状态,或不同发育阶段)的转录组数据,研究人员可以识别出差异表达的基因,这对于揭示基因家族成员在特定条件下的作用机制和生物学功能非常有帮助。
功能注释:基因家族中的许多基因可能尚未有详细的功能注释。转录组数据可以帮助研究人员通过基因的表达模式推断其可能的功能,尤其是通过与已知功能的基因表达模式进行比较。
基因表达调控机制的研究:通过分析转录组数据,研究人员可以探索基因表达的调控机制,例如转录因子的结合位点,这对于理解基因家族成员是如何被精确调控的非常重要。
新基因的发现:转录组测序(RNA-Seq)可以揭示基因组中未被注释的转录活动区域,有助于发现基因家族中的新成员。
进化关系分析:通过比较不同物种中基因家族的转录组数据,研究人员可以探讨基因家族的进化历史,识别进化过程中保守的基因以及物种特异的基因扩张事件。
表型和基因型之间的联系:转录组数据可以帮助建立基因表达变化与特定表型之间的关联,这对于理解基因家族成员如何共同作用以调控复杂的生物学性状非常有价值。
总之,转录组数据为研究基因家族提供了一个强大的工具,可以深入理解基因的表达模式、调控机制、功能以及它们如何在不同生物学背景下作用,从而揭示它们在生命过程中的重要性。
鸡NLRs基因系统进化树构建
为进一步分析不同物种间 NLR 基因的亲缘
关系,基于哺乳类、鸟类、爬行类、两栖类和鱼类代
表物种的 NLR 基因蛋白序列,利用 MEGAX 进行
多重序列比对构建了 NJ 树
这个要怎么做呀
构建NLR(Nucleotide-binding domain and Leucine-rich Repeat containing)基因家族的系统进化树是了解这个基因家族在不同物种间进化关系的重要方法。下面是根据你提供的信息,利用MEGAX软件进行NJ(Neighbor-Joining)树构建的基本步骤:
收集数据:首先,你需要收集各个物种(哺乳类、鸟类、爬行类、两栖类和鱼类)的NLR基因蛋白序列。这些数据可以从公共数据库如NCBI、Ensembl或UniProt获取。
序列格式转换:确保所有的序列都是以适合MEGAX软件的格式保存的。MEGAX支持多种格式,包括FASTA格式。如果你的序列不是在这种格式,你可能需要使用序列转换工具进行格式转换。
打开MEGAX:安装并打开MEGAX软件。MEGAX是一个用于分子进化分析的综合软件,可以在其官方网站免费下载。
多重序列比对(MSA):在MEGAX中,使用“Align/Build Alignment”功能对你的NLR蛋白序列进行多重序列比对。比对可以使用ClustalW、MUSCLE等算法。确保比对后的序列质量良好,必要时可以手动调整。
构建NJ树:
- 使用“Phylogeny”菜单下的“Construct/Test Neighbor-Joining Tree”功能来构建系统进化树。
- 你需要选择一个适当的模型。对于蛋白质序列,通常使用JTT、Dayhoff或WAG模型。
- 确定其他参数,如是否进行Bootstrap分析来评估树的可靠性。Bootstrap通常设置为1000次重采样,以获得可信的支持率。
- 启动分析。分析完成后,MEGAX会展示NJ树,你可以对树进行调整和美化,如改变分支颜色、添加标签等。
解读和保存你的进化树:分析和解读NJ树,理解不同物种间NLR基因的亲缘关系。保存你的树状图用于报告或进一步分析。
以上步骤大致介绍了如何使用MEGAX软件构建NLR基因的NJ系统进化树。每一步都有很多详细的选项和设置,具体可以参考MEGAX的用户手册和在线资源来获取更详细的指导。这个过程需要一定的生物信息学知识和实践经验,如果你是第一次进行这类分析,可能需要一些时间来熟悉软件和分析方法。



