找到PMEL基因位点,做GWAS
性染色体:plink –file BS –chr Z –from-bp 27059520 –to-bp 27074222 –make-bed –out output_BS –allow-extra-chr –chr-set 39
1,在NCBI中搜索PMEL基因
看到PMEL基因的位置在34号染色体,1339431-1343416位置
2,用plink代码在采样取得的vcf,转换成map,ped。命名为BB.map,BB.ped
plink –vcf BB.vcf –recode –out BB -chr-set 39
3,用plink代码在34号染色体,1339431-1343416位置进行筛选
plink –file BB –chr 34 –from-bp 1339431 –to-bp 1343416 –make-bed –out output_BB –chr-set 39
plink –file BB –chr 34 –from-bp 1339431 –to-bp 1345613 –make-bed –out output_BB –chr-set 39
4,解读输出的数据
(1)output_BB.bed,二进制,01表示
(2)output_BB.bim,在34号染色体的1339431-1343416位置找到67个snp变异
(3)output_BB.fam,
5,再用plink把得到的bim,fam,bed转换成ped和map格式
plink –bfile output_BB –recode –out new_choice_BB –chr-set 39
生成的文件内容
(1)new_choice_BB.map生成的文件与bim很像,67行
(2)new_choice_BB.map.ped
除了前面6列的样品信息外,还有134列,正好等位基因,是67的二倍。
6,综上,应该能找到PMEL基因在每个样品中的变异信息。
我尝试再试一下3,表型关联分析:
plink –bfile output_BB –pheno phenotypes.txt –allow-no-sex –logistic –adjust –output_choice_ logistic_results –chr-set 39
用R语言作图:曼哈顿图和QQ图
不想弄了,文档在这,曼哈顿图和QQ图
链接:https://caiyun.139.com/m/i?065CtJ9Q4UoY2
提取码:yj1U
复制内容打开移动云盘PC客户端,操作更方便哦
要在R语言中使用qqman包来生成Q-Q图和曼哈顿图,你需要遵循以下步骤。首先确保你已经安装了qqman包。如果还没有安装,可以通过运行以下代码来安装:
1 | install.packages("qqman") |
安装完成后,加载qqman包:
1 | library(qqman) |
接下来,使用qqman包中的函数来生成图表。这里以生成曼哈顿图为例,假设你已经有了GWAS分析的结果数据,并且该数据已经被加载到R中一个名为gwasResults的数据框架(data.frame)中。你的数据应该包含至少三列:染色体位置(通常是CHR),基因位点位置(通常是BP),以及每个位点的P值(通常是P)。
1 | manhattan(gwasResults) |
若要生成Q-Q图,可以使用如下代码:
1 | qq(gwasResults$P) |
这里,gwasResults$P指的是你的GWAS结果中P值的列。注意,gwasResults需要被替换为你自己的数据框架变量名,而P则是该数据框架中包含P值的列的名称。
qqman包的灵活性还允许你自定义图表的各个方面,比如颜色、点的大小,以及其他图形参数。更多详细信息和高级用法,推荐查看包的vignette:
1 | vignette('qqman') |
这将提供更多关于如何使用qqman包以及如何利用它来可视化你的GWAS结果的示例和指导。
你的代码已经很好地使用了qqman包来生成曼哈顿图和Q-Q图。这是分析GWAS数据时的一个常见步骤,可以帮助你识别与特定表型相关的位点。不过,有几个改进的建议可以使你的代码更加健壯、易于理解和维护:
添加图形输出结束命令:你开始了一个JPEG图形设备来保存曼哈顿图,但是没有关闭这个图形设备。在生成完图像后,使用
dev.off()来确保图形文件被正确保存并且后续的图形输出不会被误写入同一文件。增强图形的可读性和美观性:虽然
qqman的默认设置已经可以生成不错的图形,但是考虑到不同的出版需求,你可能想要调整图形的大小、字体大小、颜色等。这可以通过额外的参数来实现,例如,为manhattan和qq函数添加cex.axis(轴上文字的大小)、cex.lab(标签文字的大小)和col(用于高亮显著位点的颜色)参数。检查数据的完整性和准确性:在制图之前,对你的数据进行一些基本的检查,比如是否有遗失值(NA),或者某些列的数据类型是否正确(例如,染色体编号
CHR是否被正确读取为因子或字符型,而不是数值型)。使用注释和函数封装:如果你计划在不同的分析中重复使用这段代码,或者分享给同事,添加注释和将代码片段封装成函数会很有帮助。这样不仅能使代码更易于理解,还能提高代码的重用性。
综上,你的代码可以优化如下:
1 | install.packages("qqman") |
这些优化可以帮助你更好地管理和展示你的分析结果。
在R中,生成不同格式的图形文件(如JPEG, PNG, SVG, PDF等)可以通过使用特定的图形设备函数来完成。每种格式基本上都有对应的函数来开启和配置图形输出。以下是一些常见格式的示例:
JPEG
你已经使用了jpeg()函数来保存JPEG格式的图像。jpeg()函数开启一个JPEG图形设备,所有的图形输出都会被写入指定的文件,直到调用dev.off()。
1 | jpeg("your_plot.jpeg") |
SVG
使用svg()函数来保存图形为SVG格式,这种格式非常适合网页显示或任何需要可伸缩图形的场合。
1 | svg("your_plot.svg") |
PNG
PNG是另一种常用的图形格式,特别适合网页使用。使用png()函数来保存图形为PNG格式。
1 | png("your_plot.png") |
PDF格式非常适合打印和发布,因为它可以在不同的设备和打印机之间保持一致的外观。使用pdf()函数来保存图形为PDF格式。
1 | pdf("your_plot.pdf") |
使用示例
这是一个完整的示例,展示如何生成并保存SVG格式的Q-Q图:
1 | install.packages("qqman") |
在使用这些函数时,你可以设置一些参数,比如图形的尺寸、分辨率(对于基于像素的格式)等,来优化输出的图形文件。每个函数的文档中都有详细的参数列表和说明。
在qqman包的manhattan函数中,直接显示超过特定阈值的SNP标签不是内置的功能。不过,你可以通过在生成曼哈顿图之后,使用R的基础绘图函数手动添加这些标签。
以下是一个基本的示例,展示如何实现这一功能。这里假设我们的阈值是 -log10(p-value) 大于5,我们想要为所有满足这一条件的SNP添加标签。
1 | # 生成曼哈顿图 |
这段代码首先生成了曼哈顿图,然后通过subset函数找出所有-log10(P)值大于5的SNP。with函数用于将这些SNP的位置(基因位点位置BP)和相应的-log10(P)值传递给text函数,text函数则在图上相应的位置添加SNP的标签(labels = SNP)。
请注意,这种方法可能会导致标签相互重叠,特别是当标记点非常靠近或者有很多点需要标记的时候。可以通过调整text函数中的cex(字符扩展大小)和pos(文本位置)参数来尝试减少重叠。在实践中,可能还需要对数据进行一些过滤或者使用其他技巧(如仅标记最显著的几个SNP或者使用交互式图形)来优化标签的显示。
另外,如果你的数据集非常大,绘图和标注过程可能会比较慢。在这种情况下,考虑对数据进行适当的预处理,或者使用更高效的绘图工具(如ggplot2)可能会有所帮助。
在生物信息学中,全基因组关联研究(GWAS)是一种常用的方法,用于在不同物种的基因组中识别与特定表型相关联的基因变异。关于您的问题,是否可以将两个不同物种的数据合并到一起进行GWAS分析,这取决于多个因素,包括但不限于物种的遗传背景、研究目的以及数据的可比性。
分开做GWAS的理由:
遗传差异性:不同物种之间的遗传差异可能非常大,这意味着它们可能在基因组结构、基因的存在与否、以及单核苷酸多态性(SNPs)的分布等方面存在显著差异。这些差异可能导致数据不具可比性,进而影响GWAS分析的准确性和解释性。
表型定义:即使是相似的表型,在不同物种中也可能由不同的遗传因素控制。合并不同物种的数据可能会掩盖特定物种内的遗传信号。
统计功效:将不同物种的数据合并可能会增加背景噪声,从而降低识别相关遗传变异的统计功效。
合并做GWAS的考虑:
进化相近的物种:如果两个物种在进化上非常接近,并且遗传背景相似,那么在某些情况下,可以考虑合并数据,特别是当研究的是高度保守的遗传特征时。
元分析:而不是直接合并原始数据,一种可行的策略是对每个物种分别进行GWAS,然后通过元分析方法合并结果。这种方法可以识别跨物种共享的遗传信号,同时考虑到种间的差异。
跨物种比较:在一些研究中,目的可能是要比较不同物种之间的遗传差异,而不仅仅是识别与特定表型相关的遗传变异。在这种情况下,可以使用专门设计的比较基因组学方法,而不是传统的GWAS方法。
结论:
通常情况下,建议对每个物种单独进行GWAS分析,这是因为物种间的遗传背景差异可能导致数据不具可比性,从而影响分析的准确性。如果有合并分析的需求,需要谨慎评估并采用适当的统计方法和生物信息学工具,可能还需要专门的设计和方法来克服跨物种分析中的挑战。在特定情况下,元分析或者跨物种比较研究可能是更合适的方法。
GWAS后续:曼哈顿图的显著位点和周围基因的富集分析
通过R代码
1 | # 定义显著性阈值 |
1.png
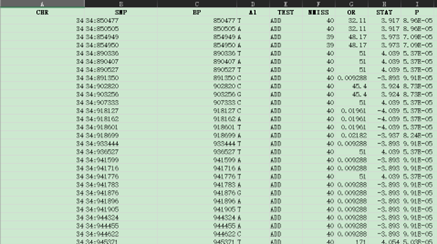
筛选曼哈顿图中小于-log10(1e-4)的snp
最后从8212626snp筛选出173个snp,都是34号染色体。范围从850000到150000
在ensemble的biomart中筛选34号染色体的850000到150000。
注释到73个基因
富集分析一下
上面的是R语言注释到的
用DIVID注释下
1,GO:0006357调控RNA聚合酶II启动子的转录:这些基因参与调节RNA聚合酶II启动子上的转录过程。有丝分裂细胞周期:这些基因参与有丝分裂细胞周期,也就是细胞从一个有丝分裂到下一个有丝分裂的过程。
2. GO:0000278
3. GO:0000226微管细胞骨架的组织:这些基因参与调节细胞内微管骨架的组织和结构。细胞核:这些基因定位于细胞核内,可能在核内发挥功能。
4. GO:0005634
5. GO:0000978RNA聚合酶II核心启动子近区特异性DNA结合:这些基因具有特异性地与RNA聚合酶II核心启动子的近区DNA结合。RNA聚合酶II转录因子活性,特异性DNA结合:这些基因表现出特异性的RNA聚合酶II转录因子活性,并与DNA特异性结合。
6. GO:0000981
7. GO:0016208~AMP结合:这些基因具有与AMP(腺苷酸)结合的能力。
这四个条目是关于基因调控的不同通路或功能的参考Kegg Pathway ID(Kegg通路编号),含义如下:
- gga04530: Tight junction - 紧密连接通路,指细胞膜上的一种结构,通过细胞间蛋白连接而形成。紧密连接对于维持细胞屏障和选择性渗透性至关重要。
- gga04145: Phagosome - 噬菌体通路,指吞噬单个或多个微生物或其他颗粒物的过程。此通路涉及到吞噬、分解、展示和发表免疫应答等步骤。
- gga04540: Gap junction - 间隙连接通路,是指细胞之间形成的一种连接方式。这种连接允许细胞之间通过跨膜通道共享信号和小分子物质,从而实现组织或群体功能。
- gga04814: Motor proteins - 运动蛋白通路,是指细胞中的运动蛋白参与的调控过程。运动蛋白包括肌动蛋白、微管动力蛋白等,它们对于细胞内输运、肌肉收缩、神经元轴突生长等生物学过程发挥重要作用。
这些通路或功能与基因参与的生物学过程密切相关,通过研究基因对这些通路或功能的调控,可以进一步了解这些生物学过程在分子水平上的机制。



