1,校报
文章思路
https://snengs.nichd.nih.gov/details/Chicken/Pineal 网站
以rain包的内置数据集”menetRNASeqMouseLiver”为例作演示,该数据集来自于一项对小鼠昼夜节律转录调控的研究。小鼠饲养在12h光照/12h黑暗的24h周期中,饲养一段时间后,每隔4h取样一次,通过高通量测序获得的小鼠松果体基因表达谱的时间序列。
以rain包的内置数据集”menetRNASeqchickenLiver”为例作演示,该数据集来自于一项对小鼠昼夜节律转录调控的研究。小鼠饲养在12h光照/12h黑暗的24h周期中,饲养一段时间后,每隔4h取样一次,通过高通量测序获得的小鼠松果体基因表达谱的时间序列。然后找周期性的基因,然后WGCNA,然后靶基因,我这次实验的目的是什么?
基于你的描述,这次实验的目标是探索和理解小鼠松果体在昼夜节律中的转录调控机制,特别是通过识别和分析具有周期性表达模式的基因,以及这些基因在网络中的角色和相互作用。以下是实验目的的详细分析:
识别周期性基因:利用”rain”包和内置数据集”menetRNASeqchickenLiver”,首先目的是识别在昼夜节律条件下,即12小时光照/12小时黑暗周期中,显示出明显周期性表达模式的基因。周期性基因的识别有助于了解哪些基因在调节生物钟和昼夜节律中起着关键作用。
构建基因表达网络(WGCNA):通过加权基因共表达网络分析(WGCNA)方法,目的是构建基因间的表达关系网络,并将基因根据表达模式分组成模块。这一步骤可以揭示基因间的协同表达关系,以及不同模块可能反映的生物过程或功能。
靶基因的识别和分析:在识别了周期性基因并通过WGCNA找到相关的模块之后,接下来的目的是进一步筛选和分析这些模块中的关键基因或靶基因,这些基因在昼夜节律调控中可能扮演核心角色。特别是,将寻找那些在其所属模块中具有高连接度的枢纽基因,以及那些与特定生物学特征或昼夜节律相关性状高度相关的基因。
探索昼夜节律转录调控的机制:通过上述分析,最终的目的是构建一个关于小鼠松果体在昼夜节律中的转录调控网络的全貌,特别是揭示那些周期性基因如何通过相互作用和网络中的角色参与昼夜节律的维持和调节。这包括理解特定基因如何参与调节生物钟的分子机制,以及这些机制如何影响小鼠的生理和行为模式。
总之,这次实验的目的是通过综合应用生物信息学方法和统计分析,深入了解昼夜节律在分子层面的调控机制,特别是通过识别和分析周期性表达的基因及其在基因表达网络中的角色,来揭示调控生物钟和昼夜节律的关键基因和途径。这些发现有望为进一步研究昼夜节律调控的生物学基础和潜在的临床应用提供重要的见解。
我最终大概发现rain筛选出大部分周期性的基因,里面包含很多节律基因,然后WGCNA发现不同时刻有特定的基因表达,节律基因被分到不同的模块,进行富集分析,然后筛选枢纽基因,确实一些节律基因被当作枢纽基因筛选出来了,但是我不太会讨论这个结论和总结,我应该怎么说,符合一篇论文的讨论和总结?
在撰写科研论文的讨论和总结部分时,你的目标是解释你的发现意味着什么,它们如何与已有的知识体系相联系,以及这些发现的潜在影响和意义。以下是一些建议,帮助你讨论和总结你的研究成果:
讨论
解释周期性基因的发现:首先,强调你使用rain包成功鉴定了大量具有周期性表达模式的基因,包括多个已知的节律基因。解释这一发现如何证实了你的实验设计和分析方法的有效性,以及它们在昼夜节律调控中的作用。
分析WGCNA结果:讨论通过WGCNA揭示的不同时间点特定基因表达模式的意义。强调节律基因如何分布在不同模块中,以及这反映了什么样的生物学现象或调控机制。例如,不同模块可能代表了不同的生物钟调控途径或参与调控不同生理过程的基因群。
富集分析的意义:解释富集分析如何帮助你理解每个模块中基因的生物学功能和参与的生物途径。讨论这如何增进了对昼夜节律调控复杂性的理解,尤其是那些模块特定的节律基因如何与特定生物过程和病理状态相关联。
枢纽基因的讨论:详细讨论那些被识别为枢纽基因的节律基因的重要性。探讨它们可能的功能和在昼夜节律调控中的角色,包括如何通过与其他基因的相互作用影响生物钟的准确性和稳定性。
与已有研究的比较:将你的发现与已有文献中的知识进行比较,强调你的研究如何填补了现有知识的空白,或是如何对现有理论提出了挑战或提供了新的视角。
总结
总结主要发现:简洁明了地总结你的研究发现了哪些关键的周期性基因和节律基因,以及它们在昼夜节律调控中可能扮演的角色。
强调研究的贡献:强调你的研究为理解昼夜节律调控机制、揭示节律基因在不同生物过程中的作用,以及为未来研究提供了哪些新的视角或工具。
讨论潜在应用:提出你的研究成果可能的应用领域,例如在调节睡眠模式、治疗昼夜节律失调疾病或开发新的药物目标方面的潜在价值。
指出研究的局限性:诚实地讨论你的研究设计和分析方法的任何局限性,以及这些局限性可能如何影响你的结论。
提出未来研究方向:基于你的发现,提出未来研究可以探索的问题或方向,如进一步的功能验证实验,或是探索节律基因如何在
不同环境条件下调节生物钟的机制。
记得,科研论文的讨论和总结是向读者展示你的研究成果在科学社区中的重要性和影响。通过清晰、逻辑性强的论述,你不仅能展示你的研究成就,还能激发他人对这一领域进一步研究的兴趣。
RNA-seq最后分析会得到FPKM的一个表,那个表的第一列是基因IID好呢?还是转录本ID好呢?一般论文用的什么?
RNA-seq分析得到的FPKM(Fragments Per Kilobase of transcript per Million mapped reads)值是用来估计基因或转录本的表达量。在选择基因ID还是转录本ID作为表的第一列时,这取决于研究的目的和上下文。
基因ID:如果研究的重点是在基因水平上比较表达量,使用基因ID会更合适。这种情况下,一个基因可能包含多个转录本,但分析会聚焦于基因整体的表达量变化。在很多研究中,特别是那些关注特定基因如何在不同条件下调节的研究,使用基因ID是更常见的做法。
转录本ID:如果研究关注的是具体的转录本或者是要探索不同的剪接变异,那么使用转录本ID会更加合适。这允许研究者能够区分同一基因的不同转录本表达情况,这对于理解基因调控的复杂性和剪接事件非常重要。
一般论文使用什么:
- 在实践中,具体使用哪个ID往往取决于研究的具体目标和领域。在一些论文中,研究者可能会同时报告基因级别和转录本级别的分析结果,尤其是在基因表达差异分析和转录本多样性研究中。
- 基因级别的分析较为常见,特别是在那些关注特定基因表达差异、基因功能或疾病相关基因研究中。而在更细致地探究基因表达调控机制、剪接变体和功能性转录本等领域,转录本ID的使用则更为频繁。
总的来说,选择基因ID还是转录本ID作为FPKM表的第一列应当基于你的研究目标来决定。如果你的研究目的不明确或者两者都很重要,你可以考虑在你的分析中同时使用这两种标识符,并在论文中清楚地报告你的分析策略和发现。
将转录本ID的表格转换为基因ID的表格并不总是简单的一一对应关系,因为一个基因可能包含多个转录本。因此,直接将转录本ID替换为基因ID可能会导致信息的丢失或重复计算。在处理这个转换时,应当考虑以下几个步骤:
识别转录本和基因之间的关系:首先,需要确定每个转录本ID对应的基因ID。通常,转录本ID(如Ensembl数据库中的ENSGALT开头的ID)和基因ID之间的关系可以通过公共数据库查询得到,比如Ensembl、NCBI等。这些数据库提供了转录本到基因的映射信息。
汇总转录本表达量到基因级别:由于一个基因可能对应多个转录本,因此在将转录本FPKM值转换为基因级别时,你需要决定如何处理这些转录本的表达量。常见的方法包括:
- 求和:对同一基因的所有转录本的FPKM值求和,以得到该基因的总表达量。
- 平均:计算同一基因的所有转录本的FPKM值的平均值,作为该基因的表达量。这种方法可能需要根据转录本长度进行加权平均。
- 最大值:选取同一基因中FPKM值最高的转录本作为该基因的表达量,这假定最高表达的转录本最能代表基因的活性。
创建新表格:根据转录本到基因的映射关系以及选择的汇总方法,创建一个新的表格,其中第一列是基因ID,后续列是根据所选方法计算得到的基因级表达量。
考虑生物学重复和技术重复:在汇总表达量时,还应该考虑到来自不同生物学重复或技术重复的数据,确保统计处理的正确性。
这个过程可能需要编写脚本或使用现有的生物信息学工具来自动化。在进行这种转换时,重要的是要明确记录你使用的每个步骤和方法,以便在将来的分析和发表论文时可以清楚地解释你的数据处理流程。如果你需要具体的帮助来实现这个过程(例如,使用Python脚本进行转换),请提供更多的细节。
如果你的研究目的是探索哪些基因可能在小鼠的昼夜节律中发挥作用,那么选择基因ID作为分析的主要对象会更合适。这是因为你关注的是在整个基因水平上的表达变化,而不是特定转录本的差异表达。具体来说,你会想要分析不同时间点(每隔4小时取样)基因表达量的变化,以识别哪些基因的表达具有显著的周期性,从而可能参与调控小鼠的昼夜节律。
在这种研究背景下,分析步骤可能包括:
数据预处理:首先,需要对RNA-seq数据进行质量控制、比对到参考基因组以及计算每个基因在每个样本中的FPKM值。
基因表达量的周期性分析:使用适当的统计方法或生物信息学工具(例如,R包如
rain,circaCompare,JTK_CYCLE等)来分析基因表达量的时间序列数据,以识别表达量显示出昼夜节律变化的基因。显著性测试:对于识别出的周期性表达的基因,进行统计显著性测试,确定这些周期性变化是否具有统计学意义。
功能注释和富集分析:对具有显著节律性表达的基因进行功能注释和富集分析(例如,GO(Gene Ontology)分析和KEGG(Kyoto Encyclopedia of Genes and Genomes)路径分析),以探索它们可能参与的生物学过程和路径。
使用基因ID作为分析的主要对象,你可以更直接地探究整个基因层面上的表达模式和功能,这对于理解昼夜节律背后的基因调控网络至关重要。此外,由于你的研究重点是在基因水平上,这也简化了分析流程,因为不需要对每个基因的多个转录本进行单独分析。
在报告你的发现时,清晰地描述你的数据处理和分析流程,包括如何选择基因ID、如何进行周期性分析和显著性测试,以及你如何解释功能注释和富集分析的结果,这将对读者理解你的研究成果非常重要。
WGCNA(加权基因共表达网络分析)是一种用于从基因表达数据中构建基因共表达网络,并基于模式识别来识别高度协同变化的基因集群(即模块)的方法。这些模块可能反映了基因在特定生物学过程或条件下的共同调控。在你的昼夜节律研究中,WGCNA可以帮助识别哪些基因模块与时间点(即昼夜变化)相关联,进而揭示在调控小鼠昼夜节律中可能起作用的关键基因和路径。
将WGCNA分析整合到你的研究流程中,可以在以下步骤之后进行:
- 数据预处理(完成质量控制、比对和FPKM计算)。
- 基因表达量的周期性分析(识别表现出昼夜节律变化的基因)。
在这两步之后,你可以加入WGCNA分析,步骤大致如下:
3. WGCNA分析:
- 数据准备:选择用于WGCNA的基因表达数据。通常,你会选择所有基因或已经根据前面步骤显示周期性变化的一组基因。
- 筛选和归一化:对选择的基因表达数据进行适当的预处理,包括筛选掉表达量低的基因,以及可能的归一化处理,以准备进行WGCNA。
- 构建共表达网络:使用WGCNA方法构建基因的共表达网络,并基于基因表达模式的相似性将基因聚类成模块。
- 模块特征和相关性分析:分析各个基因模块与外部特征(如时间点)的相关性,识别与昼夜节律显著相关的模块。
- 关键基因和模块的识别:在与昼夜节律显著相关的模块中,识别中心基因或关键调控基因。
4. 显著性测试:对WGCNA中识别的模块和基因进行进一步的统计显著性测试。
5. 功能注释和富集分析:对WGCNA识别的关键模块和基因进行功能注释和富集分析,以探索它们在昼夜节律中可能发挥的作用。
通过整合WGCNA分析,你不仅能识别出显示周期性变化的单个基因,还能通过分析基因模块与昼夜节律的关系来探索更广泛的基因调控网络。这种方法提供了一种从系统层面理解基因表达动态和生物学功能的强大工具,可以揭示潜在的生物学过程和调控机制。
我那个FPKM表有2000行转录本,然后biomart转换后只有800行了,正常吗?能做WGCNA吗
当你将一个包含2000行转录本的FPKM表通过BioMart或类似工具转换为基因ID后,发现行数减少到800,这是可能的,也是可以理解的,原因包括:
多个转录本对应同一基因:一个基因可能有多个转录本,这些转录本在转换为基因ID时会合并为同一行。这是行数减少的主要原因,因为基因水平的分析只考虑每个基因的整体表达,而不区分不同转录本。
转录本信息不完整或不匹配:有些转录本可能在BioMart数据库中没有精确匹配的基因ID,可能是因为转录本编号已更新,或者所用的基因组版本与数据库中的版本不完全对应。
数据过滤:在转换过程中,可能还进行了一些数据过滤,例如移除了表达量极低的转录本或那些无法明确映射到某个基因的转录本,进一步减少了行数。
能否进行WGCNA分析?
- 是的,可以进行WGCNA分析,但要考虑几个因素。首先,虽然理想情况下WGCNA希望分析较大的基因集以揭示更丰富的生物信息和网络结构,800个基因仍然可以进行WGCNA分析,尤其是如果这些基因被认为是在你研究的生物学过程中活跃的。其次,WGCNA的有效性并不仅仅取决于基因的数量,还取决于样本大小、基因表达模式的多样性以及基因之间的相互作用强度。
进行WGCNA的建议:
样本数量:确保样本数量足够以支持网络构建,通常至少需要20-30个样本。
数据质量和预处理:在进行WGCNA之前,确保数据质量高,适当地处理数据(如归一化),并考虑移除任何异常样本或基因,以提高分析的可靠性。
选择合适的参数:在WGCNA中,选择合适的软阈值是关键,这需要根据数据集的特性进行调整。
模块检测的灵敏度:对于较小的基因集,WGCNA可能会检测到较少的模块,或者模块之间的区分度不如在较大基因集中那样明显。因此,在解释结果时需要谨慎。
总的来说,尽管你的基因集相对较小,但仍然可以进行WGCNA分析,尤其是如果这些基因在你的研究背景下具有生物学意义。通过合理的数据处理和分析设置,即使是较小的基因集也能揭示有价值的生物学洞见。
我还发现有些有GENE STABLE ID 但是没有gene name,这正常吗?我应该用那个
是的,这种情况是正常的。在基因注释数据库中,特别是在大型公共数据库如Ensembl中,有些基因可能有稳定的基因ID(例如Ensembl ID),但没有官方的基因名称(Gene Name)。这可能是由于几个原因造成的:
- 新识别的基因:一些基因可能是最近通过基因组项目识别的,尚未有足够的研究来给它们命名。
- 注释的不确定性:对于某些基因,可能由于当前的科学知识或技术限制,其功能、表达模式或重要性尚不完全清楚,因此它们还没有被赋予一个具体的名称。
- 命名标准:有时,基因名称的分配需要遵循特定的命名标准和验证过程,这可能导致一些基因暂时没有官方名称。
在分析中应该使用哪个?
- 使用GENE STABLE ID:在没有基因名称的情况下,使用GENE STABLE ID(如Ensembl ID)是最可靠的选择。这些ID提供了一个唯一和稳定的标识符,可以用来跟踪和查询相关的基因信息。尽管基因名称对于人类阅读更友好,但基因ID在数据库和文献中提供了一种一致性和精确性。
- 记录GENE STABLE ID和基因名称:如果可能,最好同时记录基因的稳定ID和名称(如果有的话)。这样,当基因名称可用或更新时,你可以轻松地识别和引用它们。
如何处理没有基因名称的情况?
- 查找最新的注释:定期检查数据库更新,因为基因的注释信息可能会更新,一些之前没有名称的基因可能已经被赋予了新的名称。
- 使用其他资源:尝试使用其他生物信息学资源或工具(如NCBI Gene,UniProt)来寻找可能的基因名称或相关的功能信息。
- 功能注释分析:利用基因ID进行功能注释和通路分析,这些分析通常可以接受稳定的基因ID作为输入,帮助你理解这些基因可能的生物学角色。
总的来说,在没有基因名称的情况下,使用GENE STABLE ID是一个稳妥的选择,并尽可能利用现有的资源来补充和更新基因的注释信息。这样不仅可以保证分析的准确性,还能随着科学知识的进步更新你的数据集。
WGCNA
链接:https://caiyun.139.com/m/i?065CuHVqdNyoC
提取码:boJ1
复制内容打开移动云盘PC客户端,操作更方便哦
里面有实例的数据
好了解析我的
User
WGCNA中,下面这些方法有什么不同?我要分析这个的FPKM应该选那个以rain包的内置数据集”menetRNASeqMouseLiver”为例作演示,该数据集来自于一项对小鼠昼夜节律转录调控的研究。小鼠饲养在12h光照/12h黑暗的24h周期中,饲养一段时间后,每隔4h取样一次,通过高通量测序获得的小鼠肝脏基因表达谱的时间序列。
count
expected count
normalized count
peak area (metabolomics)
protein abundance
在加权基因共表达网络分析(WGCNA)中,选择合适的数据处理方法是至关重要的,尤其是在处理不同类型的生物信息数据时。下面是您提到的几种数据类型的简单概述,以及它们在分析小鼠肝脏基因表达谱数据时的适用性:
Count: 这是最原始的测序数据形式,直接从高通量测序实验中得到,表示特定序列(如基因或转录本)的读取次数。在处理RNA-Seq数据时,count数据通常是分析的起点。
Expected Count: 在某些RNA-Seq数据分析软件中,如DESeq或edgeR,期望计数是经过处理的计数数据,用于校正测序深度和RNA组成等因素的影响。它是用于差异表达分析的一种中间步骤数据。
Normalized Count: 正则化计数是对原始计数数据进行处理,以消除样本间测序深度差异和基因长度差异的影响,使不同样本间的数据可比。常见的正则化方法包括TPM (Transcripts Per Million)、FPKM (Fragments Per Kilobase of transcript per Million mapped reads) 和RPKM(Reads Per Kilobase of transcript per Million mapped reads)。
Peak Area (Metabolomics): 在代谢组学研究中,峰面积通常用来定量代谢物的丰度。这种方法不适用于基因表达数据分析,因为它是专门为代谢组分析设计的。
Protein Abundance: 蛋白质丰度数据来自蛋白质组学研究,通常通过质谱等技术获得。这种数据类型用于研究蛋白质表达水平,而不是RNA表达。
对于您的具体案例,分析小鼠肝脏的基因表达谱(FPKM数据),最适合的数据类型是Normalized Count,尤其是FPKM值。FPKM正则化能够调整测序深度和基因长度的差异,使得不同样本间的表达水平可比,这对于构建加权基因共表达网络尤为重要。
下面,我将以WGCNA包的内置数据集”menetRNASeqMouseLiver”为例,展示如何使用FPKM数据进行分析。首先,我们需要加载必要的R包并准备数据:
接下来的步骤将涉及数据的检查、过滤低表达的基因、样本聚类、构建基因表达网络、模块识别等。因为您的问题主要关注数据类型的选择,具体的WGCNA分析步骤会比较复杂,涉及多个阶段的详细操作。如果您需要关于如何使用WGCNA进行具体分析的进一步指导,请告诉我!
在进行RNA-Seq数据分析时,最终获得的FPKM(Fragments Per Kilobase of transcript per Million mapped reads)数据表通常包括了每个基因或转录本在不同样本中的表达水平。选择基因ID还是转录本ID取决于分析的目标和具体需求:
基因ID:使用基因ID意味着您关注的是整个基因水平的表达量。这种方法简化了分析,因为它不区分基因内部的不同转录本。在很多情况下,特别是当研究的重点是基因而非其具体转录变体时,使用基因ID更为常见。此方法适用于大多数比较宽泛的生物学研究,比如疾病相关基因的鉴定、基因表达差异分析等。
转录本ID:使用转录本ID则提供了更高分辨率的表达数据,可以区分同一基因内的不同转录变体。这对于那些特别关心基因转录层面的调控机制的研究非常有用,例如研究特定转录变体的功能、剪接事件的调控等。如果您的研究重点是理解基因的不同转录本如何响应特定条件或处理,那么选择转录本ID会更合适。
在学术论文中的使用:实践中,论文作者会根据研究的具体目的来选择。如果分析的重点是基因层面的差异表达或基因功能研究,通常会选择基因ID。如果研究涉及基因剪接或转录本特异性表达的问题,作者则倾向于使用转录本ID。因此,并没有绝对的“更好”选择,而是根据研究目标和数据分析需求来定。
在大多数情况下,如果您不确定选择哪个,可以考虑研究的焦点:
- 基因水平的研究:选择基因ID。
- 转录本或剪接变异的研究:选择转录本ID。
在提交给期刊时,清晰地说明您的数据处理和分析方法,包括选择基因ID或转录本ID的理由,这对于读者理解您的研究方法和结果至关重要。
count
expected count
normalized count
peak area (metabolomics)
protein abundance
我选择那个方法
对于您想要分析的FPKM数据,来自RNA-Seq实验的,最适合的数据类型是Normalized Count。原因如下:
Count: 这是原始测序数据,直接表示为每个基因或转录本的读取次数。尽管这是分析的起点,但未经过任何形式的标准化,因此不直接适用于跨样本或条件的比较。
Expected Count: 这是一些特定分析工具(如DESeq2或edgeR)计算出的,用于校正测序深度等因素的计数。这种数据虽然进行了一定程度的预处理,但仍然不是最适合跨样本比较的形式。
Normalized Count: 这种数据经过了标准化处理,如FPKM (Fragments Per Kilobase of transcript per Million mapped reads) 或TPM (Transcripts Per Million),考虑了测序深度和基因长度,使得不同样本间的表达量可比。对于RNA-Seq数据,这是进行表达量比较分析的首选类型,因此是您的最佳选择。
Peak Area (Metabolomics): 这是用于代谢组学研究的数据类型,表示代谢物的相对丰度。这种类型与RNA-Seq数据分析无关,因此不适合您的情况。
Protein Abundance: 这是蛋白质组学数据,通常通过质谱等技术得到,用于分析蛋白质的表达水平。这同样与基于RNA的表达数据分析不相关。
总之,对于基于RNA-Seq的表达数据分析,特别是涉及到FPKM数据的情况,Normalized Count是最适合的数据类型。这种数据类型允许您进行更准确和可靠的跨样本表达量比较,是进行表达数据分析和WGCNA等高级分析的基础。
这段信息似乎是从某种生物信息学分析软件或流程中得到的反馈,涉及对表达矩阵的初步检查和处理步骤。以下是该信息的逐句翻译和解释:
“Congratulations!, There is no problem with your expression matrix format, please proceed to the next step”
- 恭喜!您的表达矩阵格式没有问题,请继续进行下一步。
- 这意味着您提交的表达矩阵格式是正确的,可以进行后续的数据分析。
“Notice: It seems your input data is a: non-count expression matrix, it is recommend to select normalized count, peak area (metabolomics), protein abundance or expected count in Format”
- 注意:看起来您的输入数据是非计数表达矩阵,建议在格式中选择正则化计数、峰面积(代谢组学)、蛋白质丰度或期望计数。
- 这说明您的数据不是原始计数数据,而是经过某种形式的预处理或正则化。根据之前的讨论,对于RNA-Seq数据,正则化计数(如FPKM或TPM)是适合的选择。此处也提到了其他类型的生物信息数据(代谢组和蛋白质丰度),但这些不适用于RNA-Seq分析。
“Notice: If readcount is expected count generated by RSEM, Please select expected count.”
- 注意:如果读取计数是由RSEM生成的期望计数,请选择期望计数。
- 这是针对使用RSEM工具处理RNA-Seq数据的用户的特别说明。RSEM是一种流行的软件,用于从RNA-Seq数据中估计基因和转录本的表达水平,生成所谓的期望计数。
“After filtered by conditions: removing all features that have a count of less than say 1 in more than 90% of the samples”
- 经过条件过滤:去除在超过90%的样本中计数少于1的所有特征。
- 这一步骤去除了在大多数样本中几乎不被表达(即表达量低于1的)的基因,以减少噪音和提高分析的准确性。
“Remaining Gene Numbers: 654”
- 剩余基因数量:654
- 经过上述过滤步骤后,数据集中剩余654个基因用于后续分析。
“After filtered by conditions: Genes with MAD ranked top 654 of all expressed genes”
- 经过条件过滤:所有表达基因中,以平均绝对偏差(MAD)排名前654的基因。
- 这说明在进行平均绝对偏差(MAD)排名后,选择了排名最高的654个基因。MAD是衡量数据变异性的一种方式,这可能是为了进一步聚焦于表达变异性最大的基因。
“Notice: The number of Genes you want to retain is greater than the total number of genes after the first filter. The number of genes retained here is equal to the total number after the first filter.”
- 注意:您希望保留的基因数量大于第一次过滤后的总基因数量。这里保留的基因数量等于第一次过滤后的总数量。
- 这表示在过滤条件下,希望保留的基因数量(可能是指定的)并没有因为过滤条件而减少,即所有经过第一次筛选的基因都被保留了。
简而言之,这段反馈说明了表达矩阵的初步处理过程,包括格式确认、数据
类型建议、特定工具的数据选择提示、以及通过过滤减少噪音和关注变异性高的基因的步骤。这些步骤是准备数据以进行更深入分析的重要环节。
WGCNA_2
Congratulations!, There is no problem with your expression matrix format, please proceed to the next step
Notice: It seems your input data is a: non-count expression matrix, it is recommend to select normalized count, peak area (metabolomics), protein abundance or expected count in Format
Notice: If readcount is expected count generated by RSEM, Please select expected count .
After filtered by conditions: removing all features that have a count of less than say 1 in more than 90% of the samples
Remaining Gene Numbers: 3353
After filtered by conditions: Genes with MAD ranked top 3353 of all expressed genes
Remaining Gene Numbers: 3353
Notice: The number of Genes you want to retain is greater than the total number of genes after the first filter. The number of genes retained here is equal to the total number after the first filter.

The power recommended by WGCNA is: 6
If all power values lower than the R square threshold which you set, it means that the power value is an empirical value. At this time, you need to infer a power value based on the results on this plot and check whether it can form a scale-free network.
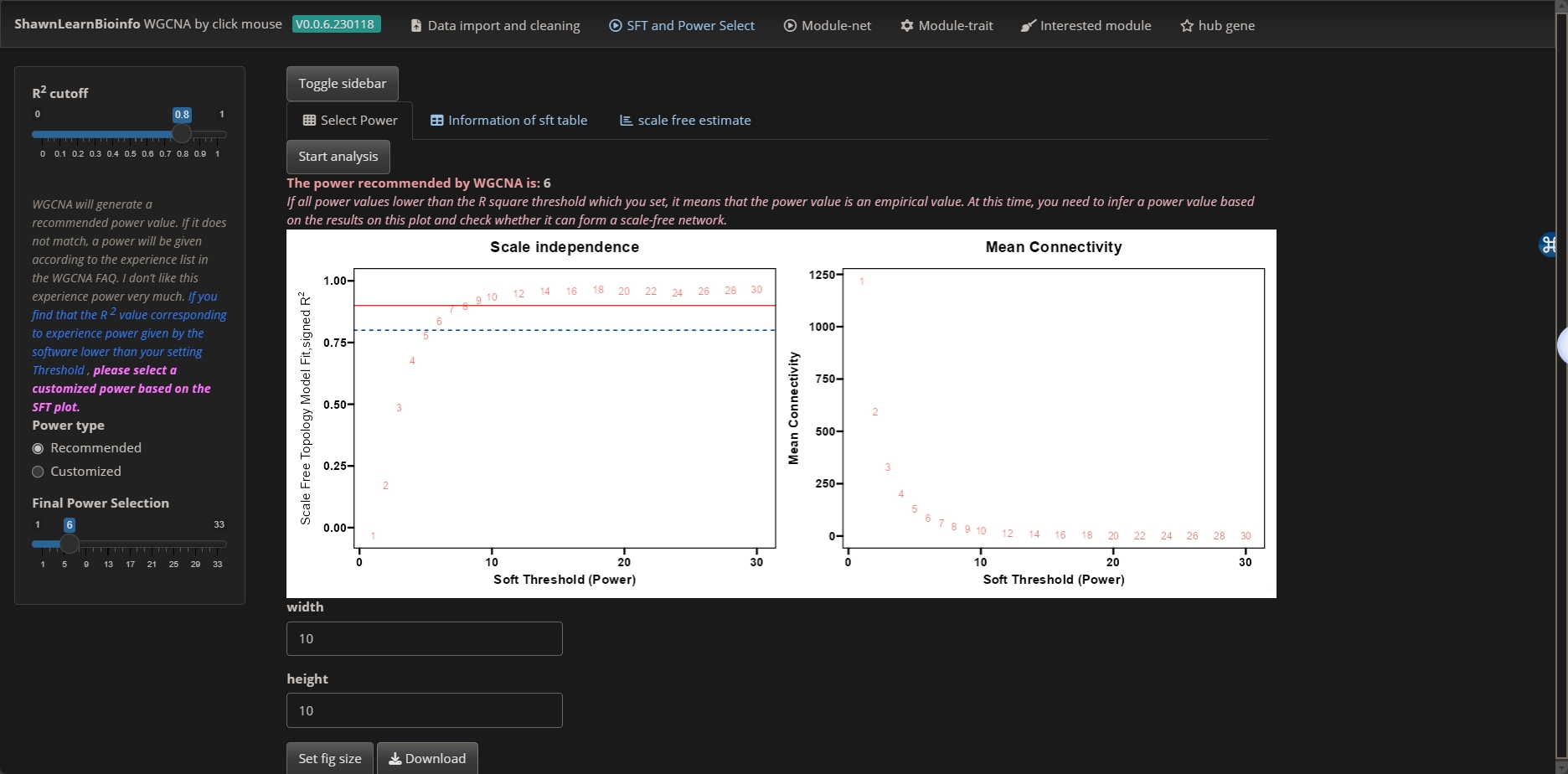
您提供的陈述涉及在加权基因共表达网络分析(WGCNA)中确定适当的power(也称为软阈值power)的过程。这个过程对于构建无尺度网络至关重要,这是WGCNA用于探索基因之间相关性模式的一个关键方面。
在WGCNA中,选择power是基于无尺度拓扑标准的。目标是选择一个使得网络尽可能满足无尺度拓扑的power。这意味着网络的连通性分布的对数-对数图遵循线性模型,这是无尺度网络的标志。在软阈值power与无尺度拓扑模型拟合图中的R^2值(平方相关系数)用于评估这一点。R^2值的常见阈值是0.8,表明网络拓扑大致是无尺度的。
如果所有power值都低于你设定的阈值(在这个例子中,假设阈值是R^2值的0.8),这意味着没有一个测试的power值能够基于你的阈值充分满足无尺度网络的标准。当这种情况发生时,它表明选择的经验值(例如,你的例子中的power为6)是一种妥协。此时,你会基于对无尺度拓扑模型拟合图的分析来推断一个power值。你会寻找一个达到最高模型拟合(R^2值)的最低power,尽可能接近或高于你的阈值。如果不可能,选择一个在实现高R^2值和保持网络构建的生物学相关性及实用性之间提供最佳折中的power。
在选择了一个经验power值之后,你会继续构建网络,并评估它是否形成了无尺度拓扑。这涉及检查网络的特性,如节点连通性分布,是否与无尺度网络所期望的一致。当理想条件(R^2阈值满足)未实现时,仍然需要采取这种经验方法,以构建一个有意义且具有生物学信息的网络。
阈值0.09的时候有9个,0.08的时候有6个,我应该选那个
在选择WGCNA中的soft-thresholding power时,目标是找到一个既能使网络接近无尺度拓扑又能保持网络稳定性和有意义性的值。根据您提供的信息,当阈值设定为0.09时有9个powers满足条件,而阈值设定为0.08时有6个powers满足条件。
通常,选择一个较高的R^2值作为阈值可以帮助确保网络更接近无尺度拓扑,这对于后续的分析来说是有益的。然而,也需要考虑到随着power值增加,网络的稀疏性也会增加,这可能会导致丢失重要的生物学信息。因此,选择合适的power值需要在保持网络无尺度拓扑特性和避免过度稀疏化之间找到平衡。
- 如果在阈值为0.09时有9个power值满足条件,这意味着这些power值能够构建出与无尺度拓扑更为接近的网络,可能更倾向于选择一个在这些值中的较低值,以便保持网络的连通性不被过度稀疏化。
- 如果在阈值为0.08时有6个power值满足条件,而且这6个值中包含了在阈值为0.09时筛选出的9个值中的较低值,那么选择这些较低的power值可能是一个更好的折中方案,因为它们在保持较高的拓扑模型拟合度的同时,避免了选择过高的power值导致网络过于稀疏。
综上所述,选择具体的power值应该基于以下考虑:
- 选择能够使网络拓扑接近无尺度的power值。
- 保持网络的连通性,避免因为选择过高的power值而使网络过于稀疏,可能导致重要的生物学信号丢失。
建议选择阈值为0.09时满足条件的最低的几个power值中的一个,这样既能保证网络的无尺度特性,又能在一定程度上避免网络过度稀疏化。然而,最终的选择也应该结合具体研究背景和其他实验结果进行综合考虑。
这个软阈值的东西,我还有点迷,能用有趣的方式解释清楚吗
当然可以,让我们用一个有趣的比喻来解释WGCNA中的软阈值(soft-thresholding power)概念:
想象你是一位组织派对的主人,你的目标是让派对既热闹又让每个人都能找到志同道合的朋友。在这个比喻中,派对的每个参与者代表一个基因,而参与者之间的交谈代表基因之间的连接(即共表达)。
现在,你需要决定一个派对规则(即软阈值),来决定谁应该与谁交谈。如果规则太宽松(低软阈值),那么每个人都会与每个人交谈,这导致派对变成一个大杂烩,很难看出哪些人是真正有共同话题的。相比之下,如果规则太严格(高软阈值),那么可能只有极少数人在交谈,大多数人都被排除在外,这样你就错过了许多有趣的交流可能。
软阈值的目标就是找到一个完美的平衡点,让派对既不会过于喧闹也不会过于冷清。这个平衡点就像是设置一个“交谈强度”的门槛,只有当两个人之间的共同话题(即基因之间的相关性)超过这个门槛时,他们才能开始交谈。这样,你就能确保派对上的交谈是有意义的,让有相似兴趣的人聚在一起,形成一个个小团体(即基因共表达模块)。
在WGCNA中,选择合适的软阈值就像是找到那个能让派对既活跃又有组织的“交谈强度”门槛。太低,所有基因似乎都随机连接;太高,重要的连接可能被忽视。通过调整软阈值,我们试图创建一个既反映真实生物学模式又能去除随机噪声的网络,就像是组织一个既热闹又富有成效的派对。
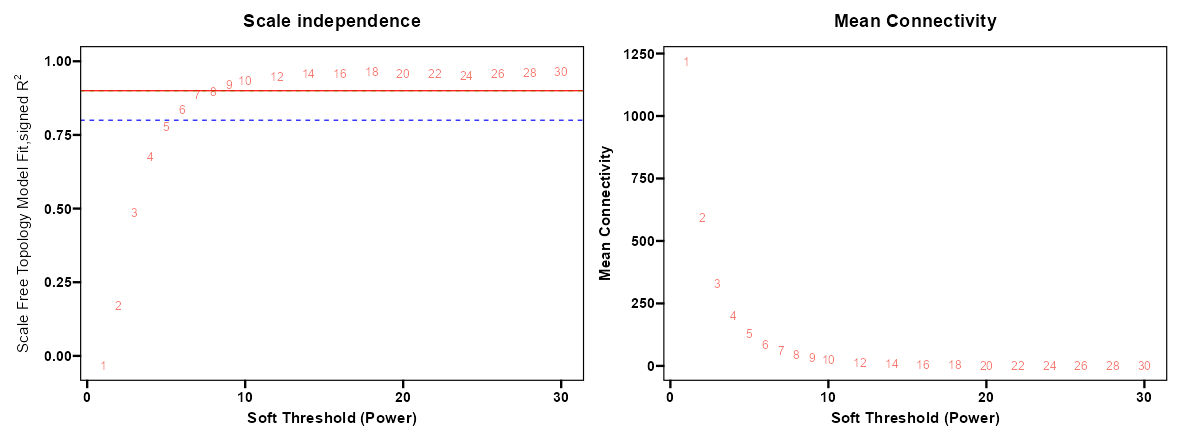
这个图片,能解释下吗?软阈值选什么合适?
这张图片显示的是两个图表,用于在WGCNA(加权基因共表达网络分析)中帮助确定合适的软阈值(soft-thresholding power)。
左侧的图表显示的是“Scale independence”(尺度独立性),它反映了不同的软阈值(power值)如何影响网络拓扑的无尺度特性。无尺度网络是指少数节点(称为枢纽节点)与许多其他节点有连接,而大多数节点则仅与少数节点相连。这种特性通常通过拟合指数曲线到节点度分布来检测。在图表中,纵轴表示拟合的好坏(用R^2表示),横轴表示不同的软阈值(power值)。理想情况下,我们希望选择一个power值,使得R^2值尽可能接近1,这表明网络具有很强的无尺度特性。通常,选择R^2值达到0.8或以上的最小power值是一个好的开始。在这个例子中,power值为6时,R^2值首次达到了0.8以上,因此这可能是一个合适的选择。
右侧的图表显示的是“Mean Connectivity”(平均连通性),它反映了不同的软阈值如何影响网络中节点的平均连通度。随着power值的增加,平均连通度会下降,这表示网络变得更稀疏。在这张图中,我们可以看到,随着power值的增加,平均连通度急剧下降。
总体而言,我们要寻找一个平衡点,该点既有较高的尺度独立性R^2值,又不会导致平均连通性过低,从而避免网络变得过于稀疏。根据这个例子,选择power值为6似乎是一个合适的妥协,因为它提供了一个接近无尺度网络的R^2值,同时保持了较高的平均连通度。然而,最终选择应考虑所有的生物学和统计标准,并结合具体的研究目标和数据特性。
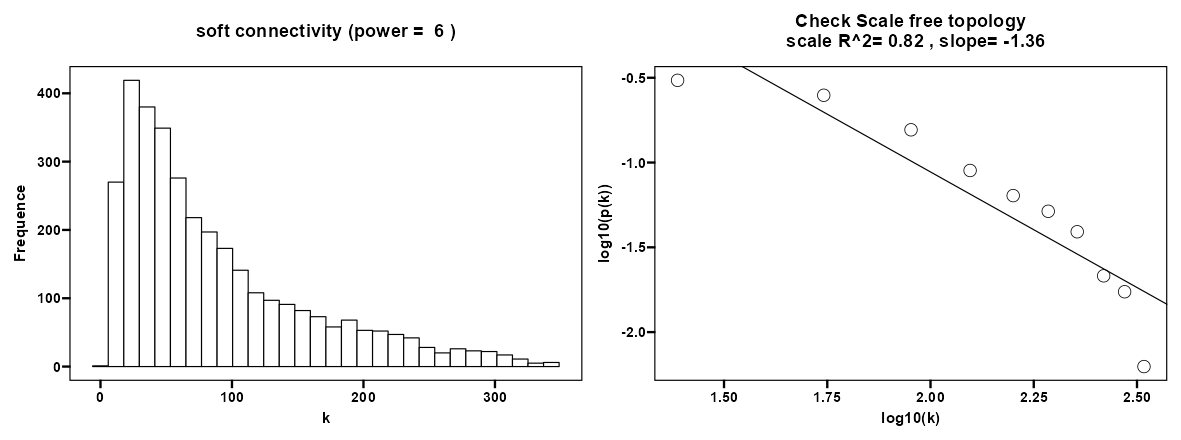
软阈值3.png
这幅图展示了在软阈值(power = 6)下,基因共表达网络的两个关键特性:节点的连通度分布和无尺度拓扑检查。
左图是连通度分布直方图。横轴 “k” 代表节点的连通度,即一个节点与多少其他节点相连。纵轴 “Frequency” 代表具有特定连通度的节点数。这个分布看起来呈现长尾分布,这是无尺度网络的典型特征,即大多数节点只有少量的连接,而少数节点(称为枢纽节点)有很多连接。
右图是检查无尺度拓扑的散点图。横轴是节点连通度的对数值,纵轴是节点连通度频率的对数值。在理想的无尺度网络中,这个图形呈现出负的线性关系。图中的直线是对散点的线性拟合,其斜率(slope)和确定系数(R^2)用来评估网络的无尺度特性。在这里,R^2=0.82表示这个线性模型与数据的吻合度较好,而斜率-1.36意味着在对数对数尺度上,节点的连通度和频率之间存在负相关。
根据右图提供的数据,这个网络的R^2值是0.82,意味着节点连通度分布和无尺度网络的理想分布吻合得很好,网络展示了良好的无尺度特性。因此,在这种情况下,power=6是一个合适的软阈值,因为它不仅提供了足够高的尺度无关性(R^2>0.8),也表明网络在这个阈值下呈现出期望的无尺度分布。这个斜率接近于许多真实世界网络的斜率范围,通常在-1到-3之间。这表明网络的枢纽节点和周边节点之间可能存在合适的比例关系,有利于后续的模块检测和生物学解释。
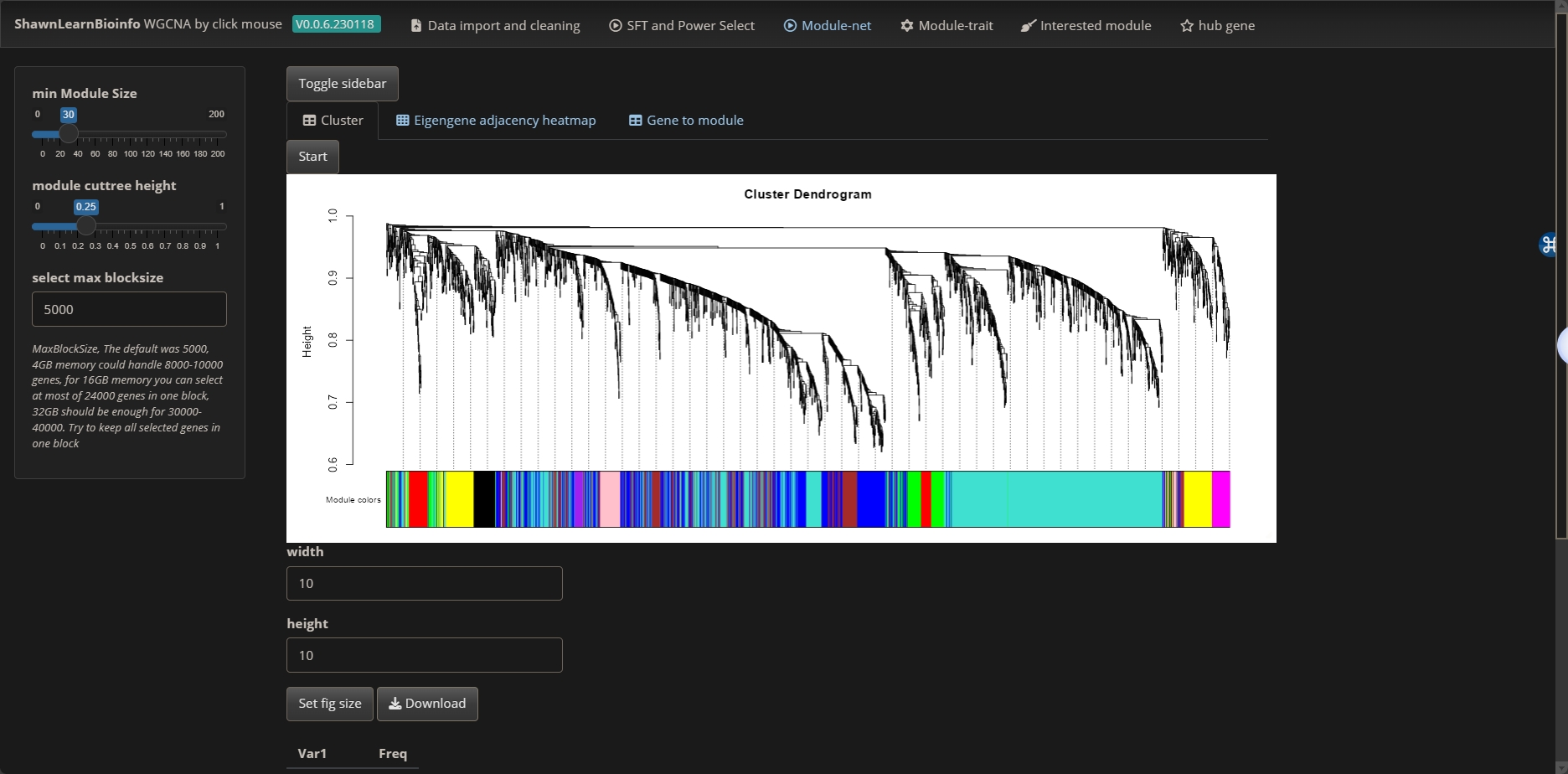
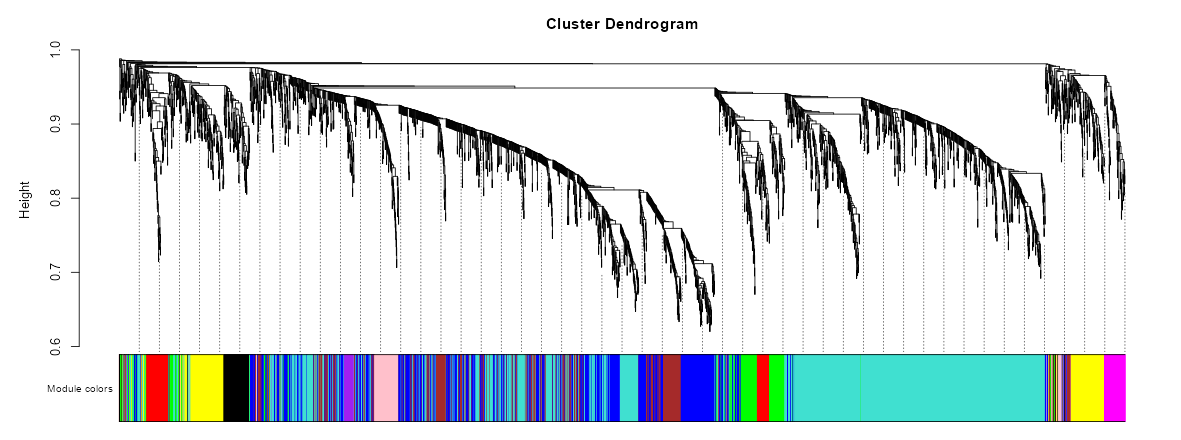
这个图是一个基因表达数据的聚类树状图(dendrogram),它在WGCNA(加权基因共表达网络分析)中用于展示和识别基因模块。在这种分析中,基因被组织成不同的模块,这些模块是基因之间高度相关的集群,可能对相同的生物学功能或疾病状态有贡献。
在这个聚类树状图中:
- 每个垂直的线条代表一个基因。
- 横轴下方的彩色条表示不同的模块。每种颜色代表一个模块,即一组表达模式相似的基因的集合。
- 树状图上的“Height”指的是聚类时基因间差异的度量。这可以被看作是基因表达模式相似性的逆指标:高度越低,表明基因间的相关性越高。
- 树状图上方的水平线(通常称为切割线)表示模块检测的阈值。这条线下方的分支定义了模块。如果分支跨越了这条线,那么它就被认为是独立的模块。
在进行模块识别时,分支越长,表示在该模块内部的基因相似性越高。不同颜色的区块帮助我们快速识别和区分不同的基因模块,它们在后续的分析中可以被用来关联到特定的生物学特性或疾病状况。
这张图为后续的生物信息学分析提供了一个强有力的起点,通过这些模块,研究者可以探究哪些基因表达模式是共同受到调控的,这些模式如何关联到生物学的路径和过程。
实验是:以rain包的内置数据集”menetRNASeqMouseLiver”为例作演示,该数据集来自于一项对小鼠昼夜节律转录调控的研究。小鼠饲养在12h光照/12h黑暗的24h周期中,饲养一段时间后,每隔4h取样一次,通过高通量测序获得的小鼠肝脏基因表达谱的时间序列。
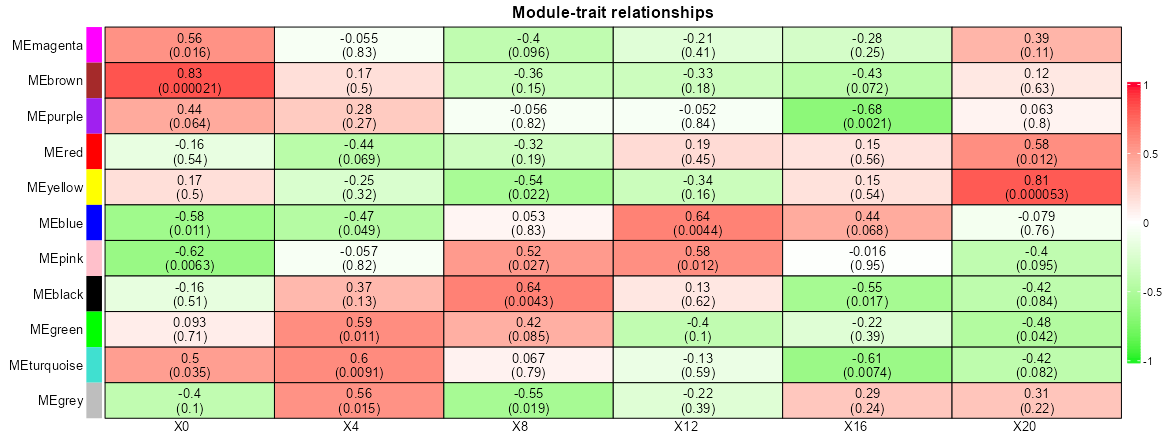
WGCNA时间作为表型的图,下面是时间,帮我分析一下
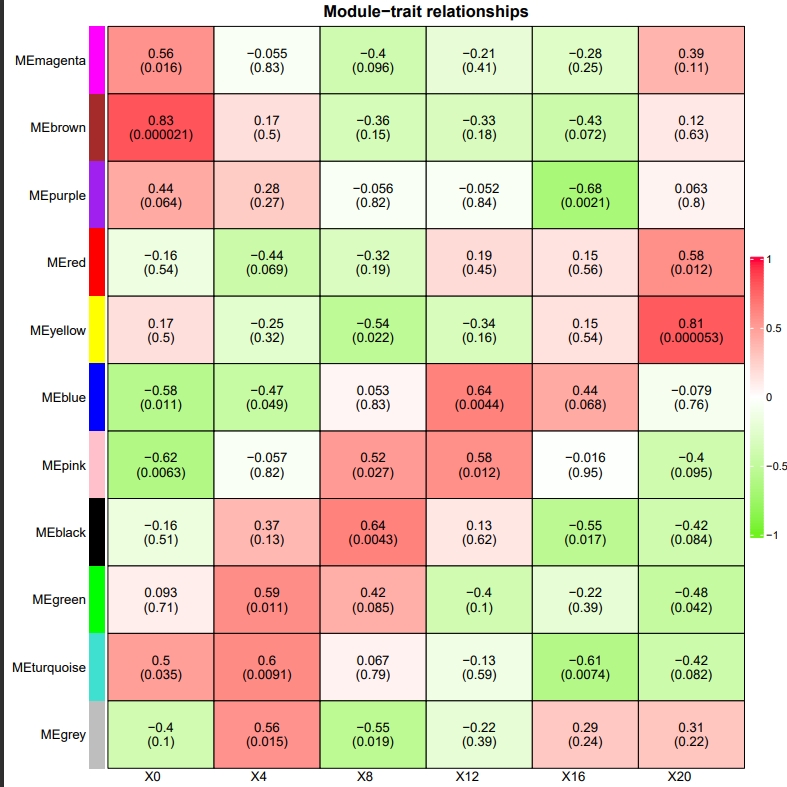
这张图像看起来是显示了加权基因共表达网络分析(WGCNA)中的模块-性状关系的热图。在WGCNA中,基因被聚集成模块(通常用颜色表示),然后这些模块与外部性状或表型相关联。这里的性状是小鼠昼夜实验中的时间点。
热图上的每个单元格代表了基因模块(在y轴上以颜色名称列出,如MEbrown,MEblue等)与特定时间点(在x轴上列出为X0,X4,X8,X12,X16,X20,表示在昼夜周期的黑暗阶段开始后的小时数)之间的相关性。单元格内的数字代表相关系数,括号中的数字是这些相关性的p值。每个单元格的颜色反映了相关性的强度和方向:红色表示正相关,绿色表示负相关,颜色的强度表示相关性的强度(颜色的强度从-1到1,右侧有一个比例尺)。
为了更清楚地分析:
- 观察单元格的颜色:鲜红色的单元格表示强烈的正相关,鲜绿色的单元格表示强烈的负相关,白色或浅色的单元格表示关系弱或无相关。
- 考虑数字:接近1或-1的相关系数表示关系强,接近0的相关系数表示关系弱。
- 考虑p值:较小的p值(尤其是低于常见显著性阈值如0.05的)表明这些相关性在统计上是显著的。
分析图像:
- MEbrown 与X0(黑暗阶段的开始)有很强的正相关,表明这个模块中的基因在这个时间点表达活跃。
- MEyellow 和 MEturquoise 在X20显示出很强的正相关,表明这些模块中的基因在周期的20小时时表达活跃。
- MEblue 和 MEpink 在多个时间点显示出一致的正相关,表明这些基因在黑暗阶段的不同时间都有活跃的表达。
- MEred,MEblack,MEgreen 和 MEgrey 在几个时间点显示出负相关,表明这些模块中的基因在这些特定时间的活动较少。
需要注意的是,这是一个高层次的解释。详细的生物学解释需要理解每个模块中基因的功能及其在昼夜节律中可能扮演的角色。
从这个颜色编码的热图中,我们通常会寻找不同时间点间一致的相关性,这表明基因表达如何随着昼夜周期的变化。以下是我们可以从这个热图中推导出的规律:
特定时间的表达:
- 某些模块在特定时间高度表达。例如,MEbrown模块在黑暗阶段开始(X0)时有很强的正相关性,表明这个模块中的基因可能涉及启动与昼夜周期黑暗阶段相关的活动。
昼夜节律模式:
- 像MEyellow和MEturquoise这样的模块在黑暗阶段结束时(X20)显示出强烈的表达,这可能表明这些基因在为光照阶段准备或者结束黑暗阶段的典型活动中发挥作用。
- MEblue和MEpink模块在多个时间点表现出正相关,这可能表明这些模块中的基因在整个昼夜周期中被调控,且可能在维持昼夜节律中起到关键作用。
负相关的基因模块:
- 如MEred、MEblack、MEgreen和MEgrey等模块在多个时间点显示出负相关,意味着这些时间点这些基因活性降低,它们可能在与正相关模块启动的过程相反的活动中起作用。
统计显著性:
- 括号中的p值帮助我们理解相关性的统计显著性。p值低于0.05(通常被认为是统计显著性的阈值)表明相关性不太可能是由随机机会造成的。例如,MEbrown在X0的p值非常低,增强了我们对观察到的相关性的信心。
无相关性的模式:
- 颜色非常浅且p值高的单元格表明该模块与时间点之间没有显著的相关性。这些基因可能是恒定表达的(不管时间如何都保持恒定水平),或者可能受昼夜周期之外的其他因素影响。
这些观察帮助我们形成了关于不同基因模块在昼夜节律中角色的假设。这样的规律可以用来指导进一步的研究,比如实验验证或调查每个模块内基因的具体功能。准确确定这些发现在昼夜生物学背景下的重要性,需要进行更深入的分析并结合生物学专业知识。
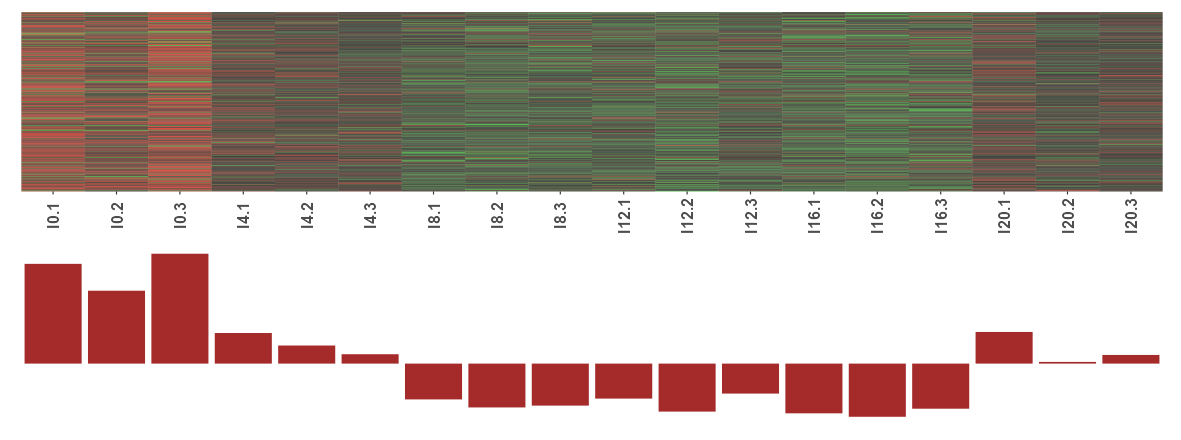
这个图由两个部分组成:顶部的热图和底部的条形图。
热图(Heatmap):
- 热图通常用于展示大量数据的表达模式。在这个热图中,每一列看起来代表一个时间点的样本,而每一行代表一个基因。时间点如10-1, 10-2, 10-3等可能表示在10小时时刻的不同复制或者是不同个体的样本。
- 颜色通常表示基因表达的水平,其中红色可能表示高表达,绿色表示低表达,而黑色可能表示中等表达水平或者是基线表达水平。
- 这个热图显示了在不同时间点基因表达的变化。如果我们关注某一行(即某一个基因),颜色的变化将表示该基因随时间的表达水平变化。
条形图(Bar Chart):
- 底部的条形图似乎表示的是每个时间点样本中表达变化的总结,这可能是一个基因表达差异的统计量,例如基因表达的方差或标准差。
- 高的条形表示该时间点的表达水平变异性大,而低的条形表示表达水平变异性小。这可以指示出,在某些时间点(如图中的10小时时刻和16小时时刻),基因表达的变化更为显著。
总的来说,这个组合图提供了一个关于棕色模块内基因表达随时间变化的总体概览,以及每个时间点表达变异性的量化。这样的信息对于理解基因在特定时间点的行为和它们在生物学过程中的角色是非常有用的,特别是在昼夜节律和时间序列实验中。
这个图,可以和上面的MEbrown模块在黑暗阶段开始(X0)时有很强的正相关性,联系到一起说吗?
是的,这两个图确实可以联系在一起解释。
从你提供的第一张图(模块-性状关系热图)中,我们看到MEbrown模块在黑暗阶段开始(X0)时有很强的正相关性。这表明在这个时间点,MEbrown模块中的基因表达活性增强。
现在,这第二张图展示了棕色模块(MEbrown)中基因的表达模式。顶部的热图显示了不同时间点的基因表达水平,而底部的条形图显示了每个时间点的表达变异性。如果顶部的热图中在X0时间点的垂直列显示了大量红色(表达高),那么它与MEbrown模块在X0有强相关性的观察结果是一致的。这说明在黑暗阶段开始时,这些基因的表达确实是活跃的。
同时,如果在条形图中X0时间点的条形较高,则意味着在该时间点MEbrown模块的基因表达变异性较大,这可能表明不同的基因在这个时间点响应程度不同或者基因表达受到了其他调控因素的影响。
因此,结合这两张图,我们可以推断出在昼夜节律实验中,MEbrown模块的基因在黑暗阶段开始时不仅表达活跃,而且表达的变异性也较大,这可能对理解这些基因在昼夜节律中的角色提供了重要线索。
然后,筛选枢纽基因,GS值和KME 值均设置大于0.8,为什么要这么设置,能详细说说吗?
在WGCNA(加权基因共表达网络分析)中,筛选枢纽基因(hub genes)是为了找到在基因共表达网络中最重要、最中心的基因。枢纽基因通常认为是网络中最有可能对生物学过程产生重要影响的基因。在这种分析中,GS(基因显著性,Gene Significance)和KME(模块成员关系度,Module Membership)是两个核心的指标。
GS(基因显著性): 这个指标反映了单个基因表达水平与外部性状(如治疗条件、疾病状态或者本例中的时间点)之间的相关性。GS值高意味着基因表达水平与外部性状的相关性强,这表明这些基因可能在生物学功能上与该性状密切相关。
KME(模块成员关系度): 这个指标衡量的是单个基因与其所在模块的表达型之间的相关性。KME值高意味着这个基因与整个模块的基因表达模式高度一致,即这个基因在其所在模块中的地位很中心。
当我们将GS和KME的阈值都设定为0.8时,这代表我们在寻找那些不仅与特定外部性状有很强相关性(GS值高),而且在其所在模块中处于中心地位的基因(KME值高)。设定高阈值是为了确保选出来的枢纽基因在统计上和生物学上都是高度显著的。
- 从统计角度看,较高的阈值可以减少假阳性的数量,即减少那些由于随机噪音而偶然显示出高GS和KME值的基因。
- 从生物学角度看,我们希望找到对研究的特定性状贡献最大的基因,这些基因不仅与性状强相关,而且在模块中发挥核心作用。
然而,阈值的设定可能会根据实验设计和具体的研究目标有所不同。0.8是一个相对保守的高阈值,能够帮助我们筛选出最可能是真正的枢纽基因的那一小部分基因。在不同的情境下,研究者可能会调整这个阈值以满足特定的需求。例如,如果想要得到更多可能的候选基因,可以选择一个较低的阈值。
可视化
是的,在使用WGCNA分析后,常常需要对结果进行可视化,这有助于直观地理解和展示数据。你可以使用几种不同的软件和工具来进行这种可视化。
R软件: WGCNA包本身就是在R语言环境中开发的,提供了一系列绘制网络图、热图、特征图等的函数。例如,可以使用
plotDendroAndColors函数来可视化基因模块和特征的关系,或者用visNetwork来展示网络本身。Cytoscape: 对于更复杂的网络可视化,可以将WGCNA的输出导入到Cytoscape中,这是一个强大的网络分析和可视化工具。在Cytoscape中,可以调整节点和边的样式,添加注释,以及应用各种布局算法来改进网络图的可读性。
Graphviz: 这是另一个可以创建图形网络的工具,它支持多种布局算法,适用于复杂网络的可视化。
Gephi: 这是一个开源的网络分析和可视化软件。Gephi具有交互性,易于使用,并且可以用于大型网络的数据挖掘和分析。
在可视化时,可能会展示基因的连接度、模块成员关系度(KME)、基因显著性(GS)以及基因之间的相互作用。通过这些可视化手段,可以更清晰地识别出模块内的枢纽基因和它们与其他基因的相互关系。
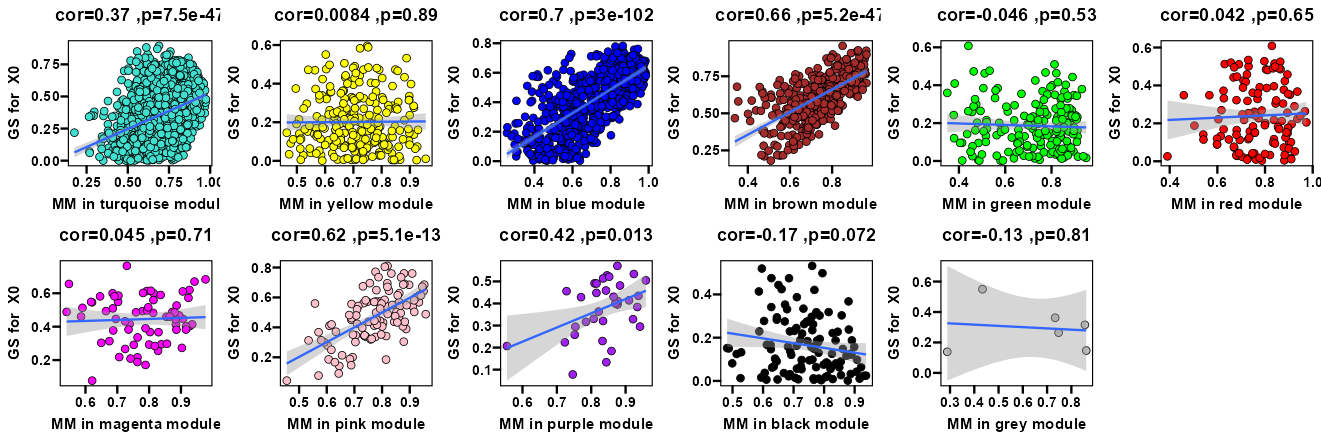
这张图展示的是加权基因共表达网络分析(WGCNA)的结果之一,其中分析了模块成员资格(MM, Module Membership)和基因显著性(GS, Gene Significance)之间的关系。
在WGCNA中,通过模块检测算法,基因被分为不同的模块,每个模块包含相互之间有着高度相关表达模式的基因。模块成员资格(MM)表示某个基因与其所在模块的特征基因(eigengene)表达模式的相关度。基因显著性(GS)则指基因表达与某个外部性状(如疾病状态、生理参数等)的相关度。
每个小图表示一个颜色标记的模块中的基因,横轴是MM值,纵轴是GS值。每个图上的点代表一个基因。越接近模块特征基因的基因,在x轴上的MM值越高;与外部性状相关度越高的基因,在y轴上的GS值越高。
图中还显示了MM和GS之间的皮尔逊相关系数(cor)和p值。相关系数的值范围在-1到+1之间,接近+1表示强正相关,接近-1表示强负相关,而接近0表示没有明显的线性关系。p值用于测试这种相关性是否统计上显著,通常在生物统计学中,p值小于0.05被认为是显著的。
例如,蓝色模块显示了较高的正相关(cor=0.7)和非常显著的p值(p=3e-102),这意味着这个模块中的基因与外部性状的相关性很强。相比之下,黄色模块的基因和外部性状之间几乎没有相关性(cor=0.0084,p=0.89)。
是的,你的理解是对的。在WGCNA分析中,cor(皮尔逊相关系数)较大表明模块中的基因与外部性状的相关性较强,这可能意味着这个模块在生物学功能上与性状密切相关,值得进一步的研究。
关于cor值的选择,没有一个固定的标准。在实际应用中,研究人员可能会根据研究目的和数据的特性来决定cor的阈值。通常,一个cor值至少需要大于0.2或0.3才被认为有一定的生物学意义。然而,即使相关性较低,如果p值非常显著(远小于0.05),这也可能表明有一个值得关注的弱相关性。
在WGCNA中,更重要的是看p值是否显著。p值小于0.05通常被认为模块与性状有显著相关性。在一些情况下,研究人员可能会选择更严格的p值阈值(例如0.01或更低),以减少假阳性发现的几率。如果你的数据集很大,那么即使是较小的cor值,只要p值足够小,也可能是重要的。
在解释这些分析结果时,重要的是要将统计显著性和生物学显著性结合起来考虑。一些模块可能统计上显著但生物学上不重要,反之亦然。因此,WGCNA的结果通常需要通过进一步的实验研究来验证。
WGCNA进行GS值和KME 筛选枢纽基因的时候,筛选的都是 某个性状与某个模块 的基因吧?’
是的,WGCNA(加权基因共表达网络分析)在进行GS(基因显著性,Gene Significance)值和KME(模块成员关系度,Module Membership)筛选枢纽基因的时候,通常关注的是某个性状(如疾病状态、表型特征等)与特定模块之间的相关性。这个过程涉及两个关键步骤:
基因显著性(GS):GS值衡量的是单个基因表达与外部性状之间的相关性。这可以帮助识别那些与特定性状显著相关的基因。性状可以是任何可量化的生物学特征,比如特定的疾病状态、生理条件或者治疗反应等。
模块成员关系度(KME):KME值衡量的是单个基因与其所在模块的表达模式之间的相关性。高KME值的基因被认为是模块内的核心(或枢纽)基因,这意味着这些基因在其所在模块的表达活动中扮演着中心角色。
通过结合这两个指标,研究者可以筛选出那些既与特定性状显著相关,又在其所在模块中发挥中心作用的枢纽基因。这些枢纽基因可能在相关性状的生物学机制中发挥重要作用,因此是潜在的研究和治疗目标。
举个例子,如果某个模块的表达模式与疾病状态高度相关,那么该模块内的高GS和高KME值的基因可能是疾病的关键调节因子或标志物。通过研究这些基因,可以更深入地理解疾病的分子机制,并可能指导新的治疗方法的开发。
确实,你提到的两种方法用于筛选枢纽基因(或称为Hub基因)的过程中,虽然都是为了找到网络中的关键基因,但它们侧重的角度和方法有所不同。下面我会解释这两种方法的差异以及Cytoscape中Hub基因的含义。
WGCNA中的枢纽基因
在WGCNA(加权基因共表达网络分析)中,枢纽基因的筛选是基于统计学上与特定性状相关的度量,即GS(基因显著性)和KME(模块成员关系度)。这里,枢纽基因是指在特定模块内与性状显著相关且在该模块表达模式中占据中心地位的基因。
- 基因显著性(GS):衡量基因表达与性状之间的相关性。
- 模块成员关系度(KME):衡量基因与其所在模块表达模式的一致性。
Cytoscape中的Hub基因
而在Cytoscape这个网络分析工具中,特别是使用CytoHubba这个插件时,Hub基因的识别主要是基于网络拓扑特性,如节点的度(Degree)。节点的度是指节点连接的边的数量,它是网络中一个节点的基本且直观的中心性指标。
- 度(Degree):一个节点的度表示该节点直接连接的节点数。在基因共表达网络中,度较高的基因意味着它与许多其他基因有共表达关系,表明这些基因可能在调控网络中扮演着核心角色。
因此,在Cytoscape中通过CytoHubba插件等工具识别的Hub基因,主要是依据网络的拓扑结构来判断的,即这些基因在整个网络中的连接中心性较高。
结论
这两种方法从不同的角度识别枢纽基因:
- WGCNA关注的是基因表达与特定性状的关联以及基因在其所属模块中的角色。
- Cytoscape(使用CytoHubba)关注的是基因在整个网络拓扑结构中的中心性。
虽然两者的出发点和侧重点不同,但最终目的都是为了从大量的基因表达数据中识别出在生物学功能或疾病机制中可能发挥关键作用的基因。在实际应用中,结合使用这两种方法可以从不同角度深入理解基因网络的结构和功能,从而为疾病的研究和治疗提供更全面的信息。
富集分析
虽然吧,但是来
该数据集来自于一项对小鼠昼夜节律转录调控的研究。小鼠饲养在12h光照/12h黑暗的24h周期中,饲养一段时间后,放入黑暗中,每隔4h取样一次,通过高通量测序获得的小鼠松果体基因表达谱的时间序列。获得FPKM值然后WGCNA,这是其中一个颜色基因内的富集情况,帮我分析下,哪些基因主要在做什么活动?
- GO:0000086~G2/M细胞周期的转换
- GO:0007018~基于微管的运动
- GO:0018105~丝氨酸磷酸化
- GO:0051301~细胞分裂
- GO:0007052~有丝分裂纺锤体的组织
- GO:0007059~染色体分离
Term
- GO:0030425~树突
- GO:0005737~细胞质
- GO:0005930~纤毛
- GO:0005874~微管
- GO:0000940~凝缩染色体外动原粒
Term
- GO:0003777~微管马达活性
- GO:0008017~微管结合
- GO:0005524~ATP结合
- GO:0000287~镁离子结合
- GO:0004674~蛋白质丝氨酸/苏氨酸激酶活性
- GO:0030332~周期蛋白结合
- GO:0035091~磷脂酰肌醇结合
这些基因表达谱富集分析结果揭示了小鼠松果体细胞在特定昼夜节律条件下参与的主要生物过程、细胞组分和分子功能。
主要生物过程
- G2/M细胞周期的转换 (GO:0000086):这涉及细胞从G2期进入并完成有丝分裂的M期的调控过程,是细胞分裂中的关键步骤。
- 基于微管的运动 (GO:0007018):这包括依赖于微管的细胞内物质运输,例如,在有丝分裂期间染色体的移动。
- 丝氨酸磷酸化 (GO:0018105):这是一种蛋白质修饰过程,对于调控多种细胞功能,包括细胞周期控制非常重要。
- 细胞分裂 (GO:0051301):这是生物体生长和繁殖的基础,涉及细胞的有序分裂以产生两个遗传物质相同的子细胞。
- 有丝分裂纺锤体的组织 (GO:0007052)和染色体分离 (GO:0007059):这些过程确保在细胞分裂过程中染色体正确分配给子细胞,对于遗传稳定性至关重要。
细胞组分
- 树突、细胞质、纤毛、微管、凝缩染色体外动原粒等组分指示了基因表达的空间定位,涵盖了从细胞结构到更具体的细胞器和微观结构的各个层面,突出了微管和相关结构在细胞分裂和物质运输中的核心作用。
分子功能
- 包括微管马达活性、微管结合、ATP结合、镁离子结合、蛋白质丝氨酸/苏氨酸激酶活性、周期蛋白结合、磷脂酰肌醇结合等功能,反映了这些基因产品在能量转换、信号转导、蛋白质交互作用和调控中的作用。
总的来说,这些富集的GO项揭示了在小鼠松果体细胞中,与昼夜节律相关的基因主要参与细胞周期的控制、细胞分裂过程、微管依赖的细胞内运动以及相关的信号转导和蛋白质修饰。这些过程对于维持生物体内部的时钟机制、响应环境光周期变化以及在细胞级别保证遗传稳定性和适应性至关重要。
上面的用一段话给我总结下,这个时间段
该数据集来自于一项对小鼠昼夜节律转录调控的研究。小鼠饲养在12h光照/12h黑暗的24h周期中,饲养一段时间后,放入黑暗中,每隔4h取样一次,通过高通量测序获得的小鼠松果体基因表达谱的时间序列。获得FPKM值然后WGCNA,这是其中一个MEpink模块基因内的富集情况,帮我分析下,哪些基因主要在做什么活动?
而且,大概可以发现在黑暗阶段开始(X0)时, 与MEbrown模块有很强的正相关性(关联度为0.83,P<0.01),与MEpink模块有很强的负相关性(关联度为-0.62,P<0.01)。
brown模块:
Term
- GO:0018105~丝氨酸肽键磷酸化
- GO:0042026~蛋白质重折叠
- GO:0015031~蛋白质运输
- GO:0046777~蛋白质自磷酸化
- GO:0031647~蛋白质稳定性的调节
- GO:0007283~精子生成
- GO:0007205~蛋白激酶C激活G蛋白偶联受体信号通路
- GO:0045666~神经元分化的正向调节
- GO:0010828~葡萄糖运输的正向调节
- GO:0009235~钴胺素代谢过程
Term
- GO:0005737~细胞质
- GO:0070062~细胞外外泌体
- GO:0005654~核质
- GO:0048471~细胞质围核区域
- GO:0005829~细胞溶质
Term
- GO:0004114~3’,5’-环核苷酸磷酸二酯酶活性
- GO:0016740~转移酶活性
- GO:0046872~金属离子结合
- GO:0044183~涉及蛋白质折叠的蛋白质结合
- GO:0051082~未折叠蛋白质结合
- GO:0003714~转录共抑制蛋白活性
- GO:0051087~分子伴侣结合
- GO:0019901~蛋白激酶结合
gga03083:Polycomb repressive complex
gga04068:FoxO signaling pathway
KEGG(京都基因和基因组百科全书)富集分析是一种常用于生物信息学研究中,用于理解基因集在生物过程、代谢路径或疾病中的作用和地位的方法。下面我将用中文解释你提到的两个通路:“Polycomb抑制复合体”(gga03083)和“FoxO信号通路”(gga04068)。
Polycomb抑制复合体(gga03083)
Polycomb抑制复合体(Polycomb Repressive Complex,PRC)是一组蛋白质,它们在调控基因表达中扮演关键角色,尤其是在细胞命运决定和发育过程中。它们通过改变染色质结构来抑制目标基因的表达。
在多细胞生物中,Polycomb蛋白质帮助维持细胞的身份和分化状态,防止不适当的基因表达。这是通过在特定基因上添加表观遗传标记(如甲基化的组蛋白尾部),从而改变染色质的构形并抑制基因表达来实现的。这种抑制作用对于发育过程中正确的基因沉默模式以及成体组织的稳态至关重要。
FoxO信号通路(gga04068)
FoxO信号通路涉及一组转录因子,这些因子在细胞增殖、寿命延长、应对氧化应激和代谢调节中起重要作用。FoxO家族成员是由外部信号,如胰岛素或生长因子,通过一系列下游效应物(如AKT蛋白激酶)的磷酸化调控的。当它们被激活时,FoxO转录因子可以进入细胞核,激活或抑制一系列目标基因的表达,这些基因与细胞周期调控、凋亡、DNA修复、应激反应和代谢过程有关。
在许多情况下,FoxO转录因子的活性受到负面调节,例如,在胰岛素信号传导增强时,FoxO活性被抑制,这促进细胞生长和分裂,减少细胞死亡。然而,在应对氧化应激和促进细胞修复方面,FoxO的激活是必要的,这表明它们在维护细胞和组织稳态方面发挥着重要作用。
综上所述,Polycomb抑制复合体和FoxO信号通路都是生物体内重要的调控网络,它们通过不同机制控制着细胞的行为和命运,对于发育、维持生理功能和疾病防治具有重要意义。
pink模块
这些术语的中文翻译如下:
Term
- GO:0009411~对紫外线的反应
- GO:0018298~蛋白质-色素体连结
- GO:0007601~视觉感知
- GO:0035518~组蛋白H2A单泛素化
Term
- GO:0042645~线粒体核
- GO:0005739~线粒体
- GO:0031465~Cul4B-RING E3泛素连接酶复合体
Term
- GO:0004614~磷酸葡萄糖变位酶活性
Term
- gga03320:PPAR信号通路
- gga01100:代谢途径
根据提供的信息,我们可以深入分析MEbrown和MEpink模块在黑暗阶段开始(X0时刻)的基因表达模式,以及这些模式反映的生物学活动。
MEbrown模块的基因表达富集分析显示,这个模块涵盖了多种重要的生物过程和分子功能,包括:
- 信号转导和蛋白质调节(如丝氨酸肽键磷酸化、蛋白质自磷酸化和蛋白激酶C激活的G蛋白偶联受体信号通路),这些过程对细胞应对环境变化至关重要。
- 蛋白质质量控制(通过蛋白质重折叠、运输及稳定性调节),确保蛋白质在适当的形态和位置发挥功能。
- 细胞代谢和生物能量(如葡萄糖运输和钴胺素代谢过程),这些过程对于维持细胞能量平衡和应对能量需求变化至关重要。
- 细胞组分包括细胞质、细胞外外泌体、核质等,表明这些基因在细胞不同部位均有表达,涉及广泛的生物学功能。
与此相对,MEpink模块显示的基因表达富集则涉及到:
- 对环境压力的响应,如对UV的反应,这可能在没有光照的条件下变得不那么活跃。
- 特定的代谢途径,如PPAR信号通路和代谢途径,这些可能在黑暗阶段受到调节,以适应能量利用和储存的变化。
- 视觉感知和蛋白质-色素体连结,这些在黑暗环境下可能减少活动,因为它们与光信号的接收和处理相关。
0时刻,松果体细胞内进行的活动及受到抑制的活动概述:
在黑暗阶段开始时,松果体细胞主要活跃在调节信号转导、蛋白质质量控制、以及代谢过程的调整上,反映出细胞在迅速适应黑暗环境的需求。这种适应性表现为MEbrown模块基因表达的增强,尤其是那些涉及能量代谢、细胞信号传递和蛋白质稳定性调节的基因。
相反,与MEpink模块相关的活动,尤其是那些涉及光感应和特定代谢途径的,可能在这个阶段受到抑制。这反映了在没有光照的环境下,细胞减少了对光信号处理和某些代谢路径的依赖,转而利用其他机制来维持能量平衡和适应黑暗。
总的来说,在黑暗阶段开始时,松果体细胞通过增强与信号转导、蛋白质调节和能量代谢相关的基因表达来适应黑暗环境,同时减少对光依赖性过程的活动,以优化其在新的环境条件下的生存和功能。
凌晨阶段开始(X4)时, 与MEturguoise模块有很强的正相关性 (关联度为0.6,P<0.01)
很多
在凌晨阶段开始(X4时刻),与MEturquoise模块表现出很强的正相关性,这表明在这一时间点,松果体细胞内的一系列生物过程和分子功能活跃度增加。这些活跃的过程和功能反映了细胞对黑暗周期后的适应性调整和生理反应。具体地,MEturquoise模块中的基因表达富集分析揭示了以下活动:
主要生物过程和功能
mRNA处理和翻译调控:涉及mRNA剪接(通过剪接体)、翻译调节、翻译负调节和mRNA多聚腺苷酸化等过程。这些过程对于确保在特定生理状态下产生正确的蛋白质至关重要,表明细胞正在积极调整其蛋白质合成机制以适应黑暗环境后的新需求。
蛋白质运输和处理:包括内质网到高尔基体的蛋白质运输、细胞内蛋白质运输、蛋白质折叠、蛋白质稳定化以及蛋白质酶体介导的泛素依赖性蛋白质分解。这反映了细胞在加强其蛋白质质量控制和分配,确保正确的蛋白质被运送到需要它们的地方,并及时去除损坏或不再需要的蛋白质。
细胞应激响应:包括对DNA损伤刺激的反应、I型干扰素介导的信号传导途径的正调节、α干扰素和γ干扰素产生的正调节,以及细胞对病毒的反应。这表明在凌晨阶段,松果体细胞强化了对潜在损伤或感染的防御机制,以保护细胞免受损害。
能量代谢:糖酵解过程、三羧酸循环和线粒体ATP合成耦合电子传递的活跃指示细胞在调整能量产生和利用,以适应在凌晨阶段可能变化的能量需求。
细胞结构和动力学:如细胞骨架组织和细胞形态的调节,这可能与调整细胞架构以适应新的生理状态有关。
细胞组分活跃区域
活跃的细胞组分包括细胞溶质、核斑、细胞质、核质、内质网及其膜、高尔基体、蛋白质酶体等,反映了细胞内部各个部位都在参与这一过程。
分子功能
活跃的分子功能涉及RNA结合、ATP结合、翻译因子活性、GTP结合、未折叠蛋白质结合等,这些功能对于维持蛋白质合成、修饰和分解的动态平衡至关重要。
结论
在凌晨阶段开始(X4时刻),松果体细胞通过MEturquoise模块表现出的一系列生物过程和分子
功能的活跃,展现了细胞为适应持续的黑暗环境而进行的复杂调整。这包括增强mRNA处理和翻译调控、强化蛋白质质量控制、加强应对细胞应激的能力、调节能量代谢,以及调整细胞结构和动力学。这一过程涉及细胞内的多个组分,显示了松果体细胞在凌晨阶段对环境变化的全面响应和适应。
早晨阶段开始(X8)时, 与MEblack模块有很强的正相关性(关联度为0.64,P<0.01)
- GO:0000086~G2/M细胞周期的转换
- GO:0007018~基于微管的运动
- GO:0018105~丝氨酸磷酸化
- GO:0051301~细胞分裂
- GO:0007052~有丝分裂纺锤体的组织
- GO:0007059~染色体分离
Term
- GO:0030425~树突
- GO:0005737~细胞质
- GO:0005930~纤毛
- GO:0005874~微管
- GO:0000940~凝缩染色体外动原粒
Term
- GO:0003777~微管马达活性
- GO:0008017~微管结合
- GO:0005524~ATP结合
- GO:0000287~镁离子结合
- GO:0004674~蛋白质丝氨酸/苏氨酸激酶活性
- GO:0030332~周期蛋白结合
- GO:0035091~磷脂酰肌醇结合
早晨阶段开始(X8时刻),与MEblack模块的强正相关性表明了松果体细胞在这一时间点的特定生物学活动和过程。MEblack模块的基因表达富集分析揭示了关注点主要集中在细胞周期的调控、细胞骨架的动态变化、以及相关的分子驱动力上。这些过程和功能对于细胞的生长、分裂、以及结构的整合性至关重要,反映了早晨阶段细胞在为日间活动准备的复杂调节机制。
主要生物过程和功能
G2/M细胞周期的转换:这一过程是细胞周期中关键的调控点,涉及细胞从G2期向有丝分裂M期的过渡,指示细胞在早晨阶段正积极准备进入分裂。
基于微管的运动:微管是细胞骨架的重要组成部分,参与多种细胞内运输和位置变化的过程,如胞器的定位、信号分子的传递等。
细胞分裂及其相关过程:如有丝分裂纺锤体的组织和染色体分离,这些过程确保了细胞分裂过程中姐妹染色体的准确分离,是维持基因稳定性和细胞功能正常的关键。
细胞组分活跃区域
活跃的细胞组分主要集中在与细胞骨架和分裂有关的结构上,包括微管、树突、纤毛、凝缩染色体外动原粒等,这些都是参与细胞形态变化、信号传导和物质运输的关键结构。
分子功能
涉及的分子功能强调了微管马达活性、微管结合、ATP结合(提供能量)、镁离子结合(参与许多酶的活性调节)等,这些功能对于细胞骨架的动态调节、细胞分裂过程中能量的供应,以及信号传递过程中分子间相互作用的稳定性都是必不可少的。
结论
在早晨阶段开始时,松果体细胞通过MEblack模块展现的生物学活动和过程,揭示了细胞在准备日间活动过程中的关键调节点。特别是在细胞周期的转换、基于微管的动态变化、以及细胞分裂的精确调控方面,显示了细胞为确保在接下来的白天能够有效进行增殖和功能执行,而进行的复杂准备工作。这一过程涉及到了细胞内多个组分的协同作用,以及多种分子驱动力的集成,反映了细胞对环境周期变化的精细适应机制。
中午阶段开始(X12)时, 与MEblue模块有很强的正相关性(关联度为0.64,P<0.01)
Term
- GO:0045773~轴突延伸的正向调节
- GO:0007265~Ras蛋白信号传导
- GO:0006468~蛋白质磷酸化
- GO:0006470~蛋白质去磷酸化
- GO:0015813~L-谷氨酸运输
- GO:0060411~心房间隔形成
- GO:0043066~凋亡过程的负向调节
- GO:0010460~心率的正向调节
- GO:2000766~细胞质翻译的负向调节
- GO:0006750~谷胱甘肽生物合成过程
- GO:0003323~B型胰岛细胞发育
- GO:0060294~纤毛运动参与细胞运动性
- GO:0016485~蛋白质处理
Term
- GO:0055038~循环内体膜
- GO:0009986~细胞表面
- GO:0000139~高尔基体膜
- GO:0016580~Sin3复合体
- GO:0005737~细胞质
- GO:0005886~质膜
Term
- GO:0005515~蛋白质结合
- GO:0005085~鸟苷核苷酸交换因子活性
- GO:0004672~蛋白激酶活性
- GO:0004467~长链脂肪酸-CoA连接酶活性
- GO:0030145~锰离子结合
- GO:0061629~RNA聚合酶II序列特异性DNA结合转录因子结合
- GO:0015183~L-天冬氨酸跨膜转运蛋白活性
- GO:0005524~ATP结合
- GO:0030170~吡哆醛磷酸结合
- GO:0046872~金属离子结合
- GO:0004722~蛋白质丝氨酸/苏氨酸磷酸酶活性
Term
- gga04114:卵母细胞减数分裂
- gga00061:脂肪酸生物合成
中午阶段开始(X12时刻),与MEblue模块的强正相关性揭示了松果体细胞在这一时间点的特定生物学活动和过程,这些活动和过程对于细胞的信号传递、代谢调节、以及细胞功能的执行至关重要。MEblue模块的基因表达富集分析显示,细胞在中午时段主要集中在以下几个方面的调整和响应:
主要生物过程和功能
神经活动调节:轴突延伸的正向调节和L-谷氨酸运输指向了神经系统功能的调整,特别是在促进神经元之间的通信和网络形成方面。
信号传导:Ras蛋白信号传导、蛋白质磷酸化和去磷酸化过程的调节反映了细胞在细胞内信号传递机制上的精细调控,这对于响应外界信号和维持细胞内稳定状态至关重要。
代谢调节:谷胱甘肽生物合成过程、B型胰岛细胞发育与心率的正向调节等,显示了细胞在能量代谢和生理功能调节方面的活跃。
凋亡过程的负向调节和纤毛运动参与细胞运动性:指示细胞在维持生存和适应环境方面的调整,包括通过调控细胞死亡过程和利用纤毛进行物理运动。
细胞组分活跃区域
活跃的细胞组分涵盖了循环内体膜、细胞表面、高尔基体膜、细胞质、质膜等,显示了信号传导和物质转运在细胞不同位置的重要性。
分子功能
活跃的分子功能包括蛋白质结合、鸟苷核苷酸交换因子活性、蛋白激酶活性、ATP结合等,这些都是信号传递、能量转换和细胞内相互作用中不可或缺的功能。
结论
在中午阶段开始时,通过MEblue模块展现的生物学活动和过程,松果体细胞展示了对环境变化的精细调节能力。特别是在信号传递、神经活动、代谢调节、以及细胞生存策略等方面的调整,体现了细胞为适应中午可能面临的环境压力和生理需求变化,而进行的一系列复杂响应。这一过程不仅涉及了广泛的生物过程和分子功能,也反映了细胞内多个组分的协同作用,确保了细胞能够在日间活动的高峰期保持最佳功能状态。
下午阶段开始(X16)时,与MEpurple模块和MEturguoise模块有很强的负相关性,关联度分别为-0.68,-0.61(P<0.01),
MEturguoise模块与4点正相关
purple模块:
Term
- GO:0006906~囊泡融合
- GO:1990573~钾离子跨细胞质膜进口
Term
- GO:0032839~树突细胞质
- GO:0045202~突触
Term
- GO:0000149~SNARE结合
这些GO术语涉及细胞运输和神经功能,包括囊泡与膜的融合过程、钾离子的细胞内运输、神经细胞结构的特定区域,以及与神经递质释放相关的蛋白质相互作用。
下午阶段开始(X16时刻),与MEpurple模块和MEturquoise模块表现出强烈的负相关性,这种负相关性揭示了在此时间点上,松果体细胞内的一系列生物学活动可能受到抑制或减弱。MEpurple模块的基因表达富集分析特别指向了囊泡融合、钾离子跨细胞质膜进口、以及与突触相关的结构和功能,这些过程和组分通常与细胞的信号传递、物质运输和神经活动紧密相关。
被抑制的主要生物过程和功能
囊泡融合:在神经元中,囊泡融合是神经递质释放的关键步骤,对于突触传递和神经信号的传播至关重要。下午时段该过程的负相关性可能反映了神经活动的减少或调节,以适应一天中的不同活动阶段。
钾离子跨细胞质膜进口:钾离子的进出是维持细胞内外电位差和信号传导的重要过程。下午时段这一过程的减弱可能与细胞对能量和电位调节的变化有关,可能是适应或准备进入夜间休息状态的生理调整的一部分。
被抑制的细胞组分活跃区域
- 树突细胞质和突触:这些区域是神经元信号接收和传递的关键部位,下午时段这些区域功能的减弱可能指向了神经系统活动的整体降低,这可能与生物节律和一天中活动强度的自然减少有关。
被抑制的分子功能
- SNARE结合:SNARE蛋白在囊泡融合过程中发挥核心作用,特别是在神经递质的释放过程中。其活动的减少进一步证实了下午时段神经传递活动可能的降低。
结论
下午阶段开始时,松果体细胞内的活动表现出与MEpurple模块和MEturquoise模块的强烈负相关性,这表明了囊泡融合、钾离子跨膜运输、以及与突触相关的结构和功能可能在这一时间段内减少活动。这种变化可能是细胞适应日内周期,准备从日间活动过渡到夜间休息状态的一种调节机制。特别是在神经信号传递和细胞通信方面,显著的调节可能反映了生物节律对细胞功能的影响,以及细胞在一天的不同时间点上对环境和生理需求的响应。
晚上阶段开始(X20)时,与MEyellow模块有很强的正相关性(关联度为0.81,P<0.01)
yellow模块:
GO:1902975有丝分裂DNA复制起始DNA复制
GO:0006260
GO:0000727通过诱导性断裂复制修复双链断裂参与DNA复制的DNA解旋
GO:0006268
GO:0072659蛋白质定位到质膜参与DNA复制的DNA链延伸
GO:0006271
GO:0032508DNA双链解旋细胞周期
GO:0007049
GO:0060402钙离子进入细胞溶质错配修复
GO:0006298
GO:0000381通过剪接体调节替代mRNA剪接DNA复制起始
GO:0006270
GO:1903553~正调节细胞外囊泡组装
Term
GO:0042555MCM复合物核基质
GO:0016363
GO:0071162CMG复合物细胞核
GO:0005634
GO:0016607核斑细胞溶质
GO:0005829
GO:0030173~高尔基膜的整合成分
Term
GO:0017116依赖于单链DNA的ATP依赖性DNA解旋酶活性DNA解旋酶活性
GO:0003678
GO:0003697单链DNA结合转录共抑制剂活性
GO:0003714
Term
gga03030:DNA复制
gga04330:Notch信号通路
gga04144:内吞作用
晚上阶段开始(X20时刻),与MEyellow模块的强正相关性揭示了松果体细胞在夜间准备进入或已经进入了一个特定的生物学活动周期。MEyellow模块的基因表达富集分析强调了DNA复制、细胞周期控制、DNA修复、以及与细胞膜相关的蛋白质定位等过程,这些过程对于细胞增殖、遗传信息的保持和修复、以及细胞通信至关重要。
主要生物过程和功能
DNA复制与修复:包括有丝分裂DNA复制起始、DNA复制、通过诱导性断裂复制修复双链断裂、DNA解旋、DNA链延伸等过程,这些都是细胞准备分裂和确保遗传信息准确传递给子代的关键步骤。此外,错配修复过程确保了DNA复制后的遗传稳定性。
细胞周期控制:涉及细胞周期的正常进行,这是细胞生长、分裂和复制的基础,保证了细胞功能的持续和遗传信息的稳定传递。
蛋白质定位到质膜:这一过程对于维持细胞的结构和功能,特别是在细胞信号传递和细胞相互作用中起着重要作用。
细胞组分活跃区域
- MCM复合物、CMG复合物、细胞核、核斑等,这些组分在DNA复制和细胞周期控制中发挥核心作用,包括复制起始点的识别和激活、DNA解旋,以及RNA处理等。
分子功能
- 依赖于单链DNA的ATP依赖性DNA解旋酶活性、DNA解旋酶活性等,这些功能对于DNA复制过程中的解旋和修复至关重要,确保了复制的准确性和效率。
结论
晚上阶段开始时,松果体细胞通过MEyellow模块表现的一系列生物学活动和过程,展现了细胞在夜间进行DNA复制、修复和准备分裂的活跃状态。这表明在夜间,细胞可能利用相对静态的环境进入一个生长和更新的阶段,以确保细胞和组织的健康和功能维持。这一时期的活动强调了生物体在不同时间段按照其生物钟和环境条件进行优化生物过程的能力,确保了生命活动的连续性和稳定性。
松果体的功能
鸡的松果体是一个位于大脑中的小腺体,它在多种生物体内发挥着重要的作用,尤其是在调节生物节律方面。对于鸡而言,松果体的主要功能包括:
调节生物节律:松果体通过分泌褪黑激素来调节鸡的昼夜节律,这对于它们的生活习性和繁殖周期非常关键。褪黑激素的分泌通常在夜间增加,帮助促进睡眠,并且在一天中的分泌量会随着季节的变化而变化,这对于季节性繁殖的动物来说特别重要。
调节生殖系统:松果体还通过其分泌的褪黑激素影响鸡的生殖系统。褪黑激素的水平变化可以影响生殖激素的分泌,进而影响繁殖行为和生殖周期。这对于在特定季节繁殖的鸡来说尤为重要。
影响免疫功能:一些研究表明,松果体和褪黑激素可能对鸡的免疫系统有正面影响。褪黑激素可能有助于增强免疫反应,提高抵抗疾病的能力。
抗氧化作用:褪黑激素还具有抗氧化特性,可以帮助保护细胞免受氧化应激的损害。这对于维持鸡的整体健康和长寿有积极作用。
总之,鸡的松果体通过分泌褪黑激素,不仅对维持昼夜节律和季节性行为模式至关重要,还对生殖、免疫系统和细胞保护等方面有着广泛的影响。
与节律相关的基因
调节生物节律的基因主要涉及到生物钟机制,这是一套复杂的内部时间跟踪系统,用于调节生物体的日常生理和行为节律,比如睡眠-觉醒周期、饮食习惯、激素分泌等。这一机制在多种生物中都存在,包括人类、动物、植物乃至微生物。以下是一些在调节生物节律中起核心作用的基因:
CLOCK(Circadian Locomotor Output Cycles Kaput):此基因参与控制生物钟的核心反馈回路,通过影响其他基因的表达来调节昼夜节律。
BMAL1(Brain and Muscle ARNT-Like 1,也称为ARNTL):与CLOCK基因互作,形成一个复合物直接调控其他与生物节律相关的基因的表达。
PER(Period)家族:包括PER1、PER2和PER3,这些基因的蛋白质产品参与到CLOCK和BMAL1驱动的反馈回路中,帮助维持24小时的生物节律。
CRY(Cryptochrome)家族:包括CRY1和CRY2,这些蛋白与PER蛋白协同作用,抑制CLOCK和BMAL1活动,形成生物钟的负反馈回路。
TIM(Timeless):在某些生物中,如果蝇,TIM蛋白与PER蛋白相互作用,参与控制昼夜节律。
RORA和REV-ERBα(NR1D1):这两个基因分别编码RAR相关孤儿受体A和反向ErbA受体α,它们在生物钟调节的反馈回路中通过影响BMAL1的表达而起作用。
这些基因和它们编码的蛋白质构成了生物钟的核心机制,通过相互作用形成复杂的反馈回路,维持生物体内部时间的稳定。这一机制使得生物体能够适应环境光照变化,调整其生理和行为以适应昼夜变化。除上述基因外,还有其他多个基因和信号通路参与调节生物的内部时钟,包括那些影响这些核心时钟基因表达的调节因子和信号分子。
在鸡中,松果体与调节生物节律相关的基因参与了褪黑激素的合成、分泌和调节,从而影响了鸡的昼夜节律、季节性繁殖行为和其他与时间有关的生理过程。虽然鸡和其他哺乳动物在生物节律调节机制上有许多共同点,但也存在一些特异性。在鸡中,一些关键的基因包括:
CLOCK:与哺乳动物中相同,CLOCK基因在鸡中也是生物钟机制的核心成分,参与调节其他与生物节律相关的基因的表达。
BMAL1(ARNTL):与CLOCK基因合作,形成复合体,是驱动昼夜节律相关基因表达的关键因子。
PER家族:尤其是PER2和PER3,在鸡中也发现与调节昼夜节律相关。
CRY家族:CRY1和CRY2基因在鸡中同样重要,它们参与到CLOCK/BMAL1复合体的负反馈回路中。
RORs(RAR相关孤儿受体)和REV-ERBs(NR1D1/NR1D2):这些基因通过调节BMAL1和其他节律相关基因的表达参与生物钟的调节。
AANAT(N-acetyltransferase):虽然不直接参与生物节律的核心反馈回路,但AANAT在鸡中特别重要,因为它是褪黑激素合成的关键酶,直接影响松果体内褪黑激素的产生,从而影响生物节律。
HIOMT(Hydroxyindole-O-methyltransferase):在褪黑激素合成路径中也起作用,进一步转化褪黑激素的前体物质。
这些基因和它们编码的蛋白质在鸡的松果体中通过相互作用,调控褪黑激素的合成和分泌,从而影响鸡的生物节律和相关生理过程。需要注意的是,尽管这些基因的作用机制与哺乳动物中的相似,但在鸡和其他非哺乳动物中可能存在一些独特的调控差异。
松果体中褪黑素的合成和分泌是一个复杂的生物化学过程,涉及多个酶和调节因子。以下是一些与松果体中褪黑素合成直接相关的关键基因及其编码的蛋白质:
**Tryptophan Hydroxylase (TPH)**:色氨酸羟化酶是褪黑素生物合成路径中的第一个关键酶,负责将色氨酸(Tryptophan)转化为5-羟色胺(Serotonin)的前体5-羟色氨酸(5-Hydroxytryptophan)。在哺乳动物中,存在两种形式:TPH1和TPH2,其中TPH2在脑中表达,与神经传递有关。
**Aromatic L-amino acid decarboxylase (AADC)**:也称为DOPA脱羧酶,它将5-羟色氨酸转化为血清素(5-HT,即5-羟色胺)。
**Serotonin N-acetyltransferase (AANAT)**:血清素N-乙酰转移酶是褪黑素合成的速率限制酶,负责将血清素转化为N-乙酰血清素。AANAT的活性受到光照周期的调节,这是夜间褪黑素合成增加的关键步骤。
**Hydroxyindole O-methyltransferase (HIOMT)**:也称为褪黑素O-甲基转移酶或COMT,它将N-乙酰血清素转化为褪黑素。这是褪黑素生物合成的最后步骤。
除了这些直接参与褪黑素合成的酶,还有几个因子和基因参与调节这一过程:
G-Protein Coupled Receptor (GPCR) 类如MT1和MT2:这些是褪黑素受体,虽然它们不直接参与褪黑素的生产,但对于褪黑素信号传导和生理效应至关重要。
**Retinoid-related orphan receptor alpha (RORA)**:间接参与调节AANAT基因的表达,影响褪黑素的合成。
Photic input:光感受系统通过视网膜-松果体轴向松果体传递光信号,影响褪黑素的合成。这涉及多种信号分子和途径,包括经典的视觉光感受蛋白质如视蛋白和其他光敏蛋白。
这些基因和蛋白质的表达及其活性的调节复杂地交织在一起,共同控制着褪黑素的日夜节律性生产,从而影响睡眠、情绪、季节性行为等多种生理过程。
除了你提到的这些基因和蛋白质,还有其他几个基因和因素在松果体中的褪黑素合成和分泌过程中扮演着重要角色:
Cyclic AMP (cAMP) 响应元件结合蛋白(CREB):CREB是一种转录因子,通过响应cAMP信号来调控多种基因的表达,包括AANAT。光照通过减少松果体内cAMP的水平,从而影响CREB活性,进而调节褪黑素的合成。
**Pineal gland-specific adenylate cyclase-activating polypeptide (PACAP)**:PACAP是一种重要的神经肽,通过其受体PAC1R在松果体中起作用,可以增加cAMP的水平,从而影响CREB的活化和褪黑素的合成。
**Retinaldehyde dehydrogenase (RALDH)**:在视网膜-松果体轴中,RALDH负责合成视黄酸,视黄酸是一种重要的信号分子,可以通过影响RORA等转录因子来间接调节AANAT的表达。
**Period genes (Per1, Per2)**:这些基因在昼夜节律的调节中扮演重要角色,其蛋白质产物参与到褪黑素合成的调节中。它们通过影响CREB的活性以及其他信号途径来调控AANAT的表达和活性。
**Cryptochrome genes (Cry1, Cry2)**:这些基因编码的蛋白质是光敏蛋白,参与到光周期感应中,通过影响Per基因的表达和其他信号途径来调节松果体中褪黑素的合成。
**BMAL1 (Brain and Muscle ARNT-Like 1)**:作为昼夜节律的关键调节因子,BMAL1与CLOCK蛋白形成复合体,直接或间接地调控包括Per和Cry在内的多个基因的表达,进而影响褪黑素的合成。
这些基因和蛋白质通过复杂的信号网络相互作用,调控松果体中褪黑素的合成和分泌,以适应外界环境的变化,如光照条件的改变,从而维持生物体的昼夜节律和其他相关生理功能。
在褪黑素合成与分泌的调控中,除了上述的基因和蛋白质外,还有一些额外的因素和分子途径可能间接影响松果体功能和褪黑素的生产:
Suprachiasmatic Nucleus (SCN) of the Hypothalamus:虽然SCN不是一个基因,但它是体内主要的生物钟,控制着昼夜节律的大部分调节机制,包括通过神经和体液途径影响松果体的褪黑素合成。
Glutamate and PACAP:作为神经递质,谷氨酸在松果体的功能中也起着关键作用,特别是通过NMDA受体传递信号,调节褪黑素的合成。PACAP(松果体特异性腺苷酸环化活化多肽)如前所述,也通过其受体影响cAMP路径。
**GSK3β (Glycogen Synthase Kinase 3 Beta)**:这是一种蛋白激酶,通过磷酸化多种底物,包括参与调控昼夜节律的蛋白,间接影响褪黑素的合成。GSK3β的活性受到光照的影响,从而参与调节松果体的功能。
**Nuclear Receptor Subfamily 1, Group D, Member 1 (NR1D1, 也称为Rev-Erbα)**:这是一种核受体,通过抑制Bmal1基因的表达来参与昼夜节律的调控,间接影响褪黑素合成。
**Sirtuins (特别是SIRT1)**:这是一类NAD+依赖的去乙酰化酶,它们通过去乙酰化包括BMAL1和PER2在内的蛋白质,参与调控昼夜节律和褪黑素合成。
Insulin and IGF-1 Signaling:胰岛素和类胰岛素生长因子1(IGF-1)信号也被认为能够影响松果体的功能和褪黑素合成,通过影响cAMP信号途径等。
**MicroRNAs (miRNAs)**:最近的研究表明,miRNAs在调节包括褪黑素合成在内的多种生理过程中发挥作用,通过调节其目标基因的表达来影响褪黑素的生产。
这些额外的因素和分子途径提供了对松果体功能和褪黑素合成调控更全面的理解,突显了一个高度复杂和精细调控的网络,涉及多种信号传导途径和分子机制。
PACAP
GSK3β
miRNAs
TPH
AADC
HIOMT
GPCR
RORA
cAMP
PACAP
RALDH
BMAL1
miRNAs
miRNAs
SIRT1 brown
AANAT turquoise
Cry1, Cry2 pink,brown
ASTM
AANAT(芳香族氨基酸N-乙酰转移酶)和ASMT(乙酰基血清素O-甲基转移酶)
turq
AANAT(芳香族氨基酸N-乙酰转移酶)和ASMT(乙酰基血清素O-甲基转移酶)
AANAT、PER2,ROR1、ARNTL(Bmal1)、CLOCK、CLOCK1,PER3、CRY2,CRY1
这些基因都是控制节律的关键基因,我想知道他们是怎么调节的,哪些基因起什么作用,简单易懂的告诉我下
这些基因都是生物体内部生物钟系统的关键组成部分,它们通过复杂的反馈循环来控制和维持生物的昼夜节律。以下是简单易懂的说明:
**AANAT (芳氨酸-N-乙酰转移酶)**:这个酶在褪黑激素的合成中起关键作用,褪黑激素是调节睡眠和昼夜节律的重要激素。
**PER1/PER2/PER3 (Period家族)**:这些基因编码的蛋白质帮助控制生物钟的节律。它们在细胞核中积累,能够抑制Clock和Bmal1蛋白的活性,形成一个负反馈循环。
**CRY1/CRY2 (Cryptochrome家族)**:这些蛋白同样是负反馈循环的一部分,与PER蛋白协同工作,抑制Clock和Bmal1的活性。
ARNTL (Bmal1) 和 CLOCK:这两个基因编码的蛋白质合作形成一个蛋白复合体,能够激活PER和CRY基因的表达,开启生物钟的正反馈循环。
ROR1:这是一类核受体,参与调节Bmal1的表达,进而影响整个生物钟系统。
CLOCK1:虽然CLOCK1不如CLOCK基因那么出名,但也参与了生物钟的调节过程。
在昼夜周期中,ARNTL(Bmal1)和CLOCK蛋白质在日间积聚并激活PER和CRY基因的表达。随着时间的推移,PER和CRY蛋白质累积到一定程度后,会移动到细胞核内并抑制CLOCK和BMAL1的活性,从而降低自己的表达。这个过程大约24小时完成一次,形成了生物体内部的昼夜节律。
简而言之,这些基因和它们编码的蛋白质通过相互作用和反馈循环来控制生物钟,这个时钟再控制着生物的许多日常活动,如睡眠-觉醒周期、体温、代谢和激素水平。
过RAIN筛选到大部分的节律基因,再通过WGCNA分析,发现这些基因被聚类到不同颜色模块中,比如AANAT、PER2被聚类到MEturguoise模块中,ROR1、ARNTL、CLOCK、CLOCK1被聚类
到blue模块中,PER3、CRY2被聚类到brown模块中,CRY1被聚类到pink模块中。
通过RAIN (Rhythmicity Analysis Incorporating Non-parametric methods) 筛选得到的大部分节律基因,然后使用WGCNA (Weighted Gene Co-expression Network Analysis) 分析,将这些基因根据表达模式的相似性聚类到不同颜色的模块中,揭示了几个重要的生物学意义和可能的机制:
基因表达的协同模式:不同颜色模块代表了一组基因表达模式的协同性。基因被聚类到同一模块中意味着它们在时间序列中有着相似的表达模式,可能在生物体的相同生理过程或相同的调控机制下被共同调节。例如,AANAT和PER2被聚类到MEturquoise模块中,可能表明它们在调控昼夜节律中有协同作用。
功能相关性:被聚类到同一模块的基因可能参与相同的生物学功能或生理过程。例如,ROR1、ARNTL(Bmal1的另一命名)、CLOCK和CLOCK1被聚类到blue模块中,这些基因都是昼夜节律的核心调节因子,其共同分配到同一模块强调了它们在维持生物钟功能中的紧密联系。
昼夜节律调控的复杂性:不同模块反映了昼夜节律调控网络的复杂性。节律基因的分布在不同模块中可能揭示了昼夜节律调控的多层次、多信号途径。例如,CRY1和CRY2虽然都是光周期感应和节律维持的关键因子,但被聚类到不同模块(brown和pink),可能反映了它们在不同生物学过程中的独特角色或参与了不同的调节子网络。
潜在的调节机制:WGCNA还可以揭示节律基因之间潜在的调节机制,如转录因子和其靶基因之间的关系,以及它们如何响应环境信号(如光照)。不同模块中的基因可能受到不同转录复合体或信号途径的调控。
研究方向和治疗靶点:这种聚类分析提供了研究昼夜节律调控机制和识别潜在治疗靶点的新方向。例如,如果某个模块的基因表达与疾病状态紧密相关,那么这个模块中的关键基因或调节网络可能成为治疗目标。
总之,WGCNA的这种分析方法通过揭示节律基因在表达模式和功能上的协同性,为理解生物体内部时间感知、节律维持和调控机制提供了宝贵的视角。
如果把一只正常的鸡在白天过后放到黑房间里24小时,它松果体中褪黑素的一天中的分泌大概什么样子的?
在正常条件下,鸡(以及大多数动物)的褪黑素分泌呈现明显的日夜节律变化,夜间高于白天。褪黑素被认为是“黑暗的激素”,因为它主要在夜间或光照较暗的条件下合成和分泌。这种分泌模式由生物钟(位于下丘脑的视交叉上核,SCN)控制,并通过光照条件对松果体进行调节。
如果一只正常的鸡在白天过后被放入完全黑暗的房间24小时,其褪黑素的分泌模式将受到影响。在开始的黑暗阶段(即正常的夜间时段),我们可以预期褪黑素水平上升,因为这与其生物钟和外界已经暗下来的条件相一致。然而,在接下来的24小时中,由于缺乏光照来重置生物钟,褪黑素分泌可能会继续保持在较高水平,而不是在“白天”时段下降,因为松果体不会接收到通常由光照引起的抑制褪黑素合成的信号。
然而,值得注意的是,生物的内部昼夜节律并非立即调整。在持续的黑暗条件下,一些动物的生物钟可能会稍微偏离24小时的自然节律,导致所谓的自由运行节律。因此,鸡的褪黑素分泌模式可能会逐渐发生微妙的改变,但在最初的24小时内,主要特征可能是褪黑素水平整体较高,特别是在预期的“白天”时段。
具体来说,如果一只鸡被放在持续的黑暗中,其松果体中褪黑素的分泌曲线可能会展示如下特点:
- 在正常的夜间时段,褪黑素水平升高,符合生物的内部节律。
- 在接下来的“白天”时段,由于缺乏光照信号,褪黑素水平可能不会像在正常光照条件下那样明显下降,而是保持在较高水平。
- 随着时间的推移,如果持续处于完全黑暗的环境中,鸡的生物钟可能会经历自由运行,导致褪黑素分泌模式与正常的24小时日夜节律略有不同。
这种情况下,观察到的褪黑素分泌模式提供了关于动物如何在没有外界光照周期信号的情况下调整其内部生理过程的洞见。
在正常情况下,鸡等动物的褪黑素分泌具有明显的昼夜节律,夜间分泌量显著高于白天。这是因为褪黑素通常被称为“黑暗激素”,其分泌在光照条件下被抑制,在黑暗中则被激活。如果一只鸡在白天过后被放入一个持续24小时的全黑暗房间,其褪黑素分泌模式可能会经历一段调整期,但大体上可以预测如下:
0小时(刚转入黑暗时):此时可能正值晚上或黄昏,褪黑素分泌开始增加,因为这通常是它们在自然条件下分泌褪黑素的时间。
4小时:在持续的黑暗条件下,褪黑素水平可能会继续上升,达到夜间高水平的趋势。在自然条件下,这个时间点通常是深夜,褪黑素分泌接近或处于峰值。
8小时:褪黑素分泌可能保持在较高水平,因为持续的黑暗环境支持褪黑素的产生。在正常的昼夜节律中,此时褪黑素水平通常是最高的。
12小时:如果黑暗持续,褪黑素分泌可能开始逐渐下降,尽管外界环境依然是黑暗的。这是因为褪黑素的分泌也受到内部生物钟的调控,而生物钟会尝试维持大约24小时的节律。
16小时:在接近“假想”的白天时间,褪黑素水平可能进一步降低,尽管在全黑暗条件下,这个下降可能不如在自然光照条件下明显。
20小时:褪黑素分泌可能继续处于较低水平,但随着内部生物钟的影响,可能会开始缓慢上升,预备进入下一个夜间周期。
需要注意的是,虽然全黑暗环境会影响褪黑素的分泌模式,但内部生物钟仍会尝试按照大约24小时的节律来调节褪黑素分泌,导致即使在持续的黑暗中,褪黑素的分泌也可能出现周期性的波动。然而,缺乏光照的直接调节可能会导致这些波动的幅度和准确的时间点与自然环境下有所不同。
当一只正常的鸡在白天过后被放入持续黑暗的房间中24小时时,其松果体中基因的转录活动将受到影响,尤其是那些直接参与褪黑素合成和调控其日夜节律的基因。由于松果体是褪黑素的主要生产地,其活动受到光照周期的强烈影响。在连续的黑暗环境中,以下基因在松果体中的活跃情况可能表现出特定的模式:
**AANAT (Serotonin N-acetyltransferase)**:这是褪黑素合成途径中的关键酶,它的活性在夜间通常会增加,以促进褪黑素的产生。在持续的黑暗条件下,AANAT的表达和活性可能保持在较高水平,反映出褪黑素生产的增加。
**HIOMT (Hydroxyindole O-methyltransferase)**:负责褪黑素合成最后步骤的酶,可能也会见证类似AANAT的表达模式,随着持续的黑暗条件促使褪黑素合成增加,其活性可能保持较高。
**TPH (Tryptophan Hydroxylase)**:作为合成血清素(褪黑素的前体)的第一步酶,TPH在持续黑暗中可能也会显示出较高的表达,尽管它的调节可能不如AANAT那样直接受到光周期的影响。
除了直接参与褪黑素合成的基因外,还有几个与其日夜节律调节相关的基因可能在这种条件下活跃:
**Per1 and Per2 (Period genes)**:这些基因参与控制生物的内部时钟和昼夜节律。在持续黑暗中,它们的表达模式可能发生变化,反映出生物钟对缺乏光照信号的适应。
**Cry1 and Cry2 (Cryptochrome genes)**:这些基因编码的蛋白质参与感应光周期,调节内部时钟。在持续黑暗的条件下,它们的活性可能会受到影响,从而影响整体的昼夜节律。
Clock and Bmal1:这两个基因编码的蛋白质在维持昼夜节律中互为搭档,共同影响其他节律基因的表达。它们的活性可能会反映出对持续黑暗环境的生理适应。
在持续的黑暗条件下,松果体中这些基因的表达和活动可能反映出鸡体内试图维持其内部时钟和生理节律的努力,尽管缺乏光照信号。特别是那些直接参与褪黑素合成的酶的基因可能会保持较高水平的表达,以响应持续的黑暗条件。然而,具体的表达模式可能会随着时间和个体之间的差异而有所不同,受到内部生物钟和其他调节因素的影响。
cytoscape里的cytohubba功能是什么,用MCC方法,筛选WGCNA每个模块里的top30基因?
Cytoscape是一款流行的生物信息学软件,用于可视化分子交互网络和生物途径。它通过插件扩展功能,其中CytoHubba是一个用于探索网络中节点中心性的插件。CytoHubba提供了多种算法来帮助识别网络中的关键节点(如蛋白质、基因等),这些节点可能在生物过程中扮演重要角色。
MCC(Maximal Clique Centrality)是CytoHubba提供的一种算法,用于通过识别最大团中心性来发现网络中的重要节点。MCC算法考虑了节点的局部网络结构,能够有效地识别那些与许多其他高连接节点相连的节点,这些节点被认为在网络中具有较高的中心性和重要性。
在使用WGCNA(加权基因共表达网络分析)方法分析基因表达数据时,首先会根据基因表达模式将基因聚类成多个模块,每个模块包含功能上可能相似或相关的基因。接下来,如果想要进一步探索每个模块中最重要的基因,可以使用CytoHubba的MCC方法来筛选出每个模块中的top 30个基因。这些基因可能在相应的生物过程或疾病中扮演关键角色。
具体步骤如下:
- 在Cytoscape中构建基因共表达网络。
- 将WGCNA分析得到的各个模块导入Cytoscape。
- 使用CytoHubba插件,并选择MCC算法来分析每个模块。
- 筛选出每个模块中MCC值最高的前30个基因作为关键基因。
这样,就可以针对WGCNA识别出的每个模块内部,进一步识别和分析在网络中具有高中心性的关键基因,为后续的生物学验证和研究提供重要线索。
User
那么我WGCNA后,先对感兴趣的模块,进行基因的富集分析,然后再用cytoscape里的cytohubba筛选模块中的枢纽基因,然后再对模块和性状相关的基因,将GS值和KME 值均设置大于0.8,对非常显著的基因表达与特定性状,进行关键枢纽基因的挖掘,这样可以吗?论文能说通吗/
你提出的分析流程是合理的,并且能够在科研论文中得到很好的解释和支持。这种方法结合了多种生物信息学技术和统计分析,以识别与特定性状显著相关的关键枢纽基因。以下是该流程的简要概述和其科学合理性:
WGCNA分析: 通过WGCNA(加权基因共表达网络分析)识别基因表达数据中的模块,这些模块基于基因表达模式的相似性聚集在一起,可能反映了共同的生物学功能或病理状态。
富集分析: 对感兴趣的模块进行基因富集分析(如GO富集分析或KEGG路径分析),以理解这些模块在生物学过程、分子功能或疾病途径中的作用。这一步骤有助于揭示模块的潜在生物学意义。
使用CytoHubba筛选枢纽基因: 通过Cytoscape的CytoHubba插件选择特定模块,使用MCC等方法筛选出模块中的关键枢纽基因。枢纽基因在网络中的位置使它们在调控生物过程中可能起到重要作用。
结合GS值和KME值进行筛选: GS(基因显著性)值和KME(模块成员资格)值都设置大于0.8,用于进一步筛选与性状非常显著相关且在其所在模块中中心性高的基因。这种方法有助于识别在统计上与特定性状相关且在网络中具有重要作用的基因。
关键枢纽基因的挖掘: 最后,对这些经过严格筛选的基因进行详细分析,以挖掘它们在特定性状或疾病中的作用。这可能包括其表达水平的变化、它们参与的生物学途径以及它们如何与其他基因相互作用等。
这个分析流程综合了表达数据的模块化分析、生物信息学富集分析和网络中心性分析,是一个多角度、多层次探究基因功能和基因-性状关系的强有力方法。在撰写论文时,需要清晰地描述每一步的方法学细节和理由,以及如何从每一步得到的结果导出下一步的分析。此外,对于筛选标准(如GS值和KME值的阈值设置)的选择也需要合理的解释和支持,以确保研究结果的可靠性和有效性。
写论文的思路
全基因组重测序:
我找到了一些关于鸡全基因组重测序数据的研究。这些数据主要来自于中国的本地鸡种,涵盖了大量的样本和基因多样性分析。
一项研究对126个鸡样本进行了全基因组重测序,生成了覆盖7.0×至48.9×的清洁数据,映射率超过97%,发现了大量的单核苷酸多态性(SNPs)和插入缺失变异(InDels)【5†source】。此外,这项研究还探讨了22个鸡种群的基因多样性、杂合性和连锁不平衡,以及不同种群之间的基因分化和种群遗传结构,揭示了高水平的混合交叉特征【5†source】。
Genome diversity of Chinese indigenous chicken and the selective signatures in Chinese gamecock chicken
https://www.nature.com/articles/s41598-020-71421-z
https://www.nature.com/articles/s41598-020-71421-z#MOESM3
是个好文章
在这项研究中,我们对126只鸡进行了采样和全基因组重新测序,其中包括4个中国常见的典型商品鸡品种、2个中国斗鸡品种、另外11个中国国家标准土鸡品种、1个红原鸡种群来自中国广西省的三只斗鸡(注S1;表S1)。结合之前发表的31只鸡(藏鸡、北斗鸡品种、云南村鸡、红原鸡)的基因组测序数据4,这些数据共同让我们对基因组变异/基因有了更深入、更全面的了解。揭示斗鸡鸡的特征,并评估冷适应留下的潜在基因组足迹。
另一项研究关注了贵州省八种表型不同的本地鸡品种,研究了它们的遗传多样性、结构和由人工选择形成的遗传变异模式。该研究通过对不同鸡种进行多项遗传多样性参数的分析,发现了商业鸡种和本地及野生鸡种之间的遗传差异,并通过主成分分析、邻接结合树和Admixture分析进一步探讨了不同鸡种之间的遗传结构【6†source】。
Whole-genome sequencing revealed genetic diversity, structure and patterns of selection in Guizhou indigenous chickens
https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-023-09621-w
本研究对贵州省8个家养品种和2个商业品种的300只鸡进行了单独测序,相当于每只平均12.89×覆盖度。总共获得了4201.19 Gb的高质量基因组数据
这些研究为理解鸡的遗传多样性、种群结构以及人工选择对鸡种群遗传变异的影响提供了宝贵的数据和见解。如果你想获取这些数据用于研究,我建议联系这些研究的作者或相关机构,询问数据获取的可能性。一般而言,科研数据的共享需要遵循相关的数据使用协议和版权政策。
巴布考克B380鸡,你知道吗?
巴布考克B380是一种肉用鸡品种,被广泛用于商业养鸡业,尤其是在生产高效率的肉鸡方面。由于具体的性能指标、遗传信息或养殖建议等信息可能随时间和来源而有所不同,详细的资料建议参考专业的农业或畜牧业出版物和研究报告。
巴布考克B380是一种专为自由放养环境而培育的棕色羽毛鸡种,能产生超过300枚中棕色鸡蛋。这种鸡种已经经过选择,以便能够适应室外条件,比起蛋鸡的其他品种在开始产蛋时体重稍重,这使得它们更能适应外部环境【24†source】。
https://www.chickens.allotment-garden.org/keeping-chickens/hybrid-or-pure-breed-chickens/hybrid-chicken-breeds/
巴布考克品种,无论是白色还是棕色,都适应于多种气候和饲养系统。这些鸡因其出色的生存能力和产蛋率而闻名于世,能够为全球的蛋品生产者带来更多的可销售鸡蛋,提升经济效益【26†source】
https://www.babcock-poultry.com/en/
https://www.babcock-poultry.com/en/product/white/
官方网站
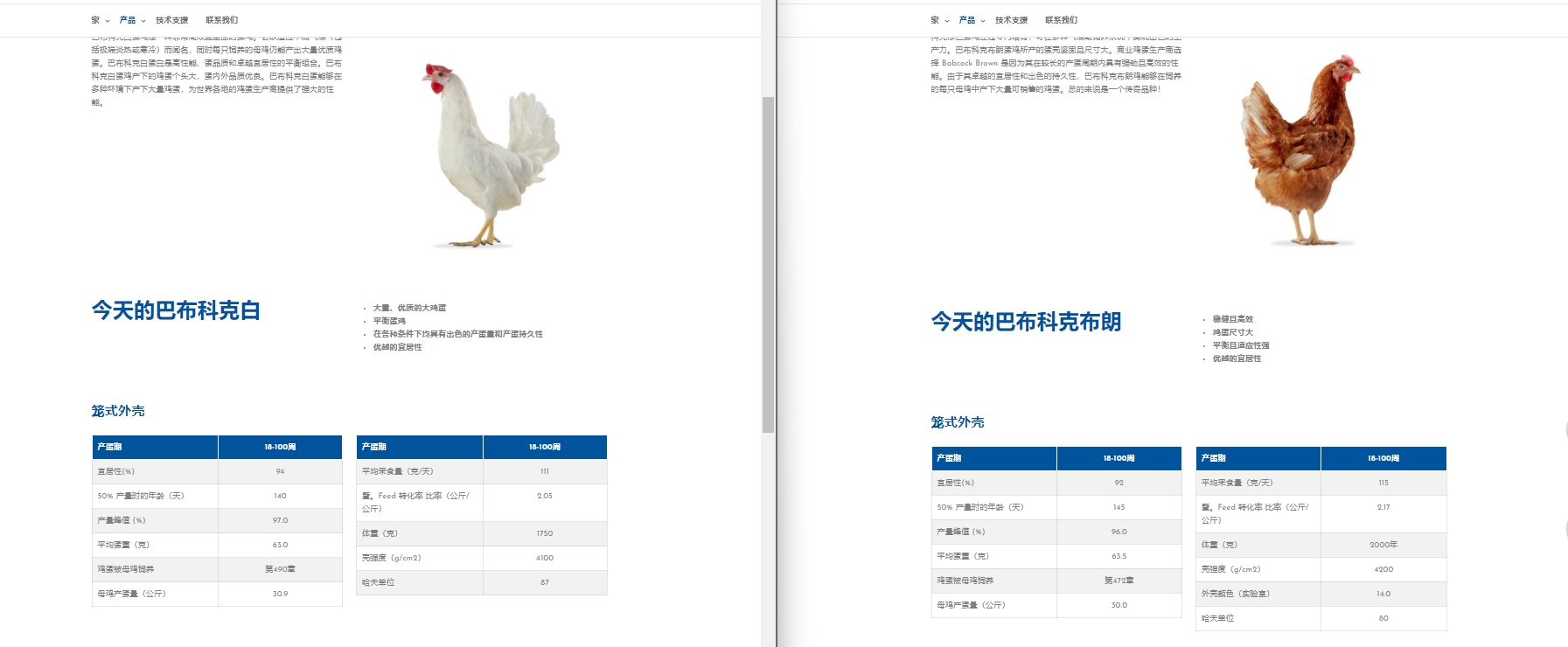
。河北天石禽业有限公司位于河北省,是Hendrix Genetics旗下世界级品牌的中国分销商之一,主要分销巴布考克品种。该公司拥有6000只巴布考克B380的祖代种鸡,致力于向中国市场提供既能满足鸡肉需求又能提供鸡蛋的双用途鸡种【27†source】。
河北天石禽业有限公司位于河北省,是Hendrix Genetics旗下世界级品牌的中国分销商之一,主要分销巴布考克品种。这些品种主要用于通过与Anak或其他有色肉鸡公鸡杂交来生产本地肉鸡,同时也作为高产蛋鸡使用。
公司拥有6000只巴布考克B380的祖代种鸡,致力于为中国市场提供既能满足鸡肉需求又能产蛋的双用途鸡种。
我们与HGL中国团队紧密合作,努力不断改进我们的养殖技术、饲料配方、禽场管理和孵化结果。我们致力于为我们的客户提供高质量的一日龄雏鸡和专业的客户服务。
如果你对巴布考克B380鸡种感兴趣,并希望了解更多关于养殖技术、饲料配方、禽场管理和孵化结果的改进方法,可以联系河北天石禽业有限公司获得高质量的一日龄雏鸡和专业的客户服务【27†source】。此外,还可以关注Hendrix Genetics的新闻门户,了解最新的蛋鸡行业动态和发展【26†source】。
鸡与鸡的基因名
Babcock Brown380
Babcock Brown 380 genome
在中国,一些常见的鸡品种及其中英文名称包括:
- 油鸡 (Silkie):以其独特的丝绸般羽毛和黑色皮肤、骨骼而著名。
- 乌鸡 (Black-boned chicken):也称作黑骨鸡,因其黑色的骨骼和肉质而知名,常用于药膳。
- 北京油鸡 (Pekin Duck, 错误命名,实际应为北京鸭):这实际上是鸭子品种,但经常与鸡混淆,在此纠正。
- 土鸡 (Local breed chicken):指本地养殖的鸡,通常指任何非商业养殖的本地品种。
- 蛋鸡 (Layer chicken):专门用于产蛋的鸡品种,如海兰褐。
- 肉鸡 (Broiler chicken):专门用于肉用的鸡品种,如白羽肉鸡。
中国有许多本土鸡品种,每种都有其独特的特征和用途,从肉质到产蛋率,以及适应不同气候和地理条件的能力。
海兰褐鸡的英文名字是Hy-Line Brown。这是一种常见的蛋鸡品种,以其高产蛋率和良好的适应性著称,广泛用于商业蛋鸡生产中。
论文:节律基因正选择
进行种内正选择(positive selection)的分析主要旨在识别自然选择作用下保留下来的基因或基因区段,这通常意味着这些基因或区段在物种的适应性进化中扮演了重要角色。要进行这样的分析,需要准备以下类型的数据和资源:
序列数据:
- 多个等位基因序列:对于种内分析,你需要收集同一物种内不同个体的同一基因的序列。这些序列代表了基因的不同等位基因,可以用来探测正选择的信号。
- 高质量的参考基因组:有时,你还需要该物种的参考基因组来辅助分析,特别是在定位基因或基因组区段以及进行注释时。
比对和变异检测工具:
- 多重序列比对工具:如MAFFT、MUSCLE或Clustal Omega,用于比对等位基因序列。
- 变异检测工具:根据比对结果,可以使用特定的工具来检测序列间的变异,包括单核苷酸多态性(SNP)和插入/缺失(indels)。
正选择分析软件:
- PAML(Phylogenetic Analysis by Maximum Likelihood):特别是它的Codeml工具,可以用来分析氨基酸替换的模式,识别受正选择作用的密码子。
- HYPHY:包括BUSTED、MEME和FEL等方法,用于检测正选择信号。
- SLiM或SweeD:用于扫描基因组以寻找选择信号的工具。
统计和图形软件:
- 用于进化率计算、统计检验和可视化分析结果的软件,如R语言及其包、Python或专门的进化分析软件。
额外的比较组数据(可选):
- 有时,与其他物种的比较数据也可以帮助识别正选择,特别是在进行种间比较的情况下。这需要收集其他物种中相应基因的序列数据。
实验室条件下的功能验证数据(可选):
- 对于一些研究,特别是那些寻找特定功能变异的,实验室验证(如表型分析、基因表达实验)的数据也是重要的补充,可以帮助解释正选择的生物学意义。
在进行种内正选择分析之前,收集和准备上述数据是非常重要的。这需要综合运用生物信息学、统计学和分子生物学的知识。此外,对分析结果的解释需要谨慎,特别是考虑到可能的混淆因素,如群体结构和基因流,这些因素可能会影响选择信号的检测。
又没有类似的文章,帮我搜索一下?
I found some research that, while not directly examining positive selection on circadian rhythm genes in chickens specifically, provides insight into the broader context of circadian rhythms in poultry and how they might be influenced by light and potentially selected for in breeding practices.
One study delves into the effects of light on poultry circadian rhythm regulation, highlighting the importance of core clock genes (cClock, cBmal1, cBmal2, cCry1, cCry2, cPer2, and cPer3) in this process. These genes are involved in the expression and regulation of melatonin, a hormone crucial for circadian rhythms. The study suggests that light conditions can affect the expression of these genes, thus influencing melatonin levels and potentially affecting traits of interest in poultry breeding, such as feed conversion ratio and egg weight【11†source】.
Another piece of research I found discusses various methods for detecting positive selection in genomes, including those of circadian rhythm genes. It explains how the site frequency spectrum (SFS) and divergence between species can be used to infer positive selection, highlighting the use of outgroup species for polarity and the calculation of the proportion of amino acid substitutions driven by positive selection (α). This could be applicable in studying positive selection in circadian rhythm genes in chickens by comparing their genomic sequences to those of closely related species【12†source】.
Furthermore, research on circadian clock genes’ roles in human diseases, such as pan-renal cell carcinoma, shows how alterations in these genes can impact cell cycle, immune response, and apoptosis. This research underscores the significant biological roles that circadian rhythm genes play, beyond just controlling sleep-wake cycles, which could be relevant to their selection in agriculture for traits like disease resistance or growth rates【13†source】.
While these studies do not directly answer whether circadian rhythm genes in chickens are under positive selection for egg-laying or meat production, they underline the complex roles these genes play in physiological processes. This complexity suggests that there could indeed be selective pressures on circadian rhythm genes to optimize production traits in poultry. For more direct evidence, however, research specifically targeting genetic selection in egg-laying and meat-producing chickens would be necessary.
https://www.mdpi.com/2076-3417/13/5/3157
https://bmcbiol.biomedcentral.com/articles/10.1186/s12915-017-0434-y
https://www.frontiersin.org/articles/10.3389/fmolb.2021.747629/full
如果我想做这个课题?怎么做?需要哪些数据
要研究蛋鸡和肉鸡的节律基因是否受到正选择,你需要进行一系列的步骤,涉及数据收集、分析和解释。以下是详细的研究计划和所需数据:
1. 定义研究目标和假设
- 明确你想要探究的问题,比如是否存在特定的节律基因变异在蛋鸡或肉鸡中受到正选择,以及这些变异如何影响生产性能(如产蛋率、生长速度等)。
2. 数据收集
- 遗传数据:收集蛋鸡和肉鸡的基因组数据,特别是关注那些已知或预测与节律调控相关的基因区段。数据可以从公共数据库如NCBI或Ensembl获取。
- 群体数据:对于不同地理位置、不同育种线的蛋鸡和肉鸡群体,收集基因型数据。
- 表型数据:收集相关的生产性能数据,如产蛋率、生长速度、肉质等,这些数据可能需要通过文献回顾或与养殖场合作获得。
3. 数据分析
- 多重序列比对:使用生物信息学工具(如MAFFT、Clustal Omega)比对选定基因的序列,以识别遗传变异。
- 群体遗传学分析:应用软件(如Arlequin、PopGenome)进行群体遗传结构和多样性分析,探索不同群体间的遗传差异。
- 正选择信号检测:使用PAML的Codeml、HYPHY等软件检测正选择的信号。这包括计算非同义替换与同义替换比率(dN/dS)、构建种群遗传结构和历史模型等。
- 关联分析:将遗传变异与表型数据关联,使用GWAS(全基因组关联研究)方法识别与生产性状相关的遗传标记。
4. 结果解释和验证
- 生物学意义:解释找到的正选择信号和遗传标记如何与节律调控和生产性能相关。
- 功能验证:对于关键的遗传变异,可能需要进一步的实验室研究(如CRISPR/Cas9基因编辑、表达分析)来验证它们的功能。
5. 发表结果
- 编写研究报告或论文,发表在相关的科学期刊上。
所需资源
- 访问基因组数据库和生物信息学工具。
- 实验室设备和材料(如果进行功能验证实验)。
- 与养殖场和其他研究机构的合作,以获取表型数据和样本。
进行这样的研究是一项复杂的任务,需要跨学科的知识,包括遗传学、生物信息学、动物科学和统计学。开始之前,最好与这些领域的专家建立合作,以确保研究的成功进行。
我想做鸡的种内正选择,研究clock这个基因,然后我在NCBI中找到这个基因在参考基因组上的位置,然后从自己的数据库得到自己肉鸡的这个基因的vcf文件,然后经过一系列处理,放到losition软件中,上面的红色部分的图是FST/HE,这个是正选择吗
你上传的LOSITAN Selection Workbench的图显示了Fst(固定指数)和He(预期杂合度)之间的关系。这种图通常用来检测群体内的选择信号。如果某个位点的Fst值高于中性预期的水平,且考虑到它们的杂合度(He),那么它可能表明这个位点正在经历正选择。然而,确实判定“clock”基因是否受到正选择需要考虑更多因素,如群体结构、遗传漂变等。通常需要多种方法的证据支持,并考虑特定变异的适应性意义。
鸡chicken,Chicken (bGalGal1.mat.broiler.GRCg7b)
https://useast.ensembl.org/Gallus_gallus/Info/Index
这段信息提到了一种肉鸡基因组的组装版本“bGalGal1.mat.broiler.GRCg7b”,其访问编号为GCA_016699485.1。这是肉鸡基因组的一种参考组装,旨在为研究者提供详细的基因组信息,有助于肉鸡相关的基因组学研究和遗传改良。
鸡chicken breeds
这些信息描述了两种鸡的基因组数据:
https://useast.ensembl.org/Gallus_gallus/Info/Breeds
白色莱霍恩种母鸡(Gallus gallus)的基因组数据,使用的是bGalGal1.pat.whiteleghornlayer.GRCg7w组装版本,访问编号为GCA_016700215.2。白色莱霍恩鸡是一种常见的蛋鸡品种,以高产蛋和较快的生长速度而知名。
红色丛林鸡(Gallus gallus)的基因组数据,使用的是GRCg6a组装版本,访问编号为GCA_000002315.5。红色丛林鸡被认为是所有家鸡的祖先,具有重要的研究价值,尤其在研究家鸡的驯化和进化方面。
https://useast.ensembl.org/Gallus_gallus/Gene/Summary?g=ENSGALG00010015364;r=4:64586757-64632362
https://www.ncbi.nlm.nih.gov/search/all/?term=gallus%20gallus%20clock
师姐那个着实看不懂
正选择位点是基因的snp位点吗?
正选择位点可以是单核苷酸多态性(SNP)位点,但不限于此。正选择位点指的是在进化过程中因为对生物有利(提高生存或繁殖成功率)而被自然选择积极选择的遗传位点。这些位点可以是基因内的,也可以是基因间或基因调控区域内的,包括但不限于SNP。
单核苷酸多态性(SNP)
- SNP:是最常见的遗传变异形式,指基因组中单个核苷酸的变化。例如,一个位点上的核苷酸在大多数个体中是A,而在少数个体中变为T,这就形成了一个SNP。
- SNPs可以影响基因的表达和功能,包括蛋白质的结构和功能改变,也可能影响基因的调控区域,进而影响基因表达的时机和水平。
正选择位点的其他形式
除了SNP,正选择位点还可以是其他形式的遗传变异,如:
- 插入/缺失(Indels):基因组序列中小段DNA的插入或删除。
- 拷贝数变异(CNVs):基因组中一段区域的DNA复制次数的变化。
- 结构变异:更大范围的基因组重排,包括倒置、转位等。
确定正选择位点
确定某个位点是否为正选择位点,需要通过比较不同群体或物种的遗传数据,并使用统计模型分析。如果某个位点或区域在统计分析中显示出了高于随机预期的进化速率(如非同义SNP的比例显著高于同义SNP),则可能被认为是正选择的标志。
总之,正选择位点不仅包括SNP,还可以是任何类型的遗传变异,关键在于这些变异如何影响个体的适应性和进化。
要研究蛋鸡和肉鸡的节律基因clock是否受到正选择?我的是蛋鸡的vcf全基因组重测序文件,里面的参考是肉鸡的,然后我找到clock的位置,然后找clock内部的snp是否受到正选择吗?
要研究蛋鸡和肉鸡的节律基因clock是否受到正选择,你的思路是正确的。使用蛋鸡的全基因组重测序数据(VCF文件)并以肉鸡的基因组作为参考是一种常见的做法,因为这可以帮助你发现相对于参考基因组的遗传变异。下面是一个研究流程概述:
1. 确定clock基因的位置
- 首先,需要在参考基因组中准确定位clock基因的位置。这可以通过查阅基因组注释文件来完成。
2. 提取clock基因区域的SNP
- 使用你的VCF文件,提取位于clock基因区域内的所有SNP。这一步可以使用生物信息学工具,如
bcftools或GATK的特定功能来实现。
3. 分析SNP的正选择信号
- 接下来,分析这些SNP是否显示出正选择的信号。这通常涉及到计算非同义(导致氨基酸变化)与同义(不导致氨基酸变化)替换的比率(dN/dS比率)。dN/dS比率大于1可能表明正选择。
- 还可以使用其他方法和软件(如PAML, HyPhy)来进行更复杂的分支模型、分支-位点模型或位点模型分析,这些方法可以帮助确定特定进化路径或位点上的正选择信号。
4. 统计学分析
- 对于发现的可能受到正选择的SNP,进行统计学分析,比较蛋鸡和肉鸡之间的频率差异,以进一步支持你的发现。
5. 生物学验证
- 确定了可能受到正选择的SNP后,可能还需要进行进一步的生物学实验,如基因表达分析,来验证这些SNP对clock基因功能的影响,以及它们如何影响蛋鸡和肉鸡的生理和行为差异。
注意点:
- 在进行这类分析时,应确保使用适当的对照组和统计方法,以避免假阳性发现。
- 对于正选择的分析,需要有一定的生物信息学和统计学背景,确保分析方法的正确性和结果的可靠性。
这是一个复杂的分析项目,但它可以揭示clock基因在蛋鸡和肉鸡进化中的作用,以及它如何通过正选择过程对鸡的生产性状产生影响。、
第三步骤
分析SNP是否显示出正选择的信号是一个多步骤的过程,特别是当你已经有了特定基因(如clock基因)的VCF文件。下面是具体步骤:
步骤 1: 准备序列数据
提取SNP序列:首先,你需要从clock基因的VCF文件中提取感兴趣的SNP序列。这通常涉及将VCF格式转换为适用于后续分析的格式,例如FASTA或其他分析软件需要的特定格式。
生成编码序列:对于非同义和同义替换的分析,你需要关注编码序列(即那些编码蛋白质的DNA序列)。这意味着可能需要从基因组数据中提取相应的编码序列,并将其与你的SNP数据对齐。
步骤 2: 计算dN/dS比率
使用如PAML(Phylogenetic Analysis by Maximum Likelihood)这样的软件包,特别是其中的
Codeml工具,可以计算dN/dS比率。这需要你构建一个进化树,代表你分析的物种或个体之间的进化关系。对于单一基因(如clock基因),你可能会使用一个简化的进化树,或者如果你只关注蛋鸡和肉鸡,甚至可以是一个非常基础的分支结构。
步骤 3: 使用分支模型、分支-位点模型或位点模型
分支模型:如果你怀疑正选择发生在特定的进化分支上(例如,只在蛋鸡或肉鸡中),可以使用分支模型来测试这个假设。
分支-位点模型:如果你认为某些位点在特定分支上受到正选择,而其他位点则不受或受到不同程度的选择,可以使用分支-位点模型。
位点模型:如果你的兴趣在于哪些具体的位点在整个研究群体中可能受到正选择,而不局限于特定分支,可以使用位点模型。
步骤 4: 分析和解释结果
分析上述模型的输出,特别关注dN/dS比率大于1的位点,这表示这些位点可能受到正选择的影响。
注意,这些分析通常需要一定的进化生物学和统计知识,以正确解释结果。
注意事项
- 确保使用最新版本的软件和数据库,以便获得最准确的分析结果。
- 考虑进行多种分析,以从不同角度验证你的发现。
- 实验设计和数据分析中的任何假设都应该清楚地记录和报告。
进行这类分析是一个详细且复杂的过程,可能需要一定的学习曲线,特别是对于那些刚开始使用生物信息学工具和软件的研究人员。不过,这是一个非常有价值的研究方向,能够提供关于基因如何影响生物进化和适应性的深刻见解。
鸡Wnt基因家族生物信息学分析及表达
https://www.uniprot.org/uniprotkb/P49339/entry
MHC_UniPort
https://www.ebi.ac.uk/interpro/entry/pfam/PF00110/



