阅读学术论文不仅能够开阔你的视野,让你深入了解领域前沿的研究和最新发现,而且它还能激发你的好奇心和批判性思维,使你能够系统地构建知识框架,更好地理解复杂的科学问题。这种经验可以让你逐步形成解决实际问题的能力,从而在你的学术或职业生涯中取得突破。
如果您想查找关于Gallus gallus(鸡)的MHC类I和类II分子的研究,以下是一些可靠的学术资源和数据库,您可以在其中搜索相关论文:
PubMed:
- 访问 PubMed 网站。
- 使用关键词如 “Gallus gallus MHC”, “chicken major histocompatibility complex”, 或 “avian immunogenetics” 进行搜索。
Google Scholar:
- 访问 Google Scholar。
- 输入搜索词,例如 “Gallus gallus MHC diversity”, “chicken MHC disease resistance”, 或其他相关的词。
ScienceDirect:
- 访问 ScienceDirect 网站。
- 搜索有关鸡MHC的论文,例如使用关键词“MHC and poultry”或“immune genes in chickens”。
Springer:
- 访问 SpringerLink。
- 在搜索框中输入相关的搜索词,如“Gallus gallus MHC”.
在这些资源中,您可以找到从基础研究到应用研究的广泛文献,包括关于MHC的多态性、免疫反应与疾病抵抗关系的研究。此外,如果您所在的机构或学校有订阅特定的科学期刊或数据库,那么通过这些机构的图书馆也可以访问到全文论文。
1,河北地方鸡羽色候选基因OX10、SLC45A2 和Tyr变异研究
河北 地方鸡 羽色 候选基因 SOX10、SLC45A2 和Tyr 变异研究
关键字,鸡;羽色;SOX10;SLC45A2;Tyr
摘要:
为了解河北地方鸡种的羽色遗传资源特征,检测羽色基因位点在不同品种鸡的分布情况,
本试验通过***PCR 以及 PCR-SSCP ***检测发现,
1,90% 以上的太行鸡和坝上长尾鸡与海兰褐和海兰灰祖代鸡一样,其 SOX10 基因上游有 8.3 kb 序列插入,表现红色野生型羽色特征;
2,而白来航鸡主要为缺失型,具有深棕羽色基因型基础。
3,太行鸡和海兰灰与白来航的 SLC45A2 基因外显子 3 的 A/G 突变位点为 G 等位基因,具有银羽基因遗传基础;
4,而坝上长尾鸡主要为 A 等位基因,具有金羽基因遗传基础。46.0% 的坝上长尾鸡和 16.7% 的太行鸡 Tyr 基因有
7.7 kb 病毒插入,存在白羽遗传基础,而祖代海兰褐、白来航和海兰灰的插入阳性率较低。
综上所述河北省地方品种中存在丰富的羽色基因库,有筛选培育各种羽色品系的遗传基础。
以下是关于河北地方鸡种羽色遗传特征研究结果的表格表示:不用看,乱的
| 品种 | SOX10 基因插入(红色野生型羽色) | SLC45A2 基因外显子 3 等位基因 | TYR 基因插入(白羽基因) |
|---|---|---|---|
| 太行鸡 | 90% | G 等位基因(银羽基因) | 16.7% |
| 坝上长尾鸡 | 90% | A 等位基因(金羽基因) | 46.0% |
| 海兰褐 | 90% | G 等位基因(银羽基因) | 插入阳性率较低 |
| 海兰灰 | 90% | G 等位基因(银羽基因) | 插入阳性率较低 |
| 白来航鸡 | 0%(缺失型) | G 等位基因(银羽基因) | 插入阳性率较低 |
此表格总结了特定遗传标记(SOX10 基因插入、SLC45A2 基因外显子 3 等位基因、TYR 基因插入)对研究中的鸡种羽色的影响,显示了河北省地方鸡种中存在丰富的羽色遗传资源,为培育各种羽色品系提供了遗传基础。
研究方法
这篇论文的研究方法包括从不同品种的鸡中收集血样进行DNA提取,然后利用PCR技术检测SOX10基因中8.3 kb插入/缺失变异,SLC45A2基因外显子3中的A/G突变,以及Tyr基因中的7.7 kb插入/缺失变异。通过这些方法,研究团队探讨了这些品种鸡羽色变异的遗传基础。
前言部分
前言部分讲述了羽毛作为禽类皮肤的附属物,其颜色的分子基础与遗传规律研究对于加强遗传资源保护、标记品种特征,以及发掘推广禽类的分子育种技术的重要性。论文提到,禽类的羽色主要由机体自身黑色素细胞合成的黑色素(包括真黑素和褐黑素)决定,不同颜色特征是由真黑素和褐黑素的比例不同造成的。黑色素的合成过程受多基因调控,各基因间的相互作用形成了不同的羽毛颜色。通过探讨深棕色(DB)基因座、鸡Z染色体上的银色等位基因(S*S)的特性,以及SLC45A2和Tyr基因的作用,论文阐述了羽色遗传变异的分子机制。
特别指出,SOX10基因上游的8.3 kb序列缺失,以及SLC45A2和Tyr基因中的特定变异,对于羽色形成的影响。这些研究对于理解不同鸡种羽色的遗传基础,以及为禽类遗传资源的保护和利用提供了科学依据。
此外,论文还提到了河北省地方鸡种,如太行鸡和坝上长尾鸡的重要性,这些品种不仅具有良好的肉蛋品质和适应性,还具有丰富的遗传多样性,为世界家禽育种资源库提供了新的素材。然而,关于这些地方鸡种羽色控制基因多态性的分布情况尚未明确,因此本研究旨在通过检测特定基因的变异,为河北地方鸡的羽色选育及遗传资源的保护奠定基础。
论文的结构如下:
摘要
前言
- 材料与方法
- 1.1 试验材料
采集华裕家禽育种有限公司的 海兰褐祖代 A、B、C、D 四系(A 和 B 系为洛岛红,呈金羽表型;C 和 D 系为洛岛白,呈银羽表型)共 120 只
大午集团海兰灰祖代 A、B、C、D 四系共 94 只,
- 1.1 试验材料
白来航 17 只;
河北张家口京星园生态农业有限公司坝上长尾鸡 24 只;河北石家庄赞皇天然公司太行鸡 23只的血液样品用于 DNA 提取
1.2 试验方法
1.2.1 DNA提取
用苯酚 - 氯仿粗提法提取待测定鸡血液的基因组 DNA,浓度在 200 ~ 400 ng/μL,均稀释至 50 ng/μL,-20 ℃储存备用1.2.2 DB基因座SOX10基因8.3 kb插入/缺失突变检测
DB 基因座 SOX10 基因 8.3 kb 插入 / 缺失突变检测 参考文献[5]设计 3 条引物进行 PCR 反应明确 DB 基因座分型,引物 SOX10-1F 和 SOX10-
1R 均位于 SOX10 基因外显子 1 内,SOX10-6R 位于8.3 kb 缺失区,引物信息见表 1。如果 SOX10_1F 和
SOX10_1R 扩增有 611 bp 目的条带,而 SOX10_1F和 SOX10_6R 无扩增产物,为野生型纯合子(DBN/DBN), 表 现 黑 色 胸 部 的 野 生 型 羽 色 特 征; 若
SOX10_1F 和 SOX10_6R 有 1 257 bp 扩增产物,而SOX10_1F 和 SOX10_1R 无扩增产物,为 DB 纯合子(DBDB/DBDB), 表 现 深 棕 色 表 型; 若 61
和 1 257 bp 2 个目的片段都有,则是杂合子(DBDB/DBN),表现深棕色表型
深棕色(DB)基因座最初定位在横斑 Fayoumi
鸡的常染色体,该基因座有 2 个等位基因:野生型
黑胸(DBN)和深棕型(DBDB)。DB 表型鸡具
有似哥伦比亚样的真黑素限制表达的黑尾红羽毛特
征,公鸡胸部为红褐色,母鸡呈现檀橙色或亮橙色。
Gunnarsson 等研究发现鸡的 DB 表型是 SOX10 基因
转录起始位点上游14 kb处8.3 kb序列缺失导致的[5]
,
缺失序列可能发挥了增强子作用,因此 SOX10 的转
录表达降低,进而影响下游黑色素合成重要基因如
Tyr 的转录,造成真黑素合成的减少,而增强了褐黑
素的表达,使鸡表现 DB 深棕羽色特征[6]
想象一下,有一个神秘的森林,里面住着两种非常特殊的鸟——太阳鸟和月亮鸟。太阳鸟拥有耀眼的红褐色羽毛,而月亮鸟则有着深邃的黑色羽毛。传说,森林中隐藏着一座古老的魔法之泉,能够决定所有鸟儿羽毛的颜色。
这座魔法之泉就像鸟类羽色遗传学中的SOX10基因,它掌管着鸟儿羽毛颜色的秘密。在这个故事中,SOX10基因就像是控制魔法之泉的钥匙,而这个钥匙有一个特殊的部分(8.3 kb序列)能够影响魔法的强度。
有一天,月亮鸟的一个祖先在探索森林时,不小心失去了这个钥匙的一个部分,这就像是SOX10基因发生了8.3 kb的缺失。因为这个部分丢失了,魔法之泉的魔法变弱了,不能像以前那样制造出纯黑的羽毛,于是月亮鸟的羽毛变成了深棕色。
科学家们(就像这个故事中的魔法师)通过一种特殊的咒语(PCR技术)来寻找失去的钥匙部分。他们设计了三根魔杖(三条引物)来确定是不是每只鸟都拥有完整的钥匙。如果只使用两根魔杖(SOX10-1F和SOX10-1R)可以召唤出一个小光球(611 bp的DNA片段),这意味着鸟儿是纯粹的月亮鸟,拥有黑色的羽毛。如果另一根魔杖(SOX10-6R)和其中一根魔杖一起使用,却什么都召唤不出来,那就说明钥匙的一个部分丢失了,鸟儿的羽毛会是深棕色的。
通过这个故事,我们可以理解,SOX10基因的8.3 kb缺失如何影响鸟儿(在现实中是鸡)的羽毛颜色,以及科学家如何通过PCR技术来检测这种遗传变异。就像魔法师揭开了森林中羽毛颜色变化的秘密,科学家们也揭示了鸡羽色遗传的奥秘。
这篇论文中讨论的DB基因座和SOX10基因8.3 kb插入/缺失突变检测的逻辑是这样的:
基因座和等位基因: 深棕色(DB)基因座最初定位于横斑Fayoumi鸡的常染色体。这个基因座有两个等位基因:野生型黑胸(DBN)和深棕型(DBDB)。
DB表型的特征: DB表型的鸡具有类似哥伦比亚样式的真黑素限制表达,公鸡胸部为红褐色,母鸡为檀橙色或亮橙色。
SOX10基因的作用: 研究发现,DB表型是由于SOX10基因转录起始位点上游14 kb处发生的8.3 kb序列缺失导致的。这个缺失可能起到了增强子的作用,降低了SOX10基因的转录表达。
对黑色素合成的影响: 由于SOX10基因表达降低,进而影响到下游黑色素合成的重要基因,如Tyr,造成真黑素的合成减少,同时增强了褐黑素的表达。这种变化导致鸡表现出DB深棕羽色特征。
PCR反应的设计: 为了明确DB基因座的分型,设计了3条引物进行PCR反应。使用的引物包括SOX10-1F和SOX10-1R(都位于SOX10基因外显子1内),以及SOX10-6R(位于8.3 kb缺失区)。
基因座分型的判断方法:
- 如果SOX10_1F和SOX10_1R扩增出611 bp的条带,而SOX10_1F和SOX10_6R没有扩增产物,表示为野生型纯合子(DBN/DBN),表现为黑色胸部的野生型羽色。
- 如果SOX10_1F和SOX10_6R扩增出1257 bp的条带,而SOX10_1F和SOX10_1R没有扩增产物,表示为DB纯合子(DBDB/DBDB),表现为深棕色表型。
- 如果两个目标片段(611 bp和1257 bp)都有扩增产物,则为杂合子(DBDB/DBN),也表现为深棕色表型。
简而言之,这篇论文通过研究SOX10基因中的特定缺失,解释了鸡的羽色变异(特别是DB深棕色表型)的分子机制,并通过特定的PCR方法来确定鸡的DB基因座分型。
- 1.2.3 金银羽基因座SLC45A2基因A>G突变的SSCP检测
- 1.2.4 隐性白基因座Tyr基因7.7 kb插入/缺失突变检测
结果与分析
- 2.1 提取基因组DNA检测
- 2.2 DB基因座SOX10基因8.3 kb插入突变检测
- 2.3 金银羽基因座SLC45A2基因A>G突变检测
- 2.4 隐性白基因座Tyr基因7.7 kb插入突变检测
结论与讨论
这个结构提供了一个清晰的框架,展示了研究的步骤、分析的结果以及最终的讨论和结论。
总结:
通过这篇论文,我们可以学到以下几点关键知识:
- 羽色遗传的分子基础: 论文详细讨论了SOX10、SLC45A2和Tyr三个基因与鸡的羽毛颜色变异之间的关系,揭示了羽色形成的分子机制。特别是SOX10基因上游的8.3 kb序列插入/缺失,SLC45A2基因A>G突变,以及Tyr基因7.7 kb插入/缺失变异对不同鸡种羽色的影响。
羽毛是禽类皮肤的附属物,羽色分子基础与遗传规律的研究,有助于加强遗传资源保护、标记品种特征,发掘推广禽类的分子育种技术[1]。
禽类色素有 2 种来源,但主要是由机体自身黑色素细胞合成产生的黑色素,包括真黑素和褐黑素 2 种[2]。
畜禽表现出的不同颜色特征是由于真黑色素和褐黑色素的比例不同[3]。
黑色素合成过程受多基因调控,各基因间相互作用形成了不同的羽毛颜色[2-4]。
深棕色(DB)基因座最初定位在横斑 Fayoumi
鸡的常染色体,该基因座有 2 个等位基因:野生型
黑胸(DBN)和深棕型(DBDB)。DB 表型鸡具
有似哥伦比亚样的真黑素限制表达的黑尾红羽毛特
征,公鸡胸部为红褐色,母鸡呈现檀橙色或亮橙色。
Gunnarsson 等研究发现鸡的 DB 表型是 SOX10 基因
转录起始位点上游14 kb处8.3 kb序列缺失导致的[5]
,
缺失序列可能发挥了增强子作用,因此 SOX10 的转
录表达降低,进而影响下游黑色素合成重要基因如
Tyr 的转录,造成真黑素合成的减少,而增强了褐黑
素的表达,使鸡表现 DB 深棕羽色特征[6]。
鸡 Z 染色体 S(银色)位点银色等位基因(SS)
特异抑制红色的伪黑素表达,不完全显性于野生型
金色(SN),其表型受其他修饰基因的影响较大,
所以在一定遗传背景下不容易分辨出银色等位基因,
但金银羽色表型是除快慢羽外用于雏鸡性别鉴定的
第 2 种表型[7]。SLC45A2(水溶载体 45 家族第二
成员)是包括 12 个跨膜区域的膜相关转运蛋白,在
黑素细胞的囊泡分类中发挥重要作用[8]。研究发现
SLC45A2 突变影响了后高尔基体水平上酪氨酸酶的
加工和运输,外显子 4 的 C/A 和外显子 3 的 A/G 2
个独立错义突变,分别形成 L347M 和 Y277C 氨基
酸变异。L347M 影响跨膜区 7,该突变与除白来航
鸡外的其他鸡品种的银色表型相关;Y277C 影响了
凸环域,与白来航鸡银羽表型相关[9]。
鸡 C 位点除了呈现完全色素的优势显性野生型
等位基因(CN)外,还有红眼白羽(CRE)、隐
性白羽(CC)和常染色体白化(CA)3 种突变型。
Chang 等发现酪氨酸酶基因(Tyr)内含子 4 的 7.7 kb
内源性白血病病毒序列插入与隐性白表型完全连
锁[10]。7.7 kb 序列的插入导致 Tyr 转录剪切异常,
使编码羧基末端跨膜结构域的外显子 5 转录缺失,
合成细胞质型而非跨膜型的功能缺陷 Tyr 酶,严重
影响黑素体的合成[11],表现隐性白羽性状[10-12]。
河北太行鸡和坝上长尾鸡是河北省境内自然形
成的地方品种,肉蛋品质优良,具有良好的适应性
和遗传多性,可为世界家禽育种资源库提供新的素
材。但目前关于羽色控制基因多态性在河北地方鸡
中的分布尚未明确。本试验检测了 SOX10 的 8.3 kb
缺失突变、SLC45A2 外显子 3 的 A/G 突变和 Tyr 基
因的 7.7 kb 序列插入突变在河北太行鸡、坝上长尾
鸡和引进品种鸡的分布情况,以期为河北地方鸡的
羽色选育以及遗传资源的保护奠定基础
河北地方鸡种的遗传多样性: 论文通过研究河北省地方鸡种,如太行鸡、坝上长尾鸡、海兰褐、海兰灰和白来航鸡的羽色遗传特征,展示了这些地方品种中存在丰富的羽色基因资源,强调了保护和利用这些遗传资源的重要性。
PCR技术在遗传研究中的应用: 论文利用PCR技术和PCR-SSCP方法来检测特定基因的变异,这展示了分子生物学技术在理解遗传特性和促进育种计划中的实用性和有效性。
遗传资源的保护与利用: 论文指出,了解和保护地方鸡种的遗传多样性不仅对维持生物多样性至关重要,也为未来的禽类育种提供了基础,尤其是在培育具有特定生产性能或适应性特征的新品种方面。
科学研究与实际应用的结合: 通过探索鸡的羽色遗传基础,论文不仅增进了我们对禽类遗传学的科学理解,还为实际育种工作提供了理论依据和方法指导,显示了科学研究与实际应用之间的密切联系。
总之,这篇论文提供了关于鸡羽色遗传学的深入分析,为禽类遗传资源的保护、遗传学研究以及育种实践提供了宝贵的知识和技术支持。
知识点
基因座和等位基因: 深棕色(DB)基因座最初定位于横斑Fayoumi鸡的常染色体。这个基因座有两个等位基因:野生型黑胸(DBN)和深棕型(DBDB)。
基因座是什么?等位基因是什么?野生型黑胸(DBN)和深棕型(DBDB),代表着什么?我有点迷。
让我来帮你理清这些概念:
基因座(Gene Locus):基因座是指染色体上一个特定位置,这个位置上的基因决定了生物体的一个特定性状。可以把它想象成一本书中的一个特定章节,这个章节讲述了某个特定故事(性状)。
等位基因(Alleles):等位基因是位于同一基因座上的两个或多个不同版本的基因。它们是由遗传变异造成的,导致同一性状表现出不同的形态。等位基因之间的差异可以是很小的(比如一个单一的DNA基础对变化),也可以是很大的(比如整段DNA序列的插入或缺失)。等位基因就像是书中的不同版本,每个版本都有细微的差别,但讲述的是同一个故事。
野生型(Wild Type, DBN):野生型通常指自然界中最常见的基因形式,或者是在特定实验起始种群中观察到的特定性状的标准形式。野生型黑胸(DBN)就是指在横斑Fayoumi鸡中,常染色体上一个特定基因座的常见形式,这个形式使得鸡的胸部呈现出黑色。
突变型(Mutant, DBDB):突变型是指基因的一个或多个等位基因发生了变异,与野生型相比,导致了性状的改变。深棕型(DBDB)就是指基因座上的等位基因发生了变异,这种变异导致鸡不是展现野生型的黑色胸部,而是展现出深棕色的羽毛。
总的来说,DBN 和 DBDB 代表了横斑Fayoumi鸡中一个特定基因座的两个不同版本(等位基因)。DBN(野生型黑胸)是更普遍、自然界中常见的形式,它导致鸡的胸部呈黑色;而DBDB(深棕型)是一个由于基因变异而产生的形式,导致鸡展现出不同于野生型的深棕色羽毛。
问:有个问题,染色体是X的话,左边一个基因座,右边一个基因座吗?
染色体的形状经常被描述为“X”形,但这实际上是染色体在细胞分裂过程中(特别是在有丝分裂的中期)的形态。在这个阶段,每个染色体都由两条相同的姊妹染色单体组成,它们在中心粒处连接在一起,形成了这个典型的“X”形状。这两条染色单体是在DNA复制过程中形成的,每条都包含一套相同的基因。
对于你的问题,“左边一个基因座,右边一个基因座”这样的描述并不准确。染色体上的基因座指的是特定基因的物理位置,而不是按左右来分布的。基因座是沿着染色体的长度分布的,可以想象成一条线上的不同点,而不是染色体的左右两边。每个基因座都有特定的坐标,这些坐标标识了基因在染色体上的精确位置。
染色体在非分裂期(即大部分时间)的形态并不是“X”形,而是呈现出较为松散的结构,这时基因在染色体内的排列是线性的,而不是成对的“X”形结构。因此,当我们讨论某个基因座位于染色体上的某个位置时,我们是在谈论这个基因在这条线性结构上的位置,而不是在“X”形结构的左右两侧。
等位基因(Alleles):等位基因是位于同一基因座上的两个或多个不同版本的基因。它们是由遗传变异造成的,导致同一性状表现出不同的形态。等位基因之间的差异可以是很小的(比如一个单一的DNA基础对变化),也可以是很大的(比如整段DNA序列的插入或缺失)。等位基因就像是书中的不同版本,每个版本都有细微的差别,但讲述的是同一个故事。
指的是基因座的位置,有很多基因,差别很小?两个等位基因是DBDB或别的,三个等位基因是DBDBDB?等等吗?
让我们更清晰地解释等位基因的概念:
等位基因指的不是基因座位置上有很多不同的基因,而是指在同一个基因座上,因为遗传变异的存在,可能有不同的基因版本。这些不同版本的基因导致同一性状(如眼色、羽色等)表现出不同的形态。等位基因是同一个基因的不同变体,每个变体在DNA序列上可能有微小或显著的差异。
一个基因座上可以有多个等位基因,但是一个个体在某个基因座上通常只有两个等位基因(除非是多倍体生物)。这是因为大多数生物是二倍体,意味着它们有一对染色体,每对染色体中的一个来自母亲,一个来自父亲。因此,一个个体在特定基因座上的等位基因可以是相同的(纯合子,如DBDB或NN),也可以是不同的(杂合子,如DBN)。
例如,当我们说DBDB、DBN这样的组合时,我们是在描述一个个体在某个基因座上的两个等位基因。如果是DBDB,那意味着这个个体从父母那里继承了两个相同的深棕色等位基因;如果是DBN,那意味着这个个体从一个亲本那里继承了深棕色等位基因,从另一个亲本那里继承了黑色等位基因。
因此,“三个等位基因是DBDBDB”这样的表述在大多数情况下并不适用,因为大多数动物和人类等二倍体生物在每个基因座上只有两个等位基因。不过,在一些特殊情况下(如某些植物、昆虫或是多倍体生物),个体可以拥有两个以上的等位基因,但这些情况比较少见。
问: 哦,大概明白了,比如人有22对常染色体,然后每一对染色体,有两条染色体,每个染色体都有基因座,但是这两条染色体基因座中的基因类似,可能版本不同
对,你的理解是正确的。在人类中,有22对常染色体(自体染色体)和1对性染色体,总共23对染色体。每对染色体中的一条来自母亲,另一条来自父亲,这就构成了个体的遗传背景。
每条染色体上都分布着许多基因座,每个基因座是一个特定基因的位置。在每个基因座上,由于遗传的多样性,可能存在不同版本的基因,即等位基因。这些等位基因可能在序列上有细微到显著的差异,这些差异是个体之间表型(如眼睛颜色、头发颜色、身高等)差异的遗传基础。
在每个基因座上,个体携带的两个等位基因可能是相同的(纯合),也可能是不同的(杂合)。这两个等位基因共同决定了某一性状的表现。例如,在血型这一性状中,ABO血型基因座上的等位基因有A、B、O三种,不同的等位基因组合会决定个体的血型是A、B、AB还是O型。
因此,正是这些在染色体上的基因座中的等位基因版本的不同,使得每个个体都具有独特的遗传特征。
哥伦比亚鸡型
哥伦比亚鸡型,特别是指哥伦比亚怀恩多特鸡,以其独特的颜色模式和身体特征而闻名。这些鸡大部分是白色的,具有醒目的黑色标记,围绕着它们的脖子(形成一种领子)、尾羽,有时在翅膀尖上也有。黑色和白色羽毛之间的对比非常明显,视觉上非常吸引人。
哥伦比亚怀恩多特鸡是一种遗产品种,以其温顺的性格、耐寒性以及双重用途能力(既提供鸡蛋又提供肉类)而闻名。它们有一个玫瑰梳型,这在寒冷的天气中非常实用,因为它不太容易受到霜bite的影响。这个品种适用于各种气候,但更喜欢较凉爽的环境,在非常温暖的气候中可能会遇到困难。
创建哥伦比亚怀恩多特鸡的过程涉及选择性育种技术,旨在实现这种特定的颜色模式。它起源于19世纪晚期的美国,并在20世纪初获得官方认可。它们的外观是通过结合白色怀恩多特鸡和条纹普利茅斯岩鸡的组合而实现的,旨在培育出既美丽又实用的鸡。它们首次向公众介绍是在世界哥伦比亚博览会上,因此得名。
哥伦比亚怀恩多特鸡不仅因其美丽而受到赞赏,而且因其友好的气质,使它们成为经验丰富的养鸡者和初学者的绝佳选择。它们以良好的觅食能力而闻名,喜欢自由放养,并且能够对各种天气条件表现出相当的韧性,使其成为不同家禽设置的多功能品种。
有关哥伦比亚怀恩多特鸡的特性、历史、包括外观、气质和育种历史的更多详细信息,您可以参考由Chicken Fans【50†source】、The Happy Chicken Coop【51†source】和Browns’ Family Farmstead【52†source】提供的综合指南和描述。

基因座和等位基因: 深棕色(DB)基因座最初定位于横斑Fayoumi鸡的常染色体。这个基因座有两个等位基因:野生型黑胸(DBN)和深棕型(DBDB)。
DB表型的特征: DB表型的鸡具有类似哥伦比亚样式的真黑素限制表达,公鸡胸部为红褐色,母鸡为檀橙色或亮橙色。
SOX10基因的作用: 研究发现,DB表型是由于SOX10基因转录起始位点上游14 kb处发生的8.3 kb序列缺失导致的。这个缺失可能起到了增强子的作用,降低了SOX10基因的转录表达。
SOX10基因的作用: 研究发现,DB表型是由于SOX10基因转录起始位点上游14 kb处发生的8.3 kb序列缺失导致的。这个缺失可能起到了增强子的作用,降低了SOX10基因的转录表达。这句话什么意思?DB表型不是受到等位基因控制吧?SOX10就是这个等位基因吗?
理解这段话的关键在于分辨“基因座”、“等位基因”和特定基因(如SOX10基因)之间的关系,以及它们如何影响鸡的DB表型(即深棕色羽毛表型)。
基因座指的是染色体上特定基因的物理位置。每个基因座可以有多个版本的基因,这些版本称为等位基因。
等位基因是存在于同一基因座上的不同版本的基因,它们通过导致蛋白质的不同形式或调节基因表达的不同级别来影响个体的性状。
在讨论的上下文中,DB基因座上的两个等位基因(野生型黑胸DBN和深棕型DBDB)决定了鸡的羽毛颜色。这意味着,羽毛颜色的不同是由位于DB基因座上的这两种不同的等位基因版本决定的。
SOX10基因的作用被提到是因为它在DB表型的形成中起着关键作用。研究发现DB表型(深棕色羽毛表型)与SOX10基因上游14 kb处发生的8.3 kb序列缺失有关。这个缺失影响了SOX10基因的表达,可能通过降低它的活性来减少黑色素的合成,进而影响羽毛的颜色。
从这个角度看,SOX10基因和它的变异(8.3 kb序列缺失)并不直接等同于DB基因座上的等位基因,但它是影响DB表型显现的一个关键因素。SOX10基因的这个特定变异对于羽毛颜色的形成有重要影响,但这并不意味着SOX10就是DB基因座上的等位基因。相反,SOX10基因的这个变异可能影响多个与羽毛颜色相关的基因座和等位基因的表达。
总的来说,DB表型的形成是一个复杂的遗传过程,涉及多个基因和调控元件的相互作用,其中SOX10基因的8.3 kb序列缺失是影响该表型一个关键的分子事件。这个缺失可能作为一个调控元件(如增强子),影响SOX10基因以及可能的其他基因的表达,从而影响羽毛的颜色。
问:CR反应的设计: 为了明确DB基因座的分型,设计了3条引物进行PCR反应。使用的引物包括SOX10-1F和SOX10-1R(都位于SOX10基因外显子1内),以及SOX10-6R(位于8.3 kb缺失区)。
基因座分型的判断方法:
如果SOX10_1F和SOX10_1R扩增出611 bp的条带,而SOX10_1F和SOX10_6R没有扩增产物,表示为野生型纯合子(DBN/DBN),表现为黑色胸部的野生型羽色。
如果SOX10_1F和SOX10_6R扩增出1257 bp的条带,而SOX10_1F和SOX10_1R没有扩增产物,表示为DB纯合子(DBDB/DBDB),表现为深棕色表型。
如果两个目标片段(611 bp和1257 bp)都有扩增产物,则为杂合子(DBDB/DBN),也表现为深棕色表型
这个应该怎么理解呢?
这段描述涉及到使用聚合酶链反应(PCR)技术来分析SOX10基因的特定变异,这是判断鸡的DB基因座型的一种方法。这里用到了三条特定的引物(SOX10-1F、SOX10-1R和SOX10-6R)来针对SOX10基因的不同区域进行扩增,以检测8.3 kb的序列是否缺失,这个序列缺失与DB表型(深棕色羽毛)密切相关。下面是这个过程的简化解释:
引物的选择和作用:
- SOX10-1F和SOX10-1R都位于SOX10基因外显子1内,这对引物用来检测正常的SOX10基因片段。
- SOX10-6R位于8.3 kb序列缺失区,与SOX10-1F一起使用时,它可以帮助检测序列缺失的情况。
PCR结果的解读:
- 野生型纯合子(DBN/DBN):如果使用SOX10-1F和SOX10-1R引物扩增出611 bp的DNA片段,而SOX10-1F和SOX10-6R没有扩增出产物,这说明SOX10基因未发生8.3 kb的缺失,鸡为野生型纯合子,表现为黑色胸部的野生型羽色。
- DB纯合子(DBDB/DBDB):如果SOX10-1F和SOX10-6R扩增出1257 bp的DNA片段,而SOX10-1F和SOX10-1R没有扩增产物,这说明SOX10基因发生了8.3 kb的序列缺失,鸡为DB纯合子,表现为深棕色表型。
- 杂合子(DBDB/DBN):如果两组引物都能扩增出目标DNA片段(即611 bp和1257 bp),则说明鸡的一个等位基因上发生了8.3 kb的缺失,而另一个等位基因上没有,鸡为杂合子,这种情况下也表现为深棕色表型。
通过这种PCR检测方法,研究者可以准确地识别出鸡的基因型,进而推断出它们的羽毛颜色表型。这是一种常用于分子生物学研究的技术,可以用来研究基因的存在与否、变异情况以及基因表达的差异。
深入研究
*** 而白来航鸡主要为 DB 基因型,其表型由于 PMEL17 基因突变形成的显性白上位作用,抑制了 DB 表型特征红胸或黄褐色而呈现全身白***
根据研究,白来航鸡的显性白表型是由PMEL17基因中的突变引起的。这个基因对于黑色素细胞的发展起着关键作用,因此被认为是控制鸡羽毛颜色的主要位点之一。特别是,显性白表型与PMEL17基因中的插入/缺失多态性相关联。这些基因变异直接影响了羽毛颜色,导致白来航鸡呈现全身白羽的表型。
PMEL17基因中的具体突变形式包括插入和缺失变异,这些变异影响了该基因编码的蛋白质结构,从而影响黑色素的合成和分布。插入/缺失多态性的发现提供了关于如何在分子水平上控制鸡的羽毛颜色的重要见解。
这表明,PMEL17基因的突变并不是简单地通过改变等位基因版本来实现的,而是通过更复杂的分子机制,如编码区域的插入或缺失,从而改变蛋白质的功能,进一步影响羽毛颜色的表现。这种显性白上位作用的机制抑制了其他可能促使羽毛呈现红胸或黄褐色的基因座的影响,导致了白来航鸡羽毛的全白表型。
以上结论基于多项研究,其中涉及对PMEL17基因突变位点的检测方法,以及对突变如何影响羽毛颜色的深入分析【70†source】【71†source】。这些研究为理解鸡羽毛颜色的遗传控制提供了宝贵的信息,并为未来的育种计划提供了科学依据。
https://pubmed.ncbi.nlm.nih.gov/20176559/
2
RNA-seq最后分析会得到FPKM的一个表,那个表的第一列是基因IID好呢?还是转录本ID好呢?一般论文用的什么?
RNA-seq分析得到的FPKM(Fragments Per Kilobase of transcript per Million mapped reads)值是用来估计基因或转录本的表达量。在选择基因ID还是转录本ID作为表的第一列时,这取决于研究的目的和上下文。
基因ID:如果研究的重点是在基因水平上比较表达量,使用基因ID会更合适。这种情况下,一个基因可能包含多个转录本,但分析会聚焦于基因整体的表达量变化。在很多研究中,特别是那些关注特定基因如何在不同条件下调节的研究,使用基因ID是更常见的做法。
转录本ID:如果研究关注的是具体的转录本或者是要探索不同的剪接变异,那么使用转录本ID会更加合适。这允许研究者能够区分同一基因的不同转录本表达情况,这对于理解基因调控的复杂性和剪接事件非常重要。
一般论文使用什么:
- 在实践中,具体使用哪个ID往往取决于研究的具体目标和领域。在一些论文中,研究者可能会同时报告基因级别和转录本级别的分析结果,尤其是在基因表达差异分析和转录本多样性研究中。
- 基因级别的分析较为常见,特别是在那些关注特定基因表达差异、基因功能或疾病相关基因研究中。而在更细致地探究基因表达调控机制、剪接变体和功能性转录本等领域,转录本ID的使用则更为频繁。
总的来说,选择基因ID还是转录本ID作为FPKM表的第一列应当基于你的研究目标来决定。如果你的研究目的不明确或者两者都很重要,你可以考虑在你的分析中同时使用这两种标识符,并在论文中清楚地报告你的分析策略和发现。
将转录本ID的表格转换为基因ID的表格并不总是简单的一一对应关系,因为一个基因可能包含多个转录本。因此,直接将转录本ID替换为基因ID可能会导致信息的丢失或重复计算。在处理这个转换时,应当考虑以下几个步骤:
识别转录本和基因之间的关系:首先,需要确定每个转录本ID对应的基因ID。通常,转录本ID(如Ensembl数据库中的ENSGALT开头的ID)和基因ID之间的关系可以通过公共数据库查询得到,比如Ensembl、NCBI等。这些数据库提供了转录本到基因的映射信息。
汇总转录本表达量到基因级别:由于一个基因可能对应多个转录本,因此在将转录本FPKM值转换为基因级别时,你需要决定如何处理这些转录本的表达量。常见的方法包括:
- 求和:对同一基因的所有转录本的FPKM值求和,以得到该基因的总表达量。
- 平均:计算同一基因的所有转录本的FPKM值的平均值,作为该基因的表达量。这种方法可能需要根据转录本长度进行加权平均。
- 最大值:选取同一基因中FPKM值最高的转录本作为该基因的表达量,这假定最高表达的转录本最能代表基因的活性。
创建新表格:根据转录本到基因的映射关系以及选择的汇总方法,创建一个新的表格,其中第一列是基因ID,后续列是根据所选方法计算得到的基因级表达量。
考虑生物学重复和技术重复:在汇总表达量时,还应该考虑到来自不同生物学重复或技术重复的数据,确保统计处理的正确性。
这个过程可能需要编写脚本或使用现有的生物信息学工具来自动化。在进行这种转换时,重要的是要明确记录你使用的每个步骤和方法,以便在将来的分析和发表论文时可以清楚地解释你的数据处理流程。如果你需要具体的帮助来实现这个过程(例如,使用Python脚本进行转换),请提供更多的细节。
如果你的研究目的是探索哪些基因可能在小鼠的昼夜节律中发挥作用,那么选择基因ID作为分析的主要对象会更合适。这是因为你关注的是在整个基因水平上的表达变化,而不是特定转录本的差异表达。具体来说,你会想要分析不同时间点(每隔4小时取样)基因表达量的变化,以识别哪些基因的表达具有显著的周期性,从而可能参与调控小鼠的昼夜节律。
在这种研究背景下,分析步骤可能包括:
数据预处理:首先,需要对RNA-seq数据进行质量控制、比对到参考基因组以及计算每个基因在每个样本中的FPKM值。
基因表达量的周期性分析:使用适当的统计方法或生物信息学工具(例如,R包如
rain,circaCompare,JTK_CYCLE等)来分析基因表达量的时间序列数据,以识别表达量显示出昼夜节律变化的基因。显著性测试:对于识别出的周期性表达的基因,进行统计显著性测试,确定这些周期性变化是否具有统计学意义。
功能注释和富集分析:对具有显著节律性表达的基因进行功能注释和富集分析(例如,GO(Gene Ontology)分析和KEGG(Kyoto Encyclopedia of Genes and Genomes)路径分析),以探索它们可能参与的生物学过程和路径。
使用基因ID作为分析的主要对象,你可以更直接地探究整个基因层面上的表达模式和功能,这对于理解昼夜节律背后的基因调控网络至关重要。此外,由于你的研究重点是在基因水平上,这也简化了分析流程,因为不需要对每个基因的多个转录本进行单独分析。
在报告你的发现时,清晰地描述你的数据处理和分析流程,包括如何选择基因ID、如何进行周期性分析和显著性测试,以及你如何解释功能注释和富集分析的结果,这将对读者理解你的研究成果非常重要。
利用混合样本池法对鸡显性白羽基因 PMEL17 突变 位点的检测
利用 混合样本池法 对鸡 显性白羽基因 PMEL17 突变 位点的检测
摘要:
显性白羽基因座是影响鸡羽色形成的重要基因座位之一,
1, 该基因座上的显性等位基因 I 会抑制黑色素合成, 从而使携带该基因的个体全身羽毛呈现白色。
2,目前已确认鸡显性白羽基因座编码 PMEL17 蛋白: 是一种黑素细胞特异性蛋白, 在黑素细胞的分化与成熟中起到重要作用, 并证明 PMEL17 基因的突变与显性白羽的形成有关。
3,文章利用混合样本池建立了一种低成本、高效率, 并能在大规模群体中检测 PMEL17 基因突变的方法,:
称为 PCR 产物混合样本池法。该方法的基本步骤如下:
首先, 提取个体基因组 DNA, 并设计相关引物对每一个体单独进行 PCR 扩增;
其次, 将 PCR 产物等比例混合, 10 个样品混在一个池中;
然后, 将 PCR 产物混合池样品于非变性聚丙烯酰胺凝胶上进行电泳;
最后, 待电泳结束后进行银染, 根据凝胶上所显条带判定是否存在突变体。
此外, 文章还将这种方法与传统基因组 DNA 混合样本池法进行了比较试验, 并利用该方法对试验鸡群显性白羽基因 PMEL17 突变进行检测, 证实该方法具有较高准确度。
摘要:
显性白羽基因座是影响鸡羽色的一个重要基因
座位, 该基因座位上的显性等位基因I 对色素沉着,
尤其是真黑色素的合成有抑制作用[1]。目前在该基
因座共发现 4 种等位基因: I (显性白羽), IS(Smoky,
烟灰色羽色), ID (Dun, 暗褐色羽色), i(野生型)。相
关研究表明, 鸡PMEL17 基因编码区的插入或缺失
突变与这些等位基因形成有关[2]。鸡PMEL17 基因包
括 12 个外显子, 全长约 4.0 kb, 共编码约 788 个氨
基酸(GenBank登录号: AY636127.1)。该基因编码一
种黑素细胞特异性蛋白, 这种存在于前黑素体基质
中的蛋白对黑色素聚积以及黑素细胞的正常发育起
着重要作用[3]。Kerje等[2]对红色原鸡、白来航等不
同品种鸡PMEL17 基因序列的分析中, 发现其第 10
外显子上的 9 bp插入突变是显性白羽性状形成的主
要原因, 所对应的等位基因为I, 而隐性等位基因i不
含该突变
这段描述涉及的是鸡的羽色遗传学,尤其是与显性白羽基因座(也就是影响羽色的特定基因区域)相关的研究。这里提到的基因座是一个遗传上的位置,它控制着鸡羽色的特征。在这个基因座上,发现了四种不同的等位基因,这些等位基因决定了鸡羽毛的颜色。
I (显性白羽): 这是一种显性等位基因,它可以抑制色素(特别是真黑色素)的合成,导致鸡拥有白色的羽毛。显性基因意味着只需要一个I基因(从父母中的任何一个那里继承)就足以表达出白羽特征。
I*S (Smoky, 烟灰色羽色): 这种等位基因导致一种烟灰色的羽色。它与I基因位于同一个基因座,但产生不同的羽色效果。
I*D (Dun, 暗褐色羽色): 类似地,这种等位基因造成鸡羽毛呈现暗褐色。
i (野生型): 这是一种隐性基因,存在于野生类型的鸡中,它不会抑制色素的合成,使得鸡能够表现出正常的羽色。
这些等位基因在同一基因座上相互竞争,决定了鸡羽毛的颜色。由于I基因是显性的,它在存在时会覆盖其他等位基因的效果,导致鸡羽毛变白。而IS和ID基因则分别导致烟灰色和暗褐色的羽毛。最后,i基因作为隐性基因,只有在没有其他显性基因(如I)存在的情况下才表现其特征,即野生型羽色。
研究表明,这些等位基因的差异可能与PMEL17基因编码区的插入或缺失突变有关。PMEL17基因编码一种特定的蛋白质,这种蛋白质在黑色素的形成和黑素细胞的正常发育中起着重要作用。特别是,显性白羽特征(由I等位基因控制)被发现与PMEL17基因的第10外显子上的9bp插入突变有关,而隐性的野生型等位基因i则不包含这种突变。
基因座,和这些等位基因的关系,能用图给我讲明白吗?
想象一个基因座像是一本书的特定章节位置,而等位基因则像是这一章节中可能出现的不同版本的故事。这个比喻可以帮助我们理解基因座和等位基因之间的关系。
基因座:这是基因组中一个特定的位置,可以想象成书中的一个章节的位置。就像章节是书中不同内容的位置一样,基因座是染色体上控制特定遗传特征(如羽色)的基因的位置。
等位基因:这些是存在于基因座上的基因的不同变体,可以想象成同一章节的不同版本的故事。每个版本都有细微的差异,这些差异导致了不同的物理表现或特征(如不同的羽色)。
在这个比喻中,I, I*S, I*D, 和 i 是同一基因座(章节位置)上的不同等位基因(章节版本)。每种等位基因都编码了不同的指令,影响着鸡羽毛的颜色。
接下来,我会用图形来展示这个概念,帮助你更好地理解。
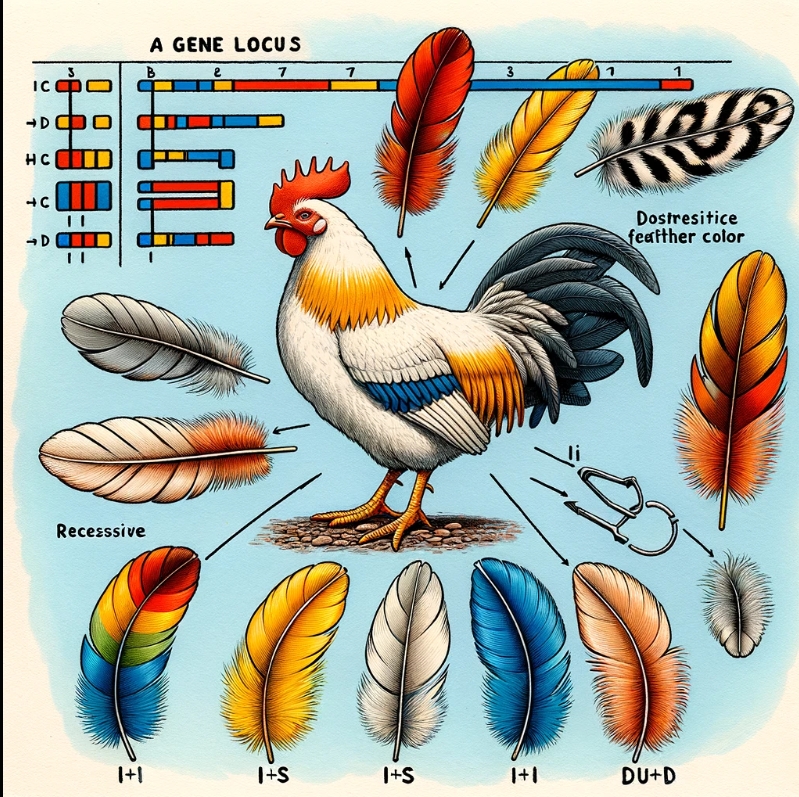
- 我们将展示一个基因座和四种等位基因之间的关系。
- 说明如何通过这些等位基因的不同组合来解释不同的羽色表型。
让我先绘制这个图。
上图展示了基因座和其四种等位基因(I, IS, ID, 和 i)之间的关系,这些等位基因负责决定鸡的羽色。图中明确表示了基因座是染色体上的一个特定位置,而I, IS, ID, 和 i作为这个位置的不同版本,导致了不同的羽色结果:I等位基因导致白色羽毛,IS导致烟灰色羽毛,ID导致暗褐色羽毛,而i等位基因则保持野生型的羽色。
图中还包含了标识显性(I)和隐性(i)等位基因的视觉符号,帮助解释这些等位基因如何通过它们的组合在同一基因座上产生多样的羽色表型。希望这个图形能帮助你更好地理解基因座与等位基因之间的关系及其对鸡羽色的影响。
混合样本池法(又称DNA pooling法)是随着基因
诊断技术的发展而建立起来的一种快速高效的基因
型判定方法。混合样本池法最初主要应用于各种遗
传疾病的基因型诊断, 后来逐渐应用到基因定位及
连锁分析中[4,5]。对大规模群体进行特定基因的基因
型判定时, 如果对每只个体分别进行判定无疑会耗
费大量成本, 混合样本池法可以很好地解决这一问
题。这种方法是将个体基因组DNA经稀释后, 按照
一定比例混合在一起, 然后利用混合后的样本即混
合样本池为模板进行相关基因PCR扩增, 并通过电
泳或其他基因判定方法进行基因型分析、突变检测、
连锁分析或基因定位等[5]。混合样本池中的每一个
池可以包含多个个体的基因组DNA, 因此, 可以一
次获得大量的基因信息, 在提高检测强度的基础上,
还 1 能降低相应成本。Lipkin等[6]运用该方法, 对抗
马立克病的鸡和易感鸡进行了相关研究, 找到了与
抗马立克病有关的QTL位点
目的
本研究尝试建立一种基于样本混合池法的高
效、低成本并能在大规模群体中判定鸡 PMEL17 基
因型的相关检测手段, 并利用此方法对不同品种鸡
的 PMEL17 基因突变位点进行检测, 从而对该方法
的准确度、精确性以及适用性进行评定
这篇论文的结构和目录概括如下:
材料和方法
- 1.1 实验动物
- 1.2 方法
- 1.2.1 血样采集及DNA提取
- 1.2.2 引物设计
- 1.2.3 PCR扩增体系和条件
- 1.2.4 混合样本池的构建
- 1.2.5 电泳检测
结果与分析
- 2.1 两种混合样本池法检测效能比较
- 2.2 不同品种鸡PMEL17基因突变位点检测
讨论
- 3.1 不同品种鸡PMEL17基因的突变状况
- 3.2 混合样本池法的检测效能
- 3.3 实验成本和工作量
此外,文章还包含了参考文献部分。这篇论文详细介绍了一种低成本、高效率的检测方法,旨在检测大规模群体中的鸡显性白羽基因PMEL17突变位点。
论文的结论主要有以下几点:
PCR产物混合样本池法的适用性:研究表明,使用PCR产物混合样本池法对鸡显性白羽基因PMEL17突变位点进行检测是一种高效、低成本的方法,适合在大规模群体中进行基因突变的检测。这种方法具有足够的准确性和灵敏性,能够在包含少量突变样本的大量样本池中检测出特定的基因突变。
显性白羽基因突变的检测:通过实验,确认了在白来航蛋鸡群体中显性白羽基因PMEL17的第10外显子存在9bp插入突变,表明这种突变与显性白羽性状的形成有直接关联。
混合样本池法与传统方法的比较:与直接使用基因组DNA混合池进行PCR扩增相比,PCR产物混合样本池法在检测精度上具有明显优势,尤其是当隐性突变样本含量较低时,这种方法的检测优势更为明显。这说明PCR产物混合样本池法能更有效地检测到低频率的突变。
实验成本和工作量的评估:研究还比较了两种不同的混合样本池法(基因组DNA混合池和PCR产物混合样本池)在实验成本和工作量方面的差异。虽然PCR产物混合样本池法在某些方面增加了实验成本,但它提高了检测效率,且在普通电泳条件下能获得较好的检测效果,因此是一种经济有效的方法。
总的来说,该研究成功地建立了一种基于样本混合池法的高效、低成本的鸡显性白羽基因突变检测方法,对于大规模的遗传研究和育种程序具有重要的应用价值。
感觉会很有趣,龙眼花芽诱导过程内源激素、mRNA-lncRNA转录组分析及TFL1启动子研究
转录组分析揭示了两种蛇类视觉系统昼夜节律相关的表达特征
转录组 分析揭示了 两种蛇类视觉系统 昼夜节律相关 的表达特征
摘要:目前大多数相关研究主要集中于哺乳动物、鸟类和鱼类,揭示了其视
网膜形态的适应性演化,当前也有部分研究报道了不同昼夜节律的蛇中视网膜细
胞形态的多样性,但少有研究利用转录组学方法,集中于视觉相关基因的表达上。
不同昼夜习性的蛇类视觉有何差异?这些差异背后的分子机制是什么?这些问
题仍有待解决。
为了探讨这些科学问题,本研究选取了两种不同昼夜节律模式的蛇类——
绿瘦蛇(Ahaetulla prasina,昼行性)和黄链蛇(Lycodon flavozonatum,夜行性),
取心脏、肝脏、肺、肾脏、肌肉、眼睛六种组织进行转录组测序,
利用测序数据对两种蛇类的转录本各自进行从头组装并注释。
加权基因共表达网络分析(WGCNA)分别得到了与两种蛇类眼睛最相关的基因模块,进行功能富集分析
后发现,两个模块内的基因分别发挥了不同的生物学功能。
昼行相关基因主要参与了神经系统发育、离子跨膜转运、G 蛋白偶联受体信号通路等信号传导相关功能,
而夜行相关基因主要富集在视觉感知,光感受器和光传导等条目。
结果表明:
绿瘦蛇眼睛中丰富的视觉信号传导基因增强了其对日光条件的适应能力和快速
反应能力,
而黄链蛇眼睛中光感相关基因的优势则增强了其在黑暗环境中的对光
的敏感性。
同时,结合差异表达分析发现,三种视蛋白基因分别在两种蛇类中差
异表达,视杆光感受器基因在黄链蛇眼睛中高表达,以适应昏暗的光条件,并表
现出对短波长光的敏感,
而绿瘦蛇的眼睛对长波长光更敏感,并以视锥光感受器
为主,这可能是由于过滤了较短波长的光以增强视觉敏锐度。另外,进一步的分
析发现,在视觉重要的光传导通路上,两种蛇的眼睛都有特定的光传导相关基因
表达,尤其是,视锥细胞相关基因在绿瘦蛇中特异高表达而视杆细胞相关基因在
黄链蛇中特异高表达,这一结果与两种蛇类的昼夜节律模式是相一致的,提示了
I
转录组分析揭示了两种蛇类视觉系统昼夜节律相关的表达特征这些基因表达的适应性进化。
本研究以两种不同昼夜节律的蛇类为代表,解析了蛇类视觉相关基因的昼夜
节律适应表达模式,为蛇类视觉适应的遗传研究提供了重要的基础,并为进一步
了解蛇的适应性进化提供了依据
关键词:转录组;游蛇科;视觉适应;光传导;昼夜节律
AI修图
“AI修图”指的是使用人工智能技术进行图像编辑和增强的过程。这可以包括图像去噪、分辨率提升、对象去除或添加、风格转换等各种操作。近年来,随着深度学习技术的发展,很多工具和服务开始提供基于AI的图像编辑功能,这些工具既有在线服务,也有桌面软件。以下是一些流行的选项:
在线服务
- Adobe Photoshop Express:Adobe提供的在线简版Photoshop,包括一些基本的AI驱动的编辑功能,如自动增强等。
- Remove.bg:专注于使用AI去除图像背景的工具,适用于快速清除或更改图像背景。
- Let’s Enhance:能够通过AI提升图像的分辨率和清晰度,适合对老照片或低质量图像进行增强。
桌面软件
- Adobe Photoshop:最新版本的Photoshop包含了多种AI驱动的工具,如内容感知填充、智能选区、图像增强等。
- Topaz Labs:提供一系列图像处理软件,特别是在去噪、锐化、放大等方面,使用AI技术达到非常专业的效果。
- GIMP:虽然GIMP本身不是基于AI的,但有很多插件和外部工具可以实现类似的功能。
使用注意事项
- 在学术和研究环境中,任何对图像的修改都需要保持数据的真实性和完整性。例如,在生物学或医学研究中,任何可能影响结果解释的图像处理都必须被清楚地标注和解释。
- 在选择使用这些工具时,请确保它们符合你的具体需求,并且你了解如何正确地应用它们以避免误导读者或破坏图像的科学价值。
AI修图工具可以大大简化图像编辑的工作,但使用时应该谨慎,确保图像的修改不会误导观众,特别是在学术和科研领域。
排版论文或进行微调时,使用AI修图主要目的是提升图像的质量、适应论文格式要求,或改善图像在文档中的表现。虽然目前大多数AI修图工具更侧重于图像内容的编辑,如背景移除、图像增强等,但也有一些工具和方法可以帮助你在排版和微调论文时提升图像质量。这里介绍几种适用的方法和工具:
图像质量提升
- 使用AI增强工具:工具如Let’s Enhance或Topaz Labs可以提升图像的分辨率,使图像在放大时仍保持清晰。这对于需要在论文中放大展示的图像特别有用。
- 调整图像对比度和亮度:一些在线工具,比如Adobe Photoshop Express,提供自动调整图像对比度和亮度的功能,可以让图像在论文中看起来更加清晰。
图像格式调整
- 裁剪和调整图像尺寸:确保图像符合论文要求的尺寸和比例。许多AI工具,包括在线服务和桌面软件,都提供裁剪和调整尺寸的功能。
- 调整图像方向和布局:在排版时,可能需要调整图像的方向或布局以适应文档的流程。这些调整可以使用基本的图像编辑工具完成,不一定需要AI功能。
图像去噪和锐化
- 图像去噪:AI去噪工具,如Topaz DeNoise AI,可以去除图像中的噪点,使图像看起来更加清晰。
- 图像锐化:AI锐化工具,如Topaz Sharpen AI,有助于恢复图像细节,特别是在图像由于压缩或放大而失真时。
自动化排版工具
虽然不直接关联到AI修图,但一些自动化排版工具可以帮助你更高效地整合和排布图像和文本,例如:
- LaTeX:一个高级排版系统,可以生成高质量的科学和数学文档。通过使用一些扩展包,比如
graphicx,可以更好地控制图像的插入和布局。 - Microsoft Word 或 Google Docs 插件:一些插件提供了改进的图像处理和排版功能,有助于自动化论文的排版过程。
使用这些工具和方法时,重要的是要记住,所有图像的修改和调整都应保持数据的真实性和准确性,尤其是在科研和学术论文中。此外,确保遵守论文提交要求中关于图像质量和格式的指导原则。
鸡Wnt基因家族生物信息学分析及表达,这个方法感觉很重要
摘 要:【目的】试 验 旨 在 对 鸡 Wnt 基因家族 进行全面系统地调查分析,并进一步明确鸡感染新城疫病毒
(Newcastlediseasevirus,NDV)后 Wnt基因家族成员的表达模式。
【方法】基于鸡基因组测序结果, 通过查询 Pfam和 UniPort数据库 获取鸡 Wnt基因家族全基因组序列,构建系统进化树,
分析 Wnt蛋白理化性质、亚细胞蛋白定位、染色体分布、保守结构域等,并进一步研究鸡感染 NDV 后 Wnt基因家族成员的表达模式。
【结果】试验共鉴定
到39个鸡 Wnt基因家族成员。系统进化树分析发现,
1,鸡 与 人 和 鼠 的 Wnt成员相似性较高,均 有12个 亚 家 族,即Wnt1、Wnt2(Wnt2a 和 Wnt2b)、Wnt3a、Wnt4、Wnt5(Wnt5a 和 Wnt5b)、Wnt6、Wnt7(Wnt7a 和 Wnt7b)、Wnt8(Wnt8a和 Wnt8b)、Wnt9(Wnt9a和 Wnt9b)、Wnt10a、Wnt11及 Wnt16成员,
2,但鸡缺少 Wnt10b。鸡 Wnt成员分布于11条染色体上,分别为 Chr1、Chr2、Chr4、Chr6、Chr7、Chr12、Chr13、Chr21、Chr26、Chr27和 Chr34,成 员 间 出 现14次串联重复,且无节段重复出现。
3,39个鸡 Wnt成员 中 共 识 别 出10个 保 守 基 序,其3′-端 较 为 保 守,除rna-XM_040653060.2(Wnt6)和rna-XM_040650634.2(Wnt5a)外,其余成员3′-端均由基序2和5组成;39个鸡 Wnt家族成员均含保守区域,即基序1和3。转录组数据分析结果表明,鸡 在 感 染 NDV 过 程 中 Wnt7a表达量显著上调。
【结论】鸡 Wnt基因家族成员在染色体上呈现非均匀分布,其基因结构较为保守,且 串 联 重 复 是 Wnt基因家族成员的
主要扩增方式。Wnt7a可能与 Wnt调节巨噬细胞参与微生物感染过程密切相关
关键词:鸡;Wnt基因家族;生物信息学;表达;进化树
方法好好记录一下
材料与方法
1.1 Wnt全基因组鉴定
从 NCBI数据库中获取鸡的基因组和注释文件数据。为了鉴定鸡中所有可能的 Wnt基因,对隐马尔 可 夫 模 型 (PF00110.22)和 UniPort 数 据 库
(https:∥ www.uniprot.org/)中 经 审 查 的 8 条Wnt 序 列 (sp|P49339|WNT11、sp|P49337|WNT4、sp|Q91029|WNT1、sp|Q98SN7|WNT2B、
sp|Q2LMP1|WNT3A、sp|P51030|WNT8C、sp|
O42280|WNT9、sp|Q3L254|WNT7B)
进 行 比 对,
同时 从 UniPort 数 据 库[9-10]中 下 载 经 审 查 的 人
(19条)和鼠(19条)Wnt基因序列
1.2 Wnt 蛋白性质分析
对1.1中 获 取 的 序 列 使 用 集 成 于 MegaX[11-12]中的 MUSCLE进行序列对齐,并通过1000次引导测试构 建 系 统 进 化 树。
使 用 ExPASy 在 线 软 件(https:∥www.expasy.org/)预 测 鸡 Wnt蛋 白 的等电点和分子质量[13];
使用 WoLFPSORT 在线软件(https:∥wolfpsort.hgc.jp/)预测鸡 Wnt蛋白的亚 细 胞 定 位[14];
使 用 TMHMM 2.0 在 线 软 件(https:∥services.healthtech.dtu.dk/service.php?TMHMM-2.0)获 取 鸡 Wnt蛋 白 的 跨 膜 蛋 白 数 和序列 长 度[15];
使 用 MEME 在 线 软 件 (https:∥meme-suite.org/meme/)分 析 鸡 Wnt蛋 白 的 保 基序[16];使用 TBtools软件进行可视化[17];在 NCBI数据库中验证蛋白功能结构域(CDD)[18]。
1.3 鸡感染 NDV时 Wnt的表达模式分析
从 NCBI数据库中获取6组转录组数据,分 别为 未 受 NDV 感 染 的 对 照 组 (登 录 号 分 别 为
SRR13021942、SRR13021943、SRR13021946)和 受NDV 感染的处理组(登录号分别为 SRR13021947、
SRR13021955、SRR13021956),
将 两 组 的3个 重 复均 一 化 后,通过各组数据中每千碱基百万片段(FPKM)值对 Wnt表达谱进行评估,并采用log2 进行转换归一化
你的问题涉及到转录组数据分析的一系列步骤,我将尽量简明地解释每个步骤及其意义。
转录组数据分析流程概述
获取数据:首先,你从NCBI数据库获取了六组转录组数据,分别代表对照组和处理组(受NDV感染)的样本。这些数据通常以测序读段(reads)的形式提供,每个读段是一个短的DNA片段序列。
质量控制:在进行下一步之前,通常需要对原始测序数据进行质量控制,移除低质量的读段和污染。
读段比对:将清洗后的读段比对到参考基因组或转录组上。这一步是为了确定每个读段在基因组中的位置。
计算表达量:在读段成功比对后,下一步是计算每个基因的表达量。FPKM(每千碱基百万片段)是一种常用的表达量衡量方式,用于描述某个基因表达水平的标准化值。
均一化和FPKM
均一化(Normalization):在生物信息学中,均一化是数据处理的一种方法,旨在消除实验设计和测序过程中的偏差。例如,不同样本的测序深度(即测序的总读段数)可能不同,直接比较原始的读段计数可能会误导。因此,需要进行均一化处理来确保样本间的可比性。
FPKM:FPKM代表“每千碱基百万片段”。它是一种衡量基因表达量的标准化方法,考虑了基因长度和测序深度的影响,从而使不同样本或基因之间的表达量可比。计算FPKM之前已经包括了一种均一化过程,以消除样本间测序深度和基因长度变异带来的影响。
log2转换
- log2转换:在得到FPKM值后,为了使数据更适合统计分析,通常会进行log2转换。这一转换有几个好处:首先,它可以帮助稳定方差,因为大的FPKM值的波动对结果的影响会比小的FPKM值大;其次,log转换后的数据更接近正态分布,这对许多统计测试来说是一个重要的前提条件。log2转换也便于在处理组和对照组之间的比较中,用对数比率直观地表示表达量的变化。
总结
所以,你的数据处理流程大致为:从NCBI获取数据 → 质量控制 → 读段比对 → 计算FPKM(已包含一种均一化) → 对FPKM值进行log2转换以进一步归一化和方便分析。
每一步都有其特定的目的和重要性,确保了最终得到的数据既可比较又适合进行生物学上的解释和分析。
下载NCBI数据库中的转录组数据并分析通常涉及多个步骤和不同的生物信息学工具。以下是一般的流程和相应代码示例,使用的是一些流行的命令行工具。请注意,实际分析可能需要根据具体数据和研究目标进行调整。
步骤1: 下载数据
使用SRA Toolkit的fastq-dump工具从NCBI的SRA数据库下载数据。安装SRA Toolkit后,可以使用以下命令下载数据:
1 | fastq-dump --split-files SRR13021942 |
--split-files参数用于将成对的测序数据分开下载。
步骤2: 质量控制
使用FastQC进行质量检查,并用Trimmomatic或fastp进行质量控制,去除低质量的序列。
1 | fastp -i SRR13021942_1.fastq -I SRR13021942_2.fastq -o SRR13021942_1_trimmed.fastq -O SRR13021942_2_trimmed.fastq |
重复上述命令,替换相应的SRR编号以处理所有样本。
步骤3: 读段比对
使用HISAT2、STAR或Bowtie2等工具将读段比对到参考基因组。
以HISAT2为例:
1 | hisat2 -x reference_genome_index -1 SRR13021942_1_trimmed.fastq -2 SRR13021942_2_trimmed.fastq -S SRR13021942_aligned.sam |
这里reference_genome_index是参考基因组的索引,需要预先建立。重复上述命令处理所有样本。
步骤4: 计算FPKM
使用StringTie或Cufflinks从比对结果中计算FPKM。
以StringTie为例:
1 | stringtie SRR13021942_aligned.bam -G reference_annotation.gtf -e -B -o SRR13021942.gtf |
这里reference_annotation.gtf是参考基因组的注释文件。
步骤5: log2转换
log2转换通常在数据汇总和分析阶段进行,这可以使用R语言来完成:
1 | # 假设df是包含FPKM值的数据框架,其中一列名为FPKM |
注意事项
- 每一步骤的具体命令和参数可能需要根据你的数据和实验设计进行调整。
- 参考基因组的索引建立、注释文件等需要根据你的目标物种准备。
- 上述过程中的每个步骤都可能需要相当的计算资源,尤其是读段比对和表达量计算步骤。
这个流程是比较通用的,但根据具体的项目要求和数据特点,可能还需要其他的步骤或调整。
2 结 果
2.1 鸡Wnt基因的鉴定
1,通过比对隐马尔可夫模型共获得39条蛋白序列,
2,通 过 比 对 UniPort中 8 条 验 证 序 列 同 样 获 得39条蛋白序列,
3,去掉重复后,最终获得39条鸡 Wnt基因序列。
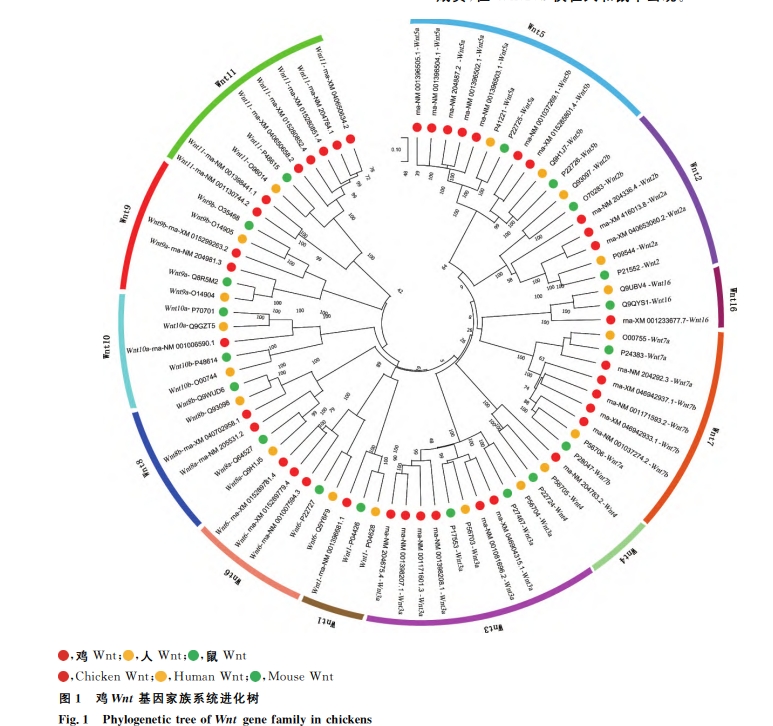
对鸡、人和鼠的 Wnt基因家族进行了系统进化树构建,结果见
图1。由图1可知,3个物种间均具有 Wnt1、Wnt2(Wnt2a和 Wnt2b)、Wnt3a、
Wnt4、Wnt5 (Wnt5a 和 Wnt5b)、Wnt6、Wnt7
(Wnt7a 和 Wnt7b)、Wnt8(Wnt8a 和 Wnt8b)、
Wnt9(Wnt9a和 Wnt9b)、Wnt10a、Wnt11及 Wnt16
成员,但 Wnt10b仅在人和鼠中出现
这句话描述的是一项生物信息学研究的过程,我会分几部分帮你理解:
隐马尔可夫模型(Hidden Markov Model, HMM):这是一种统计模型,用来描述一个含有隐状态的马尔可夫过程。在生物信息学中,隐马尔可夫模型经常用来识别或比对蛋白质或者核酸序列中的特定模式,比如蛋白质家族、基因家族、功能域等。
蛋白序列:这指的是蛋白质的构成,蛋白质由20种不同的氨基酸按照特定顺序链接而成。蛋白序列通常用一系列字母来表示,每个字母代表一种氨基酸。
UniProt数据库:UniProt(Universal Protein resource)是一个重要的蛋白质信息资源,包含大量的蛋白质序列和它们的功能信息。研究人员经常使用这个数据库来查找和比对蛋白质序列。
去掉重复后,最终获得39条鸡Wnt基因序列:这意味着在使用隐马尔可夫模型和通过UniProt数据库比对的过程中,总共识别出了39条不同的鸡Wnt基因序列。这些序列在两个不同的方法中被识别,但实际上是指向同一组39条独特的序列,表示他们没有发现新的或额外的序列超过这39条。
Wnt基因家族:Wnt基因家族是一组编码Wnt蛋白的基因。Wnt蛋白是一类在多种生物过程中发挥作用的信号分子,包括细胞生长、发育、分化等。Wnt信号通路是细胞通讯中的一个重要环节,影响动物从发育早期到成年后的多种生理过程。
系统进化树构建:系统进化树是一种表示生物间进化关系的图表。通过构建鸡、人和鼠的Wnt基因家族的系统进化树,研究人员可以分析不同物种间Wnt基因的进化关系,理解它们的相似性和差异性,以及这些基因是如何随着时间在不同物种中演化的。
简而言之,这句话描述了通过使用隐马尔可夫模型和UniProt数据库比对获得了39条独特的鸡Wnt基因序列,并进一步分析了鸡、人和鼠Wnt基因家族的进化关系。
要复现这里描述的研究过程,通常需要遵循以下步骤:
1. 准备数据和工具
- 收集蛋白质序列数据:确定研究对象,例如鸡的Wnt基因家族。需要从公共数据库(如NCBI、Ensembl等)中收集这些基因的蛋白质序列。
- 获取分析工具:安装并配置隐马尔可夫模型(HMM)分析工具(如HMMER)和系统进化分析软件(如MEGA、PhyML、或R包如
ape和phytools)。
2. 使用隐马尔可夫模型筛选蛋白序列
- 构建或获取HMM模型:如果针对的是已知的蛋白质家族,可以直接从PFAM等数据库获取相关的HMM模型。如果是新的研究对象,可能需要先通过已知序列构建HMM模型。
- 运行HMM分析:使用HMMER等工具,通过HMM模型在目标物种的蛋白序列数据库中搜索匹配序列。
3. 通过UniProt数据库比对序列
- 序列比对:使用BLAST或其他序列比对工具,在UniProt数据库中搜索你的序列,找到相应的蛋白质序列。
- 确认序列的准确性和相关性:验证比对结果,确保找到的序列确实属于研究的蛋白家族。
4. 去除重复序列
- 合并序列集合:将HMM分析和UniProt比对的结果合并。
- 去除重复项:使用序列分析软件(如CD-HIT或手动比对)识别并去除重复的蛋白序列。
5. 构建系统进化树
- 序列比对:使用ClustalW、MAFFT或其他序列比对工具对所有独特的蛋白序列进行比对。
- 选择合适的进化模型:根据序列数据的特点选择合适的进化模型。
- 构建系统进化树:使用MEGA、PhyML、BEAST等工具,根据比对结果和选定的模型构建系统进化树。
- 分析和解释进化树:分析进化树以了解不同物种间的进化关系。
6. 结果解释与报告
- 撰写研究报告:整理分析过程和结果,撰写研究报告或论文。
- 进一步分析:根据需要,可能还要进行进一步的生物信息学分析或实验验证。
注意事项
- 数据的质量和选择:确保所选序列的质量和相关性,这将直接影响分析的准确性。
- 参数的调整:在使用各种工具和软件时,可能需要根据具体情况调整参数,以获得最佳结果。
- 计算资源:系统进化分析可能需要较高的计算资源,尤其是在处理大量序列时。
这是一个基本的指南,具体步骤可能根据研究目标和可用资源有所不同。研究生物信息学通常需要一定的生物学、统计学和计算技能。
2.2 鸡Wnt基因家族成员信息分析
由表1可知,鸡 Wnt基因家族成员的等电点范围为7.46~9.63,表明该家族成员整 体 偏 碱 性;
分子质量 预 测 范 围 为30.61~44.29ku,
蛋 白 序 列 长度为272~396个氨基酸。亚细胞定位显示,
鸡Wnt基因家族大部分蛋白(32条)分泌到细胞外(extr),
2条定位于线 粒 体 基 质(mito),2 条定位于细胞核
(nucl),各有1条分别定位于细胞质膜(plas)、细胞
质核(cyto_nucl)及细胞质线粒体基质(cyto_mito)。
跨膜蛋白预测结果显示,Wnt基因家族α-螺旋跨膜数较少,只有17条蛋白发生1次跨膜。表明鸡Wnt基因家族蛋白性质差异不大,较为稳定
2.3 鸡 Wnt蛋白功能结构域
对39条鸡 Wnt蛋白功能结构域 进 行 分 析,共预测 出12种 Wnt
分 别 为 Wnt1、Wnt2(Wnt2a和
Wnt2b)、Wnt3a、Wnt4、Wnt5(Wnt5a和 Wnt5b)、
Wnt6、Wnt7(Wnt7a 和 Wnt7b)、Wnt8(Wnt8a 和
Wnt8b)、Wnt9(Wnt9a和 Wnt9b)、Wnt10a、Wnt11
及 Wnt16(图2)
没看完,不想看了。。。下次再看
知识点:
什么是基因家族?
基因家族是指一组由共同祖先基因演化而来的基因,这些基因在结构和功能上具有相似性。这种相似性通常是由于它们编码的蛋白质结构或功能上的相似,这些蛋白质可能参与相似的生物学过程或者具有相似的生化活性。基因家族成员之间的相似性是通过基因复制事件产生的,随后可能经历了功能上的分化(即获得了新的功能)或保守(即保持原有功能)。
在基因组内,基因家族的形成是生物进化的重要机制之一。基因复制可以通过不同的方式发生,包括基因组复制(整个基因组的复制)、染色体复制(整条染色体的复制)和基因复制(单个基因的复制)。这些复制事件可以增加基因组的复杂性,并通过产生基因冗余来为功能创新提供原材料。
随着时间的推移,复制的基因可能会发生突变,这些突变有时会导致新的功能的出现或现有功能的改进。这种机制使得生物能够适应环境变化,是生物多样性和复杂性增加的驱动力之一。
例如,血红蛋白家族就是一个由多个相关基因组成的基因家族,这些基因编码不同类型的血红蛋白亚单位,它们在氧气的运输和释放中发挥作用。另一个例子是细胞周期控制蛋白家族,这个家族中的蛋白质调控细胞的生长和分裂。
了解基因家族及其成员之间的关系对于揭示生物体内复杂的分子网络、了解基因如何在进化过程中获得新功能,以及开发针对特定蛋白质家族成员的药物治疗都具有重要意义。
在生物信息学中,基因家族要怎么分析?
在生物信息学中,分析基因家族涉及多个步骤,旨在识别相似的基因,了解它们的进化关系,以及推断它们的功能。以下是一个基本的分析流程:
1. 序列收集
- 数据库搜索:使用公共数据库(如NCBI、Ensembl、UniProt)搜索已知的基因或蛋白质序列。
- 同源性搜索:利用BLAST(Basic Local Alignment Search Tool)或者更先进的工具(如HMMER,基于隐马尔可夫模型的搜索工具)对特定物种的基因组数据进行同源性搜索,以识别可能属于同一基因家族的候选基因。
2. 序列比对
- 多序列比对:使用ClustalW、MAFFT、MUSCLE等工具对候选基因序列进行多序列比对,以识别它们之间的相似性和保守区域。
3. 构建进化树
- 系统发生分析:基于比对结果,使用Neighbor-Joining、最大似然法(如PhyML)、贝叶斯推断(如MrBayes)等方法构建进化树,揭示基因家族成员之间的进化关系。
- 进化树的解读:可以帮助了解基因家族的起源、分化和功能演化。
4. 功能注释
- 同源推断:基于已知功能的基因家族成员,对新识别的成员进行功能预测。
- 生物信息学工具:利用InterPro、Pfam等数据库进行蛋白质结构域分析,以推测基因产品的可能功能。
- 表达分析:研究基因表达数据(如RNA-Seq数据),了解基因家族成员在不同组织、发育阶段或应对不同环境条件时的表达模式,以推测功能。
5. 比较基因组学
- 基因组环境分析:研究基因家族成员在不同物种中的位置和上下游基因,以理解基因家族的进化和功能分化。
- 物种特异性:分析基因家族在不同物种中的扩张或缩减,探索这些变化与物种特有生态位或生物学特性的关系。
这个分析流程涉及到多种生物信息学工具和数据库,对于不同的研究目标和数据类型,可能需要调整分析策略或工具选择。此外,随着生物信息学领域的不断发展,新的工具和方法也在不断出现,可以提供更高效、准确的分析结果。
富集分析结果不一样一事情
GO (Gene Ontology) 和 KEGG (Kyoto Encyclopedia of Genes and Genomes) 富集分析是生物信息学中常用的方法,用以识别基因列表中显著富集的生物学过程、细胞组分和分子功能(GO分析),以及相关的代谢路径或疾病途径(KEGG分析)。不同平台或工具之间分析结果的差异可能由多种因素引起,包括但不限于:
基因集的更新:GO和KEGG数据库都会定期更新,以反映最新的科学发现。不同工具或服务器可能使用的数据库版本不同,从而影响分析结果。
注释差异:基因的注释(如基因符号、ENSEMBL ID等)可能会有所不同,这取决于使用的数据库版本或注释来源。基因标识符的映射错误或不完整都可能导致分析结果的差异。
统计方法的差异:富集分析涉及统计方法,比如校正p值以控制假发现率(FDR)。不同工具或平台可能使用不同的统计模型、校正方法或参数设置,这些都会影响最终的富集结果。
用户输入数据的处理:输入数据的预处理方式(如过滤、标准化等)可能在不同的工具或平台间有所不同,这也可能导致结果的差异。
算法实现的差异:即使是相同的统计方法,不同的软件包或平台可能会有不同的算法实现,特别是在处理边缘情况或优化计算性能时。
分析参数的差异:用户在不同平台上可能使用了不同的参数设置,如富集分析的显著性阈值、基因集大小的限制等。
为了减少这些差异,你可以尝试以下策略:
- 确保使用最新的数据库版本,并在可能的情况下,在不同平台间保持一致。
- 核对输入的基因列表,确保基因标识符的准确性和一致性。
- 注意参数设置,尽量在不同的工具或平台上使用相同的参数。
- 比较和理解不同工具的统计方法和算法实现,以更好地解释结果的差异。
了解这些差异的来源有助于你更准确地解释和比较不同工具或平台的富集分析结果。
要在您的Shiny应用程序中添加一个可以让用户直接复制和粘贴内容的输入方式,您可以使用文本框(textareaInput)作为输入界面。用户可以将其数据直接复制并粘贴到这个文本框中,然后您的应用程序可以读取这些输入的数据并进行处理。
下面是如何在您的UI函数中添加textareaInput的示例代码段,以及在服务器逻辑中处理这种输入方式的基本方法。请注意,这需要您根据实际情况对数据的解析和处理逻辑进行一些调整:
UI部分
在sidebarPanel中添加一个textareaInput组件,让用户可以粘贴数据:
1 | textareaInput("textData", "Paste data here:", rows = 10, placeholder = "Paste your data here. Each row should be a gene with its corresponding values, separated by commas or tabs."), |
服务器逻辑部分
在服务器函数中,添加一个响应式表达式来处理从textareaInput接收的文本数据。您需要根据实际粘贴的数据格式(例如,使用逗号、空格或制表符作为分隔符)来解析这些数据:
1 | textData <- reactive({ |
确保您的应用程序的其余部分能够适当地处理从textData这个响应式表达式中获取的数据,这可能需要您根据输入数据的格式(例如,列名)进行一些调整。此外,您可能需要在UI中添加一些说明,指导用户如何正确格式化他们粘贴的数据(例如,需要哪些列,以及如何分隔它们)。
通过添加textareaInput,用户就可以选择直接上传文件,或者复制和粘贴数据,提供了一个更灵活的数据输入方式。
孟 婕师姐论文,基因家族,,,鸡NLRs基因家族鉴定、系统进化及表达分析
鸡 NLRs基因家族 鉴定、系统进化 及 表达分析
摘 要:为深入了解鸡 NOD 样受体(NLRs)家族基因特性,
利用生物信息学方法:
通过保守结构域搜索鉴定了 鸡的NLRs基因, 分析NLRs基因 理化性质、基因结构、系统进化关系、正选择作用、共线性关系和表达情况。
结果显示:1,共鉴定出5个鸡NLRs基因,分别命名为chNLRC5、chNLRC3、chNLRP3、chNLRX1、chNOD1,
2,分布在 5 条染色体上,编码 7341 862 个氨基酸,编码蛋白相对分子质量为 82 842.01207 969.52 ku,等电点为 5.48~8.64;
3,chNLRC5、chNOD1 和chNLRP3 为酸性蛋白,
4,chNLRC3 和 chNLRX1 蛋白呈碱性,只有 chNOD1 为稳定蛋白,
5,5 个成员均为亲水蛋白,5个基因结构差别显著,分别含有 48、20、7、7、11个外显子;
6,系统进化树显示,:
NLRs 基因家族分为 5 个分支,鸡、绿头鸭、火鸡和日本鹌鹑 NLR 基因间存在较强的共线性关系;
7,对各成员进行选择压力分析结果鉴定出 13个正选择位点;鸡回肠和肺组织中检测到 NLR基因表达。
研究表明正选择效应在 NLRX1、NOD1、NLRC5 基因作用明显,NLRC5 在鸡回肠和肺组织中为表达活跃的功能基因.
关键词:鸡;NLRs基因家族;进化分析;基因表达
NOD 样受体(Nucleotide-binding oligomeriza⁃tion domain like receptors,NLRs)
是一类细胞质内模 式 识 别 受 体(Pattern recognition receptors,PRRs),可与病原相关分子模式(Pathogen-associ⁃
ated molecular patterns,PAMPs)和损伤相关分子模 式 (Damage associated molecular patterns,DAMPs)直接结合,能够启动机体的免疫应答,连
接固有免疫和适应性免疫的关键环节[1~4]。
NLRs主要由一个 C 末端 LRR 基序、一个负责 ATP 依赖的寡聚的中心 NATCH 核苷酸结合结构域和一个N 末端效应结构域组成,根据 N 端结构域可将其
分为 NLRA、NLRB、NLRC、NLRP 和 NLRX 五个亚家族[5,6]
。目前,已经发现人类含有23种NLRs,小鼠中至少有34种.
目前关于鸡 NLRs 基因家族系统的分析尚未见报道[19,20]。
本研究通过生物信息学方法鉴定出鸡NLRs所有成员,并通过对其基因结构、性质、功能和进化进行分析,
利用鸡感染 H5N1 亚型禽流感病毒、H5N2 亚型禽流感病毒的转录组数据比较了 NLRs 家族基因表达特征,为进一步研究鸡NLRs基因家族成员的生物学功能提供依据.
材料与方法
1.1 NLRs基因家族的全基因组鉴定及蛋白特性分析
鸡(Gallus gallus)基 因 组(Gallus_gallus.GRCg6a.dna_sm.toplevel.fa.gz)及 注 释 数 据(Gal⁃lus_gallus.GRCg6a.99.gff3.gz)来自 Ensembl数据库
(https://asia.ensembl.org/index.html)。在Pfam(http://pfam.xfam.org/)中查找获得 NLR 蛋白特征性结构域。
利用 TBtools 软件[21]通过保守结构域查找鸡中的 NLR家族可能成员。然后通过 CDD(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)
检测是否含有 NLR特征性序列,删除不含特征性结构域的蛋白序列,最终得到鸡 NLR 家族成员。
利 用 在 线 软 件 ProtParam(https://web.expasy.org/protparam/)预测NLR蛋白的理化性质。
1.2 鸡NLRs基因序列结构分析
NLR的外显子和内含子位置信息参考鸡基因组注释信息 gff3 文件,用 TBtools 软件提取其中信息 。
使 用 MEME[22]软 件(http://meme- suite.org/meme_5.2.0/tools/meme)预测 NLR 基因的基序,最大基序数设置为 10。最后用 TBtools 软件对结果
进行可视化分析。
1.3 NLR基因的系统进化分析
从 NCBI 数据库获取绿头鸭(Anas platyrhyn⁃chos)、人(Homo sapiens)、小鼠(Mus musculus)、斑
马鱼(Danio rerio)、非洲爪蟾蜍(Xenopus tropica⁃lis)、锦 龟(Chrysemys picta bellii)的 NLR 蛋 白 序
列[23],
使用 MEGAX[24]对上述 NLR 蛋白家族序列进行多重比对,使用Neighbor-Joining方法构建系统发生树,自展值(Bootstrap)设定为 1 000,其他参数默认
1.4 鸟类NLR基因正选择作用分析
除鸡的NLR基因序列外,本研究从NCBI数据库选取了54条NLR基因序列,以人NLR基因序列为外群构建进化树(见表1)。用最大似然法构建NLR 基因进化树,利用 EasyCodeML 软件[25]分析NLR 基因的选择压力。用 ω(ω=dN/dS)值表示基因受到何种选择压力,选用位点模型(Site Mod⁃el),假设系统发育树的不同分支受到的选择压力相同,但是不同的氨基酸位点受到的选择压力不同。
选取 3 组位点特异模型进行比较:M0(单一比率)与 M3(离散)、M1a(近中性)与 M2a(正选择)、M7(beta)与M8(beta & ω)。为了提高结果准确性,用 datamonkey 网站(http://www.datamonkey.org/analyses)的 FUBAR、MEME、FEL和 SLAC进行验证预测的正选择位点,有两种及两种以上方法预测到的位点,认为是正选择位点[26]。
1.5 NLR直系和旁系同源基因分析
鸡、绿头鸭、火鸡(Meleagris gallopavo)和日本鹌鹑(Coturnix japonica)的直系与旁系同源关系通
过 TBtools 软件的 One Step MCScanX- Super Fast
功能界面依次鉴别,并用 Advanced Circos 进行可视化分析。
1.6 鸡NLRs基因的表达分析
为了比较感染病毒前后不同组织中 NLR 基
因的表达差异,从 NCBI 的 SRA 数据库获得白来航鸡感染 H5N1亚型禽流感病毒、H5N2亚型禽流感病毒不同时期回肠和肺的转录组(ERR597318-
ERR597353)注释文件[27],运用 TBtools 软件检测质量,去除接头和低质量序列,最后拼接组装进行表达量分析。得到鸡各基因的 FPKM 值后,计算
log(2 FPKM+1)值,通过TBtools软件对鸡各基因表达情况绘制热图。
2 结果与分析
2.1 鸡NLRs的鉴定
共鉴定出5个鸡NLR成员,根据其结构域特征
分别命名为chNLRC5、chNLRC3、chNOD1、chNLRP3
和 chNLRX1(见表 1)。鸡 NLR 家族成员的蛋白
序列长度在 734~1 862 aa 之间;分子量从最小
82 842.01 ku(chNLRP3)到 最 大 207 969.52 ku
(chNLRC5)不等;等电点介于 5.48(chNLRP3)至
8.64(chNLRX1)之间。pI主要取决于氨基酸中酸
性氨基酸和碱性氨基酸的数量差,chNLRC5、
chNOD1 和 chNLRP3 蛋白呈酸性(等电点小于
7.0),chNLRC3 和 chNLRX1 蛋白呈碱性(等电点
大于7.0)。chNOD1蛋白不稳定系数小于40,说明
其是稳定蛋白;chNLRC5、chNLRC3、chNLRP3 和
chNLRX1 蛋白不稳定系数大于 40,为不稳定蛋
白。亲水性平均系数均为负数,说明 chNLR 蛋白
均为亲水性蛋白质。
不想看了,明天看。
师姐的 鸡 GATA 基因家族鉴定及生物信息学分析
摘要:为了解鸡GATA(ChGATA)家族基因特性
利用生物信息学方法
对ChGATA基因家族成员进行鉴定,分析其理化性质、染色体定位、基因结构、保守基序、进化关系、蛋白质结构及蛋白质互作关系。
结果表明,共鉴定出 6 个 ChGATA 基因,不均匀地分布在 5 条染色体上。
根据蛋白质保守结构域命名为ChGATA2—ChGATA6 和 ChTRPS1。
理化性质分析 结果显示,ChGATA 均为无稳定的亲水蛋白质,且除ChTRPS1外均为碱性蛋白质。
二级结构预测分析 结果显示,ChGATA主要由无规则卷曲组成。保守结构域分析结果显示,ChGATA均包含ZnF_GATA结构域。系统进化分析结果显示,
ChGATA基因家族可
分为 3个亚群,且同一亚群的基因结构相似、蛋白质三级结构相似、相互作用蛋白质较一致、亲缘关系较近
为提高经济效益,家禽养殖中非常关注肌肉的
快速生长,却忽视了心脏、肝脏等内脏器官发育缓
慢的问题,这可能导致家禽免疫系统发育不良[1‐3]
。
目前,关于禽类体质量增长及肌肉发育方面的研究
较多,但是对于内脏器官的发育研究较少[4‐5]
。研究
禽类内脏器官发育调控基因对家禽的健康养殖具
有重要意义。GATA是广泛存在于真核生物中的含
锌指结构的转录因子家族,在细胞增殖和器官形成
中起重要作用,其可与靶基因启动子的A/T(GATA)
A/G序列特异性结合[6]
鸡是禽类性状形成分子机制、基因功能解析、
基因组学与分子遗传学研究中的关键物种[15]
。迄今
为止,尚未见有鸡GATA(ChGATA)基因家族基本特
性的研究报道。为此,从全基因组水平对 ChGATA
基因家族进行鉴定,对其进行理化性质、结构和进
化、蛋白质互作等方面的分析,为研究 ChGATA 基
因家族生物学功能奠定基础
1 材料和方法
- 1 ChGATA基因家族成员的检索及理化性质
分析
从 Ensembl(https://asia. ensembl. org/index.html)数据库中下载鸡基因组序列和基因组结构注释文件,
利用 NCBI(https://www.ncbi.nlm.nih.gov/)BLAST,搜索已报道的 GATA 蛋白序列并与 Pfam 数据库比对,获得 GATA 基因家族的 Pfam ID(PF00320)。
用TBtools 软件[16]BLAST 搜索 ChGATA 基因家族的同源蛋白质,采用 NCBI Batch 工具的 CD-Search 程序进一步验证,
剔除冗余蛋白质序列,得到 ChGATA基因家族序列 ,
并将其定位到染色体上。
采用ProtParam 程序(https://web.expasy.org/protparam/)在线预测 ChGATA 基因家族成员的理化性质(氨基酸数目、分子质量、理论等电点、疏水性、不稳定性)
- 2 ChGATA基因家族保守结构域分析
利用 SMART 分析 ChGATA 基因家族的保守结构域,并用TBtools对结果进行可视化分析。
- 3 ChGATA 基 因 家 族 成 员 基 因 结 构 和 Motif
分析
利用 MEGA-X 软件[17]分析 ChGATA 基因家族成员进化关系;用TBtools从基因组结构注释文件中提取对应基因结构信息,预测 ChGATA 基因家族的内含子和外显子组成;
用 MEME 在线软件(https://meme‐suite.org/meme/tools/meme)对保守 Motif 进行分析,搜索Motif值设定为10,并用TBtools软件可视化分析
4 ChGATA基因家族系统进化树构建
利用 MEGA-X 软件中的 Clustal W 程序对 11个物种的 GATA 蛋白序列进行多序列比对分析,分析结果用邻接法(Neighbor‐Joining,NJ)构建系统进化树,Bootstrap 值设定为 1 000 次,其他参数默认不
变[18]
。构建的系统进化树用 iTOL 在线程序(https://itol.embl.de/)[19]修饰5 ChGATA基因家族的蛋白质结构预测
蛋白质二级结构用 SOPMA 在线程序(https://npsa‐prabi. ibcp. fr/cgi‐bin/npsa_automat. pl? page=npsa_sopma.html)预测。将鉴定出的 ChGATA 蛋白序 列 逐 一 提 交 SWISS-MODEL 网 站(https://wissmodel.expasy.org/),预测蛋白质三级结构。6 ChGATA的蛋白质互作分析
利用 STRING 在线平台[20(] https://string‐db.org/)构建蛋白质互作网络,分析 ChGATA 蛋白与其他蛋白质的相互作用。蛋白质来源设置为鸡(Gallus_gallus),其他参数默认
2 结果与分析
2. 1 ChGATA基因家族成员的鉴定及其在染色体
上的分布
共鉴定到 6 个 ChGATA 基因,根据其蛋白质保守结构域分别命名为ChGATA2、ChGATA3、ChGATA4、
动物的基因家族的生信分析,我想学习一下,但是很多知识点,我不清楚,你能告诉我哪些知识点我应该知道?
在学习动物的基因家族的生物信息学分析时,有几个关键知识点是必须掌握的。通过分析文档【3†source】中关于鸡GATA基因家族的生物信息学分析,我们可以总结出以下几个重要的知识点,这些知识点不仅适用于鸡GATA基因家族的分析,也适用于其他动物基因家族的生物信息学研究:
基因家族成员的鉴定:利用生物信息学方法鉴定基因家族成员,包括使用Ensembl数据库下载基因组序列和基因组结构注释文件,以及使用NCBI BLAST搜索和Pfam数据库比对来获得特定基因家族的成员。
基因和蛋白质的理化性质分析:包括分子质量、等电点、疏水性、不稳定性等参数的预测,以及基因的染色体定位。
保守结构域分析:利用SMART等工具分析基因或蛋白质的保守结构域,这对理解其功能和分类有重要意义。
基因结构分析:包括外显子和内含子的组成及其排列顺序,这有助于理解基因的功能和调控机制。
进化关系分析:构建系统进化树,分析基因家族成员之间以及与其他物种基因家族成员之间的进化关系,通常使用MEGA-X、Clustal W等软件。
蛋白质结构预测:包括二级和三级结构的预测,了解蛋白质的结构特征,为进一步的功能研究提供基础。
蛋白质互作分析:使用STRING等在线平台构建蛋白质相互作用网络,分析基因家族成员如何通过与其他蛋白质的相互作用参与生物过程。
掌握上述知识点是进行动物基因家族生物信息学分析的基础。此外,熟悉相关的生物信息学工具和数据库也非常重要,包括但不限于Ensembl、NCBI、Pfam、SMART、MEGA-X、Clustal W、ProtParam、MEME、TBtools、SOPMA、SWISS-MODEL和STRING等。通过系统学习和实践这些知识点和工具,你将能够有效地进行动物基因家族的生物信息学分析。
鸡TLR1A基因2个SNP位点功能分析及与坝上长尾鸡沙门氏菌感染抗性的相关分析
为探究鸡TLR1A基因g.926G>A和g.1076G>C 2个nsSNP位点多态性及其可能的功能意义,g.926G>A和g.1076G>C 2个nsSNP位点多态性,什么意思?
TLR1A基因中的g.926G>A和g.1076G>C两个nsSNP位点多态性,这里的描述涉及到几个基因学和分子生物学的概念,我来依次解释一下:
基因多态性(Gene Polymorphism):这是指基因的同一位置上,在不同个体中可以存在两种或多种序列变异。基因多态性是种群遗传多样性的重要来源,它能够影响个体对环境的适应能力、疾病易感性等。
nsSNP(Nonsynonymous Single Nucleotide Polymorphism):nsSNP是指在蛋白编码区的单核苷酸多态性,这种多态性会导致编码的氨基酸发生改变,从而可能影响蛋白质的结构和功能。简单来说,就是基因序列中的一个碱基发生变化,这种变化导致了蛋白质序列中相应位置的氨基酸的改变。
TLR1A基因:TLR1A是指Toll样受体1A基因,属于Toll样受体家族。Toll样受体是先天免疫系统中识别病原体的重要分子,它们能够识别特定的病原体相关分子模式(PAMPs),从而激活免疫应答。TLR1A基因在不同物种中都有发现,其表达的蛋白质参与了对病原体的识别和免疫应答的启动。
g.926G>A和g.1076G>C:这里指的是在TLR1A基因中,分别在基因序列的第926位和第1076位上,G(鸟嘌呤)到A(腺嘌呤)和G到C(胞嘧啶)的单核苷酸变异。这种变异属于nsSNP,因为它们位于编码区,并且这种替换可能导致编码的氨基酸发生改变。
探究这两个位点的多态性及其功能意义,可能涉及到研究这些变异如何影响TLR1A蛋白的结构和功能,从而进一步影响鸡的免疫响应。这类研究有助于理解遗传变异如何影响生物的疾病抵抗力和免疫能力,对于遗传育种、疾病预防和治疗等方面具有重要意义。
试验采用CRS-PCR-RFLP方法检测310只坝上长尾鸡(阳性127只,阴性183只)TLR1A基因2个nsSNP位点的多态性,这个方法是什么?CRS-PCR-RFLP
CRS-PCR-RFLP方法是一种结合了PCR(聚合酶链反应)和RFLP(限制片段长度多态性分析)的分子生物学技术,用于检测基因序列中的特定变异。具体到CRS-PCR-RFLP,”CRS”可能指的是某种特定的实验设计或优化的PCR步骤,但在广泛的科学文献中,CRS的具体含义可能因上下文而异。下面是这一方法各部分技术的基本原理:
PCR(Polymerase Chain Reaction,聚合酶链反应)
- PCR是一种常用的DNA扩增技术,可以快速地将极少量的DNA样本扩增成足够进行分析的数量。通过设计特异性的引物对目标DNA序列进行特定的扩增。
RFLP(Restriction Fragment Length Polymorphism,限制片段长度多态性分析)
- RFLP是一种基于限制性内切酶识别和切割特定DNA序列的技术。不同个体间的基因序列差异可能导致限制酶切割位点的存在或缺失,从而产生长度不同的DNA片段。通过电泳分析这些片段,可以检测基因中的多态性。
CRS-PCR-RFLP
- 结合PCR和RFLP技术,首先使用PCR特异性扩增感兴趣的基因区域,然后利用RFLP技术对扩增的产物进行切割,以检测基因的多态性。这种方法特别适合于检测特定位点的单核苷酸多态性(SNPs)。
- 在你提到的研究中,CRS-PCR-RFLP方法被用于检测TLR1A基因中两个nsSNP位点的多态性。通过这种方法,研究者能够区分出含有不同基因型的个体,即可以识别出特定位点上是G还是A,G还是C的变异。
- 这种方法的优势在于其高度的特异性和灵敏度,能够准确地识别和区分基因序列中的微小变异,对于遗传学研究和基因型分析非常有用。
“CRS”在某些文献中可能指特定的实验设计或变体,如特定的引物设计、扩增条件等,但在没有更多上下文的情况下,它的确切含义可能需要查阅具体的研究资料或实验协议来确定。
g.926G>A(S309N)和g.1076G>C(R359P)与鸡沙门氏菌易感性显著相关(P<0.05)。其中S309N在种间种内水平均受到正选择压力;R359P在种内受到正选择压力
其中S309N在种间种内水平均受到正选择压力;R359P在种内受到正选择压力是什么意思
这里所描述的“正选择压力”是进化生物学中的一个概念,指的是环境中的某种压力(例如,疾病、食物供应、气候变化等)促使某些遗传变异在种群中被优先保留和积累的现象。当一个遗传变异提供了某种生存或繁殖上的优势时,拥有这种变异的个体更可能生存下来并传递其基因给后代,导致这种变异在后代中变得更加普遍。这是自然选择的一种形式。
在你提到的研究中:
S309N (g.926G>A) 和 R359P (g.1076G>C) 指的是TLR1A基因中的两种特定的非同义单核苷酸多态性(nsSNP),这些变异导致蛋白质序列中第309个氨基酸由丝氨酸(S)变为天冬酰胺(N),以及第359个氨基酸由精氨酸(R)变为脯氨酸(P)。
种间正选择压力:S309N变异在不同物种之间(种间)受到正选择压力,意味着这个变异在进化过程中在多个物种中被优选保留,可能因为它在不同物种的环境中都提供了某种生存或繁殖上的优势。
种内正选择压力:S309N和R359P变异在种内(即在同一物种内的不同个体之间)受到正选择压力,意味着这些变异在特定物种的环境和生存条件下提供了优势,使得携带这些变异的个体更可能生存并繁衍后代。
这项研究发现,g.926G>A(S309N)和g.1076G>C(R359P)这两个变异与鸡对沙门氏菌的易感性显著相关,这意味着这些变异可能影响鸡对沙门氏菌感染的抵抗力。由于S309N和R359P变异受到正选择压力,这表明在鸡的进化历史中,这些变异可能帮助鸡更好地抵抗沙门氏菌或其他类型的病原体,从而在种群中被优选保留。
这个表格是使用逻辑回归方法分析了TLR1A基因在两个位点(g.926G>A和g.1076G>C)的基因型与沙门氏菌易感性之间的相关性。逻辑回归是一种统计方法,用于分析一个或多个自变量与一个二元结果变量(例如,疾病的有无)之间的关系。在这个案例中,二元结果是指是否对沙门氏菌易感(阳性或阴性)。表中列出了不同基因型与沙门氏菌易感性之间的关联强度,使用了如下几个统计指标:
- 基因型: 这一列展示了不同的基因型组合。
- 阳性/阴性: 分别表示测试结果为阳性(沙门氏菌易感)和阴性(不易感)的个体数量和百分比。
- OR (Odds Ratio, 比值比): 表示在给定条件下,事件发生的几率比。OR>1表示增加风险,OR<1表示减少风险。
- 95% CI (95% Confidence Interval, 95%置信区间): 表示OR的估计范围,如果不包含1,则认为统计显著。
- P值: 表示结果的统计显著性。P<0.05通常被认为是统计显著的,表示基因型和疾病易感性之间存在显著相关性。
- AIC (Akaike Information Criterion) 和 BIC (Bayesian Information Criterion): 是衡量模型拟合优度的指标,值越小表示模型越好。
表格中还分别采用了几种不同的遗传模型来分析数据:
- Codominant (共显性模型): 每个等位基因的影响都被独立考虑。
- Dominant (显性模型): 至少有一个特定等位基因就足以产生影响。
- Recessive (隐性模型): 需要两个特定的等位基因才能产生影响。
- Overdominant (超显性模型): 杂合子有不同的表型与两种纯合子表型。
- Log-additive (对数加性模型): 假设等位基因的效果是加性的。
从这个表格中,我们可以看出:
- 对于926位点,显性模型(G/G vs. G/A-A/A)和隐性模型(G/G-G/A vs. A/A)在统计上显著,说明这个位点的基因型与沙门氏菌的易感性有显著关联。
- 对于1076位点,共显性模型(G/G vs. G/C vs. C/C)、超显性模型(G/G-C/C vs. G/C)显示统计显著性,意味着这个位点的基因型也与沙门氏菌易感性显著相关。
简而言之,这个表格展示了TLR1A基因的两个位点的不同基因型与鸡对沙门氏菌易感性之间的显著相关性,这种相关性通过不同的遗传模型来展现。
这个表格展示了对ChTLR1A(鸡TLR1A基因)进行的正选择位点预测结果。正选择位点是指在进化过程中受到正选择压力,从而在物种中被优先保留的遗传位点。正选择通常表明这些位点的变异可能对物种有生存或繁殖优势。
在表格中,列出了一系列的氨基酸位点(例如,2G、70S、131V等),这些位点代表了蛋白质中特定位置的氨基酸残基。每个位点旁边的Δ标志表示该位点在对应的分析方法中被预测为正选择位点。
表中所用的分析方法包括:
- M8: 一种统计模型,用于检测基因序列中的正选择信号。
- FEL (Fixed Effects Likelihood): 一种计算特定位点上非同义(改变氨基酸)和同义(不改变氨基酸)替换比率的方法,用于识别受正或负选择影响的位点。
- FUBAR (Fast, Unconstrained Bayesian AppRoximation): 一种贝叶斯方法,用于快速鉴定受正选择、负选择或中性进化影响的位点。
- MEME (Mixed Effects Model of Evolution): 用于识别受正选择影响的位点,尤其是那些只在部分分支上受正选择的位点。
- SLAC (Single-Likelihood Ancestor Counting): 一种基于最大似然原理的方法,用于识别受正或负选择影响的位点。
表中还规定,只有当一个位点被三种及以上的方法预测为正选择位点时,才被认定为正选择位点,并用加粗字体表示。例如,334Q和359R位点被所有五种方法认定为正选择位点,显示这些位点在鸡TLR1A蛋白的进化中可能具有重要的生物学功能和适应性意义。
简而言之,这个表格通过不同的生物信息学方法,确定了在鸡TLR1A蛋白中哪些位点在进化过程中可能受到了正选择压力,这些位点可能对鸡种的生存和繁衍具有重要作用。
梦婕师姐大论文(D:/)鸡TLR、NLR和RLR正选择多态位点与沙门氏菌抗性的初步研究
这个题目涉及到动物免疫学和微生物学的一个研究领域。首先,我们来分解一下题目中的主要元素和它们之间的关系。
鸡:这里指的是研究的对象,即实验动物。鸡作为一种经济动物,在研究宿主对病原体的免疫反应方面具有重要价值。
TLR、NLR和RLR:这些是指三大类模式识别受体(Pattern Recognition Receptors, PRRs),它们是免疫系统识别病原体并启动免疫反应的关键蛋白。
- TLR(Toll样受体):广泛存在于动物细胞表面或内部,能识别病原体的共同结构(如细菌的脂多糖),并激活免疫反应。
- NLR(NOD样受体):主要在细胞内发挥作用,识别细胞内病原体的成分,参与炎症反应和细胞死亡的调控。
- RLR(RIG-I样受体):主要识别病毒RNA,是重要的抗病毒免疫应答调节器。
正选择多态位点:这里指的是通过正向选择(positive selection)作用下形成的遗传多态性。正选择通常意味着这些多态位点在进化过程中对于生存有利,可能增强了宿主对某些病原体的抵抗力。
沙门氏菌抗性:沙门氏菌(Salmonella)是一种常见的致病菌,可以引起食物中毒等疾病。抗性指的是宿主对沙门氏菌感染的抵抗力,这种抵抗力可以是先天的,也可以是后天获得的。
综合以上信息,这个题目的研究焦点是探索鸡中的三种模式识别受体(TLR、NLR和RLR)的遗传多态性如何影响它们对沙门氏菌的抵抗能力。具体来说,研究可能会分析这些受体的基因在不同鸡只中的变异,并研究这些变异是否与对沙门氏菌的抗性有关。这项研究对于理解动物免疫系统如何应对细菌感染具有重要意义,同时也可能为培育抗病原体能力更强的家禽品种提供遗传标记。
摘要:
TLR、NLR和RLR是先天免疫识别受体PRRs的三大免疫模式识别受体基因。
先天免疫是机体免疫防御的第一道防线,其模式识别受体PRRs直接与病原相互作用,识别各类病原。
目前,对禽类TLR亚家族的研究较为全面,然而,禽类的整个PRRs基因家族的研究仍然较少。
本研究以TLR、NLR和RLR家族基因为候选基因,在多层面筛选抵抗疾病感染的基因位点;
以地方鸡为素材进行基因分型,在抗性和易感群体中的比较基因型分布差异,筛选对沙门氏菌感染有显著遗传效应的分子遗传标记,为培育抗病品种,推动养殖业发展提供重要的理论依据。主要结果如下:
1、基于NCBI基因组数据在TLR、NLR和RLR三个亚家族中共检测到159个正选择位点。TLRs正选择位点占PRRs整体的67.9%,其中有81%的位点分布在胞外域,说明TLRs基因的进化速率大于NLRs和RLRs,且胞外域进化速率快于其他部位。
2、基于ChickenSD群体重测序数据在TLR、NLR和RLR三个亚家族中共检测到88个正选择位点。值得注意的是,原鸡群体中TLR1A基因遗传多样性最低,驯化群体中TLR2A基因遗传多样性最低;TLR3基因在原鸡及驯化鸡群遗传多样性最高。
3、沙门氏菌感染后表达数据分析显示,盲肠扁桃体组织中,TLR1A、TLR1B、TLR2A、TLR3、TLR4、TLR5、TLR7、TLR15和NLRC3在抵抗型个体中的表达明显高于易感型个体;脾脏组织中,TLR1A和TLR4在抗性群体中的表达量高于易感群体。这说明其参与了沙门氏菌免疫应答响应过程。
4、对TLR1A和TLR4的4个nsSNPs位点与沙门氏菌易感性的关联分析显示,TLR1A基因1162A>T变异位点与沙门氏菌易感性相关性不显著(P>0.05); 2443T>C位点,TT型可能为沙门氏菌抵抗型;TLR4基因1147T>C位点,CC型对TT型可能为沙门氏菌易感型;1832A>G变异位点与沙门氏菌易感性相关性不显著(P>0.05)。
Galbase https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-022-08598-2
我们收集并纳入了四个组蛋白修饰标记(H3K4me3、H3K27ac、H3K4me1 和 H3K27me3)和一个转录因子,即基于 ChIP-seq 的 CCCTC 结合因子 (CTCF),以及一个基于 ATAC-seq 的开放染色质标记,以鉴定顺式调节元件。共鉴定出488,583个顺式调控元件,占全基因组大小的49.37%
什么意思?解释一下
ChatGPT
这段话是在描述一项关于基因组学的研究。研究人员使用了几种分子生物学技术来鉴定基因调控区域,这里简要解释一下各个部分的意义:
蛋白质修饰标记: 这些是组蛋白(一种蛋白质,DNA会围绕它们卷曲形成染色质结构)的特定修饰,它们对基因表达有调控作用。
H3K4me3: 这是组蛋白H3的第4个赖氨酸残基三甲基化的标记,通常与基因启动区域有关,表示基因活跃表达。
H3K27ac: 这是H3组蛋白第27个赖氨酸残基乙酰化的标记,通常标志着基因表达调控区域。
H3K4me1: 这是H3的第4个赖氨酸单甲基化的标记,与增强子(一类调控元件)有关。
H3K27me3: 这是H3的第27个赖氨酸三甲基化的标记,通常与基因沉默有关。
转录因子CTCF: CTCF是一种重要的转录因子,它在基因表达调控和染色质结构形成中发挥作用。
ChIP-seq技术: 这是一种确定蛋白质与DNA互动位置的实验方法,可以用来鉴定转录因子和组蛋白修饰在基因组中的具体结合位点。
ATAC-seq技术: 用于鉴定基因组中的开放染色质区域,即易于转录因子和其他基因调控蛋白质结合的区域。
顺式调控元件: 这些是指在同一分子DNA上调控基因表达的序列元件,比如增强子和启动子。
综上所述,研究者收集了四种组蛋白修饰和一个转录因子CTCF的数据,以及ATAC-seq鉴定的开放染色质区域,以识别可能调控基因表达的基因组区域。他们一共鉴定了488,583个这样的调控元件,这些元件占据了基因组大约49.37%的区域。这项工作帮助了解基因如何被调控,以及基因表达模式的形成机制。
当然,让我们更深入地探讨这项研究的内容和意义。
在基因组学中,研究者不仅对基因本身感兴趣,还对控制基因何时以及如何开启或关闭的机制感兴趣。这种控制主要是通过DNA内的各种类型的调控元素来实施的,这些元素可以比作是激活或关闭基因表达的开关。
组蛋白修饰:组蛋白是DNA缠绕的蛋白质。当这些蛋白质发生化学修饰(例如,通过甲基化或乙酰化)时,它们可以影响DNA绕组蛋白的紧密程度。不同的组蛋白修饰模式可以导致基因的激活或抑制。所提到的修饰——H3K4me3、H3K27ac、H3K4me1 和 H3K27me3 ——与激活或抑制基因表达有关。
转录因子CTCF:CTCF是一种结合到特定DNA序列的蛋白质,并在组织基因组的3D结构中发挥作用,从而影响某些基因的转录可及性(即从DNA制造RNA的过程)。
ChIP-seq 和 ATAC-seq:这些技术分别用于分析蛋白质-DNA相互作用和染色质可及性。ChIP-seq(染色质免疫沉淀测序)使研究者能够识别整个基因组中与DNA相关蛋白质(如组蛋白或转录因子)的结合位点。ATAC-seq(使用测序的可及染色质转座酶检测)用于寻找开放的染色质区域,其中DNA没有紧密缠绕在组蛋白周围,表明这些区域更可能在基因调控中活跃。
顺式调控元件:这些是位于与其调控的基因相同染色体上的DNA片段。它们可以包括启动子、增强子、沉默子和隔离子,并且对于基因表达的精确控制至关重要。
在这项研究中,研究者收集了四种组蛋白修饰和CTCF蛋白的结合模式数据,以及通过ATAC-seq识别的开放染色质区域的数据。他们使用这些数据来识别可能作为开关来开启或关闭基因的潜在顺式调控元件。
结果是识别了488,583个顺式调控元件。这些元件构成了基因组的一个重要部分(49.37%),突出了调控网络在控制基因表达中的广泛性。
理解这些元素及其在基因组中的分布对于揭示细胞功能、发育和对环境因素响应的背后基因表达复杂协调是至关重要的。这些知识还可以为由基因表达失调引起的疾病的治疗策略提供信息。



