rain插件的位置
F:\Users\user\Documents\WeChat Files\wxid_kztyihy9nr5g22\FileStorage\File\2023-03\插件
F:\Users\user\Documents\WeChat Files\wxid_kztyihy9nr5g22\FileStorage\File\2023-03\插件
CSDN
https://blog.csdn.net/Mrrunsen/article/details/126623440
第十三章 节律基因的挖掘与分析
13.1简介 2
13.2 Rain插件的安装与使用 3
13.3实例:基于鸡松果体转录组数据节律基因的挖掘与分析 3
13.3.1 3
13.3.2 3
13.3.3 3
13.3.4 3
13.3.5 3
13.4 3
13.5 总结 3
13.6本章练习题 3
参考文献 3
第十三章 节律基因的挖掘与分析
13.1简介 2
13.2 Rain插件的安装与使用 3
13.3实例:基于鸡松果体转录组数据节律基因的挖掘与分析 3
13.3.1 3
13.3.2 3
13.3.3 3
13.3.4 3
13.3.5 3
13.4 3
13.5 总结 3
13.6本章练习题 3
参考文献 3
写作专题《节律基因的挖掘与分析》是一个具有挑战性和前沿性的任务,特别是当涉及到使用特定的分析工具如Rain插件,以及处理具体案例如基于鸡松果体转录组数据的分析时。以下是一些建议来帮助你构思和组织你的章节:
13.1 简介
背景介绍草稿**背景介绍**:简要介绍节律基因的重要性,以及它们如何影响生物体的生理和行为过程。
节律基因在生物体内扮演着至关重要的角色,它们是生物钟系统的基础组成部分,控制和调节着生物体的生理和行为节律。生物节律,特别是昼夜节律(circadian rhythms),对于保持生物体内部时间与地球自转周期同步至关重要,影响着睡眠-觉醒周期、饮食习惯、激素水平、体温调节以及许多其他生命活动。
节律基因通过编码调控蛋白来直接或间接地控制这些生理过程,这些蛋白质参与反馈回路,形成一个精确的内部时钟系统。例如,人类的主要节律基因包括Clock、Bmal1、Per(Period)、Cry(Cryptochrome)等,它们通过相互作用形成正反馈和负反馈回路,维持着大约24小时的生物节律。
生物节律的研究不仅增进了我们对生命基本机制的理解,还揭示了节律基因失调与多种疾病之间的联系,如睡眠障碍、抑郁症、肥胖症以及某些类型的癌症。因此,节律基因的挖掘与分析对于发现潜在的治疗靶点、开发新的药物以及改善人类健康具有重大意义。
随着生物信息学和基因组学技术的发展,我们现在能够在全基因组水平上识别和分析节律基因,提供了研究生物节律调控网络的新工具和方法。通过这些高通量技术,科学家可以探索和揭示更多未知的节律基因及其在生物钟调控中的作用,进一步深化我们对生物节律复杂性的理解。
在你的专题研究中,你可以进一步探讨特定节律基因的功能、它们在不同生物体中的保守性,以及环境因素如何影响节律基因表达模式。此外,特别关注如何利用现代生物技术,如转录组学分析和基因编辑工具,来挖掘、分析和操纵节律基因,将为生物医学研究开辟新的道路。
参考文献建议
虽然我不能直接提供论文引用,但我建议查找以下类型的文献进行参考:
综述文章,特别是那些关于生物节律和节律基因的最新进展。
原创研究文章,特别是那些应用了转录组学分析来研究节律基因表达模式的研究。
方法论文章,介绍如何使用特定的生物信息学工具和分析方法来识别和分析节律基因。
通过这些文献,你可以获得最新的研究进展
挖掘和分析节律基因的科学和实际应用意义深远,触及从基础生物学研究到临床医学,乃至农业和环境科学的多个领域。以下是几个关键领域,展示了节律基因研究的重要性和潜在应用:
1. 医学研究和治疗
- 疾病诊断与预防:研究发现,许多疾病的发生与生物节律的紊乱有关,如心血管疾病、肥胖、糖尿病、抑郁症和某些类型的癌症。通过理解节律基因的功能及其变异对疾病风险的影响,可以开发出新的疾病诊断标志物和预防策略。
- 药物疗效和副作用:药物代谢和药效往往受到生物钟的调控。节律基因研究有助于确定最佳的药物给药时间,以最大化疗效并减少副作用,这一策略被称为时间药理学或慢性药物治疗。
2. 农业生产
- 作物生长与产量:植物的生长发育和产量受到内部生物钟的控制。通过修改植物的节律基因,可以提高作物的适应性、增加产量和改善品质,特别是在面对环境压力(如干旱或温度变化)时。
- 畜牧业:在畜牧业中,动物的生长、繁殖和健康也受到生物钟的影响。了解和调节这些动物的节律基因表达,可以提高肉类和乳制品的生产效率和质量。
3. 环境保护与气候变化适应
- 物种适应性:随着全球气候变化,研究物种如何通过调整其生物节律来适应新的环境条件变得尤为重要。节律基因研究有助于预测和促进物种对快速变化环境的适应能力。
- 生态系统健康:生物节律调节着生态系统中的许多关键过程,包括授粉、捕食和迁徙。通过理解不同物种节律基因的作用,可以帮助保护生态平衡,对于维持生态系统的健康和多样性至关重要。
4. 精准医疗和个性化治疗
- 基因定制治疗:节律基因研究的进展为精准医疗提供了新的机会。通过分析个体的节律基因型,医生可以为患者设计更加个性化的治疗方案,包括药物选择和生活方式调整,以优化治疗效果。
总之,节律基因的挖掘与分析不仅增进了我们对生命过程的基本理解,还为诸多领域带来了创新的应用可能性。随着研究的深入,这些领域有
**本章目标**:概述本章的目的和结构,说明将要介绍的工具、数据和分析方法。
本章目标
本章旨在深入探索节律基因的挖掘与分析,特别是通过使用现代生物信息学工具和大数据分析方法来揭示这些基因在生物体中的作用和调控机制。我们将详细介绍和演示如何利用特定的分析工具——Rain插件——来识别和分析节律基因,以及如何将这些方法应用于具体的生物学研究案例中。通过本章的学习,读者将获得关于节律基因研究的深入理解,并掌握一套实用的技术和方法,以促进未来的科学发现和应用。
结构概览
13.1 简介:首先,我们将介绍节律基因的基本概念和它们在生物体中的重要性,以及研究这些基因对于医学、农业和环境科学等领域的潜在意义。
13.2 Rain插件的安装与使用:本节将提供Rain插件的详细安装指南,并介绍其主要功能。我们将通过实例演示如何使用Rain进行节律基因的数据处理和分析,帮助读者熟悉这一工具的操作流程。
rain介绍
根据您提供的文件,以下是关于Rain插件的介绍,可以插入到您的章节中作为13.2 Rain插件的安装与使用的一部分。
### RAIN介绍
Rain是一种高效的算法,专门设计用于检测时间序列中的节律性变化。它通过测试伞形替代假设(Mack & Wolfe, 1981)来识别时间序列数据中的节律上升和下降模式。Rain插件的核心价值在于其对时间序列分析的通用性和灵活性,使其能够广泛应用于生物学、医学、环境科学等多个领域的研究中。
主要功能和应用
- 时间序列分析:Rain能够处理一系列的时间序列数据,无论是规则还是不规则的间隔采样,提供了一种准确计算节律模式的方法。
- 灵活的数据格式支持:支持具有重复测量值或缺失值的时间序列,能够处理不同时间点的重复测量,为复杂的时间序列分析提供了强大的工具。
- 峰形识别和输出:一个关键优势是能够检测任意非对称的振荡。通过
peak.border参数控制非对称的限制,Rain可以识别时间序列中的复杂模式。
使用场景
- 生物节律研究:可以用于分析基因表达、生理活动等随时间变化的生物学过程,特别适用于探索内部生物钟和环境因素之间的相互作用。
- 环境监测:对于环境科学研究,如温度变化、海洋潮汐等自然现象的时间序列分析,Rain提供了一种有效的分析工具。
- 医学研究:在医学领域,对于分析患者生理参数(如心率、血压)随时间的变化模式,Rain能够揭示潜在的健康风险和节律失调。
实践应用
通过使用具体的数据集,例如Menet等人(2012)提供的小鼠肝脏基因表达高通量测序数据,Rain插件不仅展示了其在现实数据集上的应用能力,还揭示了除了中心分子机制(如Per1基因)之外,一些具有高度非对称振荡的基因(例如ni-2、Rnf144b、Mtrr),这些在仅假设正弦波形时可能被忽视的基因。
结论
Rain插件的开发极大地丰富了时间序列分析的工具箱,为研究者提供了一种强大而灵活的解决方案,以探索和理解复杂的时间序列数据中的节律性模式。其在多个科学领域内的应用潜力巨大,尤其是在揭示生物节律的基础机制和应对环境变化方面。
您可以根据这个介绍来构建Rain插件的使用说明,详细介绍其安装流程、基本用法以及如何应用于具体的科研项目中。
13.3 实例:基于鸡松果体转录组数据节律基因的挖掘与分析:
- 13.3.1 数据准备:介绍如何准备和预处理用于分析的转录组数据。
- 13.3.2 数据分析流程:详细阐述使用Rain插件进行数据分析的具体步骤,包括数据导入、节律基因的识别和分析等。
- 13.3.3 结果解释:如何解释分析结果,包括识别的节律基因及其表达模式。
- 13.3.4 讨论:讨论分析结果的生物学意义,以及这些发现对于理解鸡松果体功能和生物节律调控机制的影响。
- 13.3.5 挑战与展望:讨论在节律基因分析过程中遇到的挑战,以及未来研究的方向。
13.4 分析工具和方法的比较:本节将比较Rain插件与其他生物信息学工具在节律基因分析方面的优势和局限性,为读者提供更广阔的技术视角。
13.5 总结:概括本章的关键内容和学到的技能,强调节律基因研究的重要性及其在各个领域的应用潜力。
13.6 本章练习题:提供一系列练习题,旨在加深读者对本章内容的理解和掌握,包括理论问题和实际操作练习。
参考文献:列出所有用于编写本章的参考资料,提供进一步阅读和学习的资源。
通过本章的学习,读者不仅将获得关于节律基因研究的理论知识,还将掌握实际的分析技能,为进一步的科研工作和应用提供坚实的基础。
13.2 Rain插件的安装与使用
- 安装指南:提供详细的Rain插件安装步骤,包括必要的系统要求和下载链接。
- 功能介绍:解释Rain插件的主要功能和优势,包括它如何帮助识别和分析节律基因。
- 使用示例:通过简单的例子展示如何使用Rain插件进行数据分析,可以是一个简化的流程图或步骤说明。
13.3 实例:基于鸡松果体转录组数据节律基因的挖掘与分析
- 13.3.1 数据准备:介绍所使用的鸡松果体转录组数据来源、类型和预处理步骤。
- 13.3.2 数据分析流程:详细说明使用Rain插件分析节律基因的步骤,包括任何预处理、筛选和统计分析方法。
- 13.3.3 结果解释:展示分析结果,包括节律基因的识别和其表达模式的图示。
- 13.3.4 讨论:分析结果的意义,包括对生物节律的理解、潜在的生物学影响,以及可能的应用领域。
- 13.3.5 挑战与展望:讨论在分析过程中遇到的挑战,以及未来研究的可能方向。
13.4 分析工具和方法的比较
- 比较Rain插件与其他节律基因分析工具的优缺点,包括它们的应用范围、易用性、以及分析的准确性和复杂性。
13.5 总结
- 总结章节的关键点,强调节律基因分析的重要性和应用前景。
13.6 本章练习题
- 设计几个问题和练习,以帮助读者巩固本章的关键概念和分析技能。
参考文献
- 列出所有参考的研究文献、书籍和在线资源,为读者提供进一步学习的机会。
在写作过程中,确保内容准确无误,并尽可能使用图表、流程图和示例代码来辅助说明。此外,鼓励读者动手实践,可能通过提供数据集链接或模拟数据,让读者能够直接应用所学的知识。最后,保持语言清晰简洁,避免不必要的专业术语,使得内容对于广泛的读者群体都是可接
有rain插件等等的,shiny
确实存在一个名为 rain 的R包,它专注于时间序列中节律性行为的检测。rain 包使用非参数方法来识别时间序列数据中的节律,适用于处理包含10到100个测量值的时间序列。这一特性使其在分析生物数据时特别有用,比如微阵列、蛋白质质谱或代谢组测量数据,能够检测出例如昼夜节律或细胞周期等振荡行为。该包优势在于它能够处理异常值、缺失值,并且不预设任何特定的波形,使其在探测振荡行为时表现优异。
要安装 rain 包,你需要使用 Bioconductor 的 BiocManager::install() 方法,确保你的R版本适配。具体的安装命令如下:
1 | if (!requireNamespace("BiocManager", quietly = TRUE)) |
这个包的详细文档和使用指南可以通过其 Bioconductor 页面 或 rdrr.io 页面 获取。在这些页面上,你可以找到关于如何使用 rain 包进行节律性分析的详细信息,包括示例代码和函数说明,有助于你更有效地探索和分析时间序列数据中的节律性行为。
rain 包通过使用非参数方法来检测时间序列中的节律性行为,尤其适用于生物学数据分析,比如基因表达、蛋白质质谱或代谢组数据。其核心功能是识别时间序列数据中的周期性波动,而不预设任何特定波形,这使得该包在处理具有复杂波动模式的生物数据时显得尤为强大。
工作原理
- 非参数方法:
rain包利用非参数统计方法来识别数据中的节律性模式。这意味着它不依赖于事先定义的数学模型来拟合数据,而是通过数据本身的分布来探索周期性信号。 - 处理异常值和缺失值: 对于生物学数据而言,异常值和缺失值是常见的问题。
rain包设计了算法来适应这些问题,确保分析的鲁棒性。 - 优化时间序列: 该包特别适用于分析包含10到100个测量点的时间序列,使其在处理中等规模数据集时效率较高。
- 应用范围: 由于不预设特定波形,
rain包可以用来检测各种生物周期,如昼夜节律、细胞周期等,在基因组学、蛋白质组学和代谢组学等多个领域都有应用。
使用示例
虽然不能直接展示具体代码,rain 包通常的使用流程包括几个步骤:数据准备、调用rain函数进行节律性分析,以及结果的可视化和解释。通过提供时间序列数据,用户可以指定分析的周期范围、时间间隔等参数,rain 将返回周期性强度的统计显著性评估,以及周期信号的可能位置。
这种方法尤其适合于探索和验证生物数据中未知的、可能的节律性模式,而无需依赖特定的假设或复杂的数学模型。
更多关于rain包的细节,包括如何安装、如何使用以及如何解读其输出,建议查看其在Bioconductor上的官方文档以及rdrr.io上的用户指南。这些资源提供了丰富的信息和示例,可以帮助用户有效地利用rain包进行生物数据的周期性分析。
先将基因表达矩阵转置一下,行是样本(代表时间序列),列是基因
menetRNASeqMouseLiver2 <- t(menetRNASeqMouseLiver)
使用rain函数检测时间序列中的节律性
deltat为时间间隔,本示例中小鼠肝脏样本是4h采样一次
period是周期,本示例中研究的是小鼠24h的昼夜节律
measure.sequence指定每个时间点的生物学重复样本数量,本示例中每个时间点各包含3个生物学重复
peak.border,该方法检验时间过程是否由周期性的上升和下降斜率组成,上升部分相对于整个周期的分区由peak.border指定
method指定检测方法,本示例使用independent代表多个周期被解释为一个周期的重复
adjp.method,p值校正方法
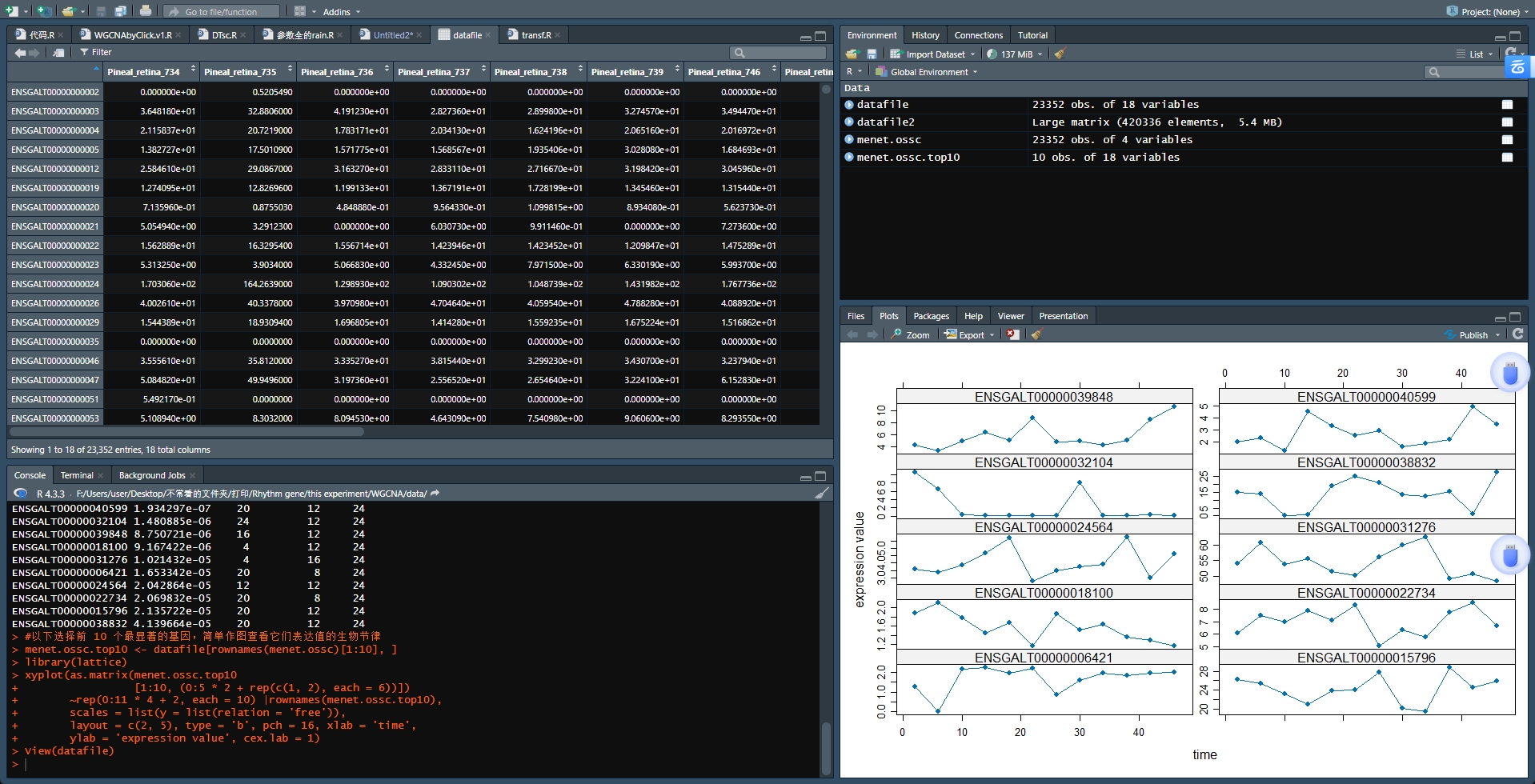
menet.ossc <- rain(menetRNASeqMouseLiver2, deltat = 4, period = 24, measure.sequence = c(3, 3, 3, 3, 3, 3),
peak.border = c(0.3, 0.7), method = ‘independent’, adjp.method = ‘ABH’, verbose = TRUE)
查看结果的前几行
head(menet.ossc)
根据显著性p值由小到大排个序
menet.ossc <- menet.ossc[order(menet.ossc$pVal), ]
head(menet.ossc, 10)
将结果保存到文件
write.table(menet.ossc, ‘menet.ossc.txt’, sep = ‘\t’, col.names = NA, quote = FALSE)
library(shiny)
library(lattice)
library(rain)
library(DT)
UI Function for uploading RDA files
mod_rda_file_ui <- function(id, label = “RDA file”){
ns <- NS(id)
tagList(
fileInput(ns(“file”), label, accept = c(“.rda”)),
checkboxInput(ns(“heading”), “Has heading”)
)
}
Server Function for processing RDA files
mod_rda_file_server <- function(id) {
moduleServer(
id,
function(input, output, session) {
userFile <- reactive({
input$file
})
dataframe <- reactive({
if (!is.null(userFile())) {
readRDS(userFile()$datapath)
}
})
return(dataframe)
}
)
}
ui <- fluidPage(
sidebarLayout(
sidebarPanel(
mod_rda_file_ui(“datafile”, “User data (.rda format)”),
numericInput(“num1”, label = “deltat”, value = 4),
numericInput(“num2”, label = “period”, value = 24),
numericInput(“num3”, label = “nr.series”, value = 2),
numericInput(“num5”, label = “period_delta”, value = 0),
selectInput(“method”, “Method”, choices = c(“Independent”, “Longitudinal”), selected = “Independent”),
checkboxInput(“na_rm”, “Remove NAs”, value = FALSE),
selectInput(“adjp_method”, “Adjust P-Value”, choices = c(“ABH”, “BY”, “BH”), selected = “ABH”),
sliderInput(“peak”, label = “Peak Border”, min = 0, max = 1, value = c(0.3, 0.7), step = 0.1),
textInput(“measure_sequence”, “Measure Sequence (comma-separated)”, value = “2,2,2,2,2,2”),
radioButtons(“verbose”, label = “Verbose”, choices = c(“TRUE”, “FALSE”), selected = “FALSE”),
actionButton(“refresh”, “Refresh”),
downloadButton(“download_data”, “Download Data”)
),
mainPanel(
dataTableOutput(“table”),
DT::dataTableOutput(“rain_matrix”),
plotOutput(“curve_plot”)
)
)
)
server <- function(input, output, session) {
datafile <- mod_rda_file_server("datafile")
observeEvent(input$refresh, {
if(is.null(datafile())) return(NULL)
measure_seq_vector <- as.numeric(unlist(strsplit(input$measure_sequence, ",")))
results <- reactive({
rain(t(as.matrix(datafile())),
deltat = input$num1,
period = input$num2,
nr.series = input$num3,
measure.sequence = measure_seq_vector,
peak.border = input$peak,
verbose = as.logical(input$verbose))
})
output$table <- renderDataTable({
datafile()
})
output$rain_matrix <- DT::renderDT({
req(results())
datatable(data.frame(results()), options = list(pageLength = 20), rownames = FALSE)
})
output$curve_plot <- renderPlot({
req(results())
best <- order(results()$pVal)[1:10]
xyplot(t(as.matrix(datafile()[best, ]))
~ rep(0:11 * input$num1 + input$num1/2, each = 10) | rownames(datafile())[best],
scales = list(y = list(relation = 'free')),
layout = c(2, 5), type = 'b', pch = 16, xlab = 'time',
ylab = 'expression value', cex.lab = 1)
})
output$download_data <- downloadHandler(
filename = function() {
paste("results", ".csv", sep = "")
},
content = function(file) {
req(results())
write.csv(as.data.frame(results()), file, row.names = FALSE)
}
)
})
}
shinyApp(ui = ui, server = server)
rain 函数是用于检测时间序列数据中的节律性的,例如基因表达随时间的变化。这在生物钟研究中特别有用,你想了解生物过程如何随周期波动,比如每24小时的昼夜节律。让我们逐个解释你提到的参数:
x:数据矩阵,行代表样本(时间序列),列代表基因。如果原始数据是基因在行、样本在列,需要对矩阵进行转置,确保每行对应一个时间点,每列对应一个基因。
deltat:样本之间的时间间隔。例如,在你的示例中,小鼠肝脏样本每隔4小时收集一次。
period:研究中的周期长度,比如提供的示例中的24小时昼夜节律。
period.delta:该参数允许测试一系列可能的周期范围,其中
0表示只测试指定的周期。如果该参数设置为非零值,则指定允许的周期偏差,以适应周期长度的轻微变化。peak.border:该参数定义周期内预期出现增加和减少的相位,相对于整个周期长度。该方法测试时间序列是否由周期性的增长和下降组成,其中增长相位由
peak.border指定。例如,c(0.3, 0.7)表示周期的增长阶段预计发生在周期的30%到70%之间。nr.series:要分析的时间序列数。默认情况下,它设置为1,表示每列(基因)都被独立处理。如果您的数据包含每个基因的多个时间序列,您将相应地调整此参数。
measure.sequence:指定每个时间点的生物学重复次数。在你的示例中,每个时间点都有2个生物学重复,由
c(2, 2, 2, 2, 2, 2)表示。method:定义用于检测节律性的方法。
independent方法将多个周期视为单个周期的重复。这对于分析数据很有用,您假设生物过程在每个周期内重复相同。na.rm:逻辑参数,指示是否应在分析之前删除NA(缺失)值。
adjp.method:用于调整p值以控制多重检验问题的方法。”ABH”是指自适应Benjamini-Hochberg程序。
verbose:逻辑参数,当设置为TRUE时,在函数执行期间提供额外的输出,用于跟踪进度或调试。
代码片段演示了如何转置基因表达矩阵,应用rain函数来检测小鼠肝脏样本每4小时收集一次,在24小时周期内的节律基因,并最终根据节律性检测的显著性(p值)对结果进行排序。最后,排序后的结果被打印出来,可以保存到文件中。
1 |
|
User
以rain包的内置数据集”menetRNASeqMouseLiver”为例作演示,该数据集来自于一项对小鼠昼夜节律转录调控的研究。小鼠饲养在12h光照/12h黑暗的24h周期中,饲养一段时间后,每隔4h取样一次,通过高通量测序获得的小鼠肝脏基因表达谱的时间序列。
我就想研究这个数据,我能通过rain处理以后,选择p值小于0.05的,然后进行WGCNA分析吗?
是的,你可以通过rain包处理menetRNASeqMouseLiver数据集后,选择p值小于0.05的基因进行WGCNA(加权基因共表达网络分析)分析。rain包会帮助你识别出具有显著节律性的基因,而WGCNA可以进一步分析这些基因在功能上的相互作用和模块化网络。这样,你可以探索具有相似表达模式的基因群体,从而理解基因功能以及它们如何相互作用来调控小鼠的昼夜节律转录。
我还有个问题啊,上面的那个数据只是采集了24小时的数据,为什么最后生成的图像,横坐标的时间已经超过24了
这种情况通常是因为在进行时间序列分析时,尤其是涉及到昼夜节律的研究,研究人员可能会连续观察多个周期以更好地理解节律模式。虽然实际采集的数据仅覆盖了24小时周期,但在分析时,可以将这个周期视作重复,以模拟和预测基因表达在随后周期的行为。因此,图像中横坐标时间超过24小时是为了展示模式的持续性和重复性,提供更全面的节律表达视图。
验证,
1 | menet.ossc <- rain(datafile2, deltat = 4, period = 24, |
三个都不一样,鬼知道当时怎么弄的
1 | menet.ossc <- rain(datafile2, deltat = 4, period = 24, measure.sequence = c(3, 3, 3, 3, 3, 3), |
链接:https://caiyun.139.com/m/i?065CtdZRkftjy
提取码:R1qS
复制内容打开移动云盘PC客户端,操作更方便哦
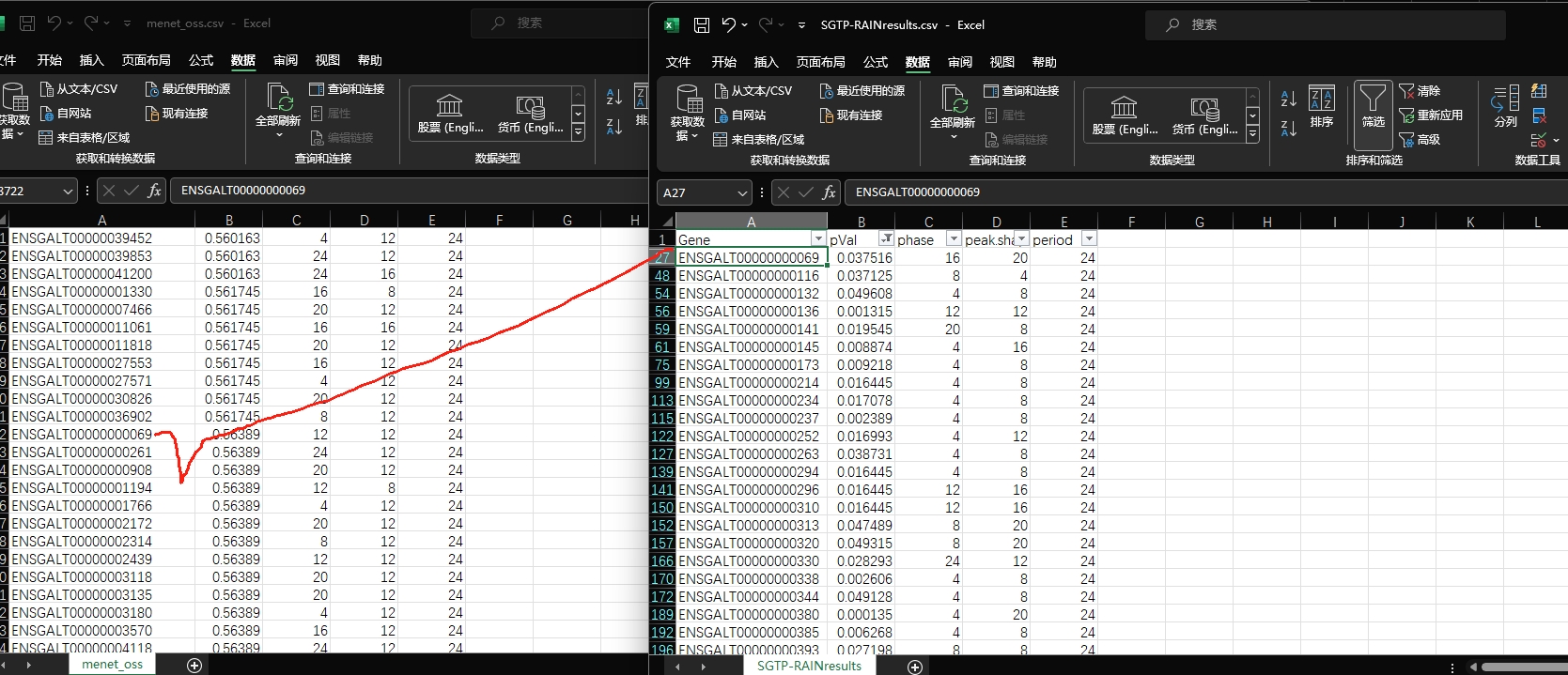
pVal phase peak.shape period
DGKE 1.52441e-10 4 16 24
DBNDD2 1.52441e-10 4 16 24
KLHL9 1.52441e-10 20 8 24
MDGA1 1.52441e-10 4 16 24
DDC 1.52441e-10 20 8 24
RBP4 1.52441e-10 4 16 24
HSPH1 1.52441e-10 4 16 24
ZDHHC5 1.52441e-10 20 8 24
GPRIN2 1.60063e-10 20 8 24
TARDBP 1.60063e-10 4 16 24
这个表格展示了一系列基因在特定条件下的表达特征,包括它们的p值(pVal)、表达相位(phase)、峰形(peak.shape)、和周期(period)。p值是用来评估结果发生的随机性的统计指标,较低的p值(通常小于0.05)意味着结果具有统计学意义,表明观察到的模式不太可能是偶然发生的。在这个表格中,所有列出的基因都有非常低的p值(约为1.5e-10),表明它们的表达模式具有高度的统计学意义。
- 相位(Phase)和周期(Period)反映了基因表达随时间变化的模式。相位可能代表了基因表达达到最高点的时间点,而周期则代表整个表达周期的长度。在这个表格中,有两组相位:4和20,都在24小时的周期内,可能指示这些基因在一天中的特定时间表达活跃。
- 峰形(Peak.shape)可能指基因表达模式的具体特征,如表达高峰的宽度或形状,这里主要有两种形状:8和16,可能代表不同的表达强度或持续时间。
现在,让我们快速浏览一下这些基因的功能:
- DGKE:参与细胞信号转导过程,特别是与脂质代谢相关。
- DBNDD2:功能可能与蛋白质降解或信号转导有关。
- KLHL9:可能涉及蛋白质泛素化和细胞周期调控。
- MDGA1:在神经系统发育中起作用,特别是在神经元定位和突触形成中。
- DDC:多巴胺合成的关键酶,涉及神经递质的产生。
- RBP4:视黄醇结合蛋白,涉及维生素A的运输。
- HSPH1:热休克蛋白,涉及蛋白质折叠和保护细胞免受应激损伤。
- ZDHHC5:可能涉及蛋白质棕榈酰化,影响蛋白质的定位和功能。
- GPRIN2:可能参与G蛋白偶联受体的信号转导。
- TARDBP:与RNA代谢相关的蛋白质,可能影响基因表达的调控。
这些基因的功能范围从信号转导、蛋白质代谢、神经系统发育到应激响应,反映了生物体内复杂的分子网络和生理过程。基因表达的周期性变化可能与生物钟或其他周期性生理过程有关,反映了生物体对环境变化的适应。
在提供的列表中,一些基因可能与生物节律的调节有关,尽管需要更深入的生物学背景和文献查证来确定它们的确切作用。基于一般知识,我可以提供一些可能与生物节律调节相关的基因的概述:
**RBP4 (视黄醇结合蛋白4)**:虽然主要在肝脏中产生并参与视黄醇的运输,但RBP4也被发现与代谢病态相关,如糖尿照行和肥胖症。其节律性表达可能反映了生物钟对代谢过程的调控作用,但直接作用于节律调节的证据较少。
**DDC (多巴胺脱羧酶)**:这个酶参与多巴胺的生产,是神经递质合成的关键步骤。多巴胺与调节情绪、睡眠和多种神经功能有关,间接可能受到生物钟的影响。虽然DDC直接参与节律调节的证据有限,但其在调控与生物钟相关的生理过程中可能起作用。
**TARDBP (TAR DNA结合蛋白)**:TARDBP编码的蛋白TDP-43与多种神经退行性疾病有关,包括肌萎缩侧索硬化症(ALS)和前颞叶痴呆。尽管TARDBP主要与疾病状态相关,但作为转录调控因子,它可能间接影响与节律相关的基因表达。
大多数提到的基因主要参与代谢、神经传导和细胞应激反应等过程,直接与生物节律调节相关的证据可能不多。然而,生物节律的调节是一个复杂的网络,涉及多种信号途径和反馈机制。这些基因的节律性表达可能反映了生物钟如何通过调节特定基因的表达来影响广泛的生理过程。
为了确定这些基因是否具有已知的节律调节作用,需要查阅专门的科学文献和数据库。例如,可以通过查看基因功能研究、表达数据库(如GeneCards、NCBI Gene)和生物节律专门研究的文献来获取更准确的信息。此外,功能基因组学实验,如基因敲除或过表达研究,可以揭示这些基因在维持生物节律中的具体作用。
在一天的不同时间点,中文的描述通常与日常活动和自然光线的变化相关联:
- 0点:午夜或子夜,这通常是深夜时分,大部分人处于睡眠状态。
- 4点:凌晨,天开始亮起,但大多数活动还没开始,人们通常还在睡眠中。
- 8点:早晨,这是一天开始的时间,人们开始他们的日常活动,如吃早餐、上班等。
- 12点:中午,通常是午饭时间,太阳位于天空的最高点。
- 16点:下午或傍晚开始的时候,人们可能正在结束工作日的活动,准备傍晚的活动。
- 20点:晚上,通常是晚饭后的休闲时间,人们会进行家庭活动、娱乐或休息。
这些时间描述可能会根据季节的变化或特定文化习惯有所不同,但通常都与上述活动相对应。
可以发现,每个阶段,总有一个或两个模块与该阶段有很强的相关性,而且互不相同。
大概可以发现在黑暗阶段开始(X0)时, 与MEbrown模块有很强的正相关性(关联度为0.83,P<0.01),与MEpink模块有很强的负相关性(关联度为-0.62,P<0.01)。
凌晨阶段开始(X4)时, 与MEturguoise模块有很强的正相关性 (关联度为0.6,P<0.01)
早晨阶段开始(X8)时, 与MEblack模块有很强的正相关性(关联度为0.64,P<0.01)
中午阶段开始(X12)时, 与MEblue模块有很强的正相关性(关联度为0.64,P<0.01)
下午阶段开始(X16)时,与MEpurple模块和MEturguoise模块有很强的负相关性,关联度分别为-0.68,-0.61(P<0.01)
晚上阶段开始(X20)时,与MEyellow模块有很强的正相关性(关联度为0.81,P<0.01)
可以说明什么问题?
这些观察到的模块与时间点的相关性揭示了基因表达与小鼠肝脏的昼夜节律之间可能存在的密切联系。以下是这些结果可能表明的几个方面:
昼夜节律的基因调控:
- 强正相关性表明,在特定时间点,相应模块中的基因表达活跃,这些基因可能参与调控与该时间段相应的生理活动。例如,在黑暗阶段开始(X0)时,MEbrown模块中的基因表达增强,可能涉及夜间活动或新陈代谢途径的启动。
基因模块的特异性表达模式:
- 每个时间段有特定的基因模块表现出强相关性,说明不同的生物学过程可能在一天中的不同时间被激活。例如,MEyellow模块可能包含了在晚上阶段开始时(X20)特别活跃的基因。
负相关性的生物学意义:
- 负相关性意味着在某个时间点,相应模块中的基因表达被抑制。这可能反映了生物钟对这些基因的负向调控,或者表明这些基因在日间活动中更活跃,而在该时间点处于静默状态。
昼夜节律的复杂性:
- 昼夜节律是一个复杂的生物过程,涉及多个基因和信号途径。正负相关性的模块表明,在昼夜节律中,有一系列精细调控的基因表达波动,这些基因可能在不同的时间点对生物钟的调节起到了关键作用。
潜在的药物靶标:
- 由于这些模块在不同时间点具有显著的表达模式,其中的枢纽基因可能是潜在的药物靶标。这对于开发治疗昼夜节律紊乱或其他与生物钟相关疾病的策略是有意义的。
综上所述,这些数据提供了关于如何不同时间段的基因表达可能与昼夜节律相关的生物学过程相互作用的重要线索。进一步的研究可能包括这些模块内各个基因的功能注解和实验验证,以深入了解它们在昼夜节律中的具体角色。



