0,大概过程,要在Windows系统上下载并安装PLINK(一款常用于基因组学数据分析的免费、开源软件),你可以按照以下步骤进行:
步骤 1: 访问PLINK官方网站
- 打开你的网页浏览器,访问PLINK的官方网站:PLINK 1.9 或 PLINK 2.0,根据你的需求选择版本。PLINK 1.9是最广泛使用的版本,适用于大多数基因型数据分析,而PLINK 2.0则提供了更多的新功能和改进。
步骤 2: 选择适合Windows的版本下载
- 在下载页面,找到适用于Windows的版本。PLINK通常提供了预编译的二进制文件,适用于Windows、Linux和Mac OS X。对于Windows用户,你应该下载
.exe格式的文件。
步骤 3: 下载并解压(如果需要)
- 直接点击下载链接,下载将自动开始。下载完成后,你可能需要解压文件(如果它们是压缩的)。PLINK的Windows版本通常不需要安装,下载的
.exe文件就是可执行文件。
步骤 4: 运行PLINK
- 下载并解压(如果需要)后,你可以通过双击
.exe文件或在命令行窗口中导航到包含PLINK可执行文件的目录来运行PLINK。为了在任何命令行窗口中都能访问PLINK,你可以将PLINK的目录添加到系统的环境变量PATH中。
添加PLINK到系统环境变量
- 右键点击“此电脑”或“我的电脑”,选择“属性”。
- 点击“高级系统设置”。
- 在系统属性窗口中,点击“环境变量”按钮。
- 在“系统变量”区域,找到并选择“Path”变量,然后点击“编辑”。
- 在“编辑环境变量”窗口,点击“新建”,然后输入PLINK可执行文件所在的目录路径。
- 点击“确定”保存你的更改。
这样设置后,你就可以在任何命令行窗口中通过简单输入plink来运行PLINK了。
注意
由于我无法提供直接的下载链接或执行网络浏览功能,请确保从官方网站或可信赖的源下载软件,避免安全风险。
0.5,用法
PLINK是一款强大的基因组数据分析工具,用于进行大规模关联研究、群体遗传结构分析等。其命令行界面虽然初看可能令人望而却步,但实际上非常灵活,能够执行广泛的遗传数据分析任务。以下是一些基本的PLINK用法示例,以帮助你开始使用这个工具。
基本语法
PLINK的基本命令格式如下:
1 | plink --file [数据文件前缀] [选项] |
或者对于PLINK 2.0,使用:
1 | plink2 --[选项] |
数据格式转换
将PED/MAP格式转换为BED/BIM/FAM格式(二进制格式,更高效):
1 | plink --file mydata --make-bed --out mydata_bed |
这会生成mydata_bed.bed、mydata_bed.bim和mydata_bed.fam文件。
基础数据分析
品质控制(例如,过滤掉缺失率高的位点和个体):
1
plink --bfile mydata --geno 0.1 --make-bed --out mydata_filtered
这个命令会过滤掉所有缺失率超过10%的位点,并将结果输出到新的二进制文件中。
计算等位基因频率:
1
plink --bfile mydata --freq --out allele_freqs
生成包含每个位点等位基因频率的
allele_freqs.frq文件。进行关联分析:
1
plink --bfile mydata --assoc --out mydata_assoc
这会进行简单的关联分析,并将结果输出到
mydata_assoc.assoc文件中。
复杂分析
计算连锁不平衡(LD):
1
plink --bfile mydata --ld rs123456 rs7891011 --out ld_between_snps
计算两个SNP之间的连锁不平衡度量,并输出到
ld_between_snps.ld。进行群体结构分析:
1
plink --bfile mydata --cluster --mds-plot 2 --out mydata_mds
使用多维尺度分析(MDS)进行群体结构分析,并将结果输出到
mydata_mds.mds文件。
注意事项
- 在执行命令前,请确保你的数据文件(如
.ped和.map或.bed、.bim、.fam)位于当前目录,或使用绝对路径指定文件位置。 - 根据你的具体需求,PLINK提供了大量的选项和参数进行调整。它的官方文档非常详细,包含了各个命令和参数的使用说明,建议在进行具体分析前仔细阅读相关部分。
PLINK的强大之处在于其广泛的功能和灵活的命令行界面,通过组合不同的命令和选项,你可以执行几乎所有类型的遗传数据分析。
用法2
2,用plink代码在采样取得的vcf,转换成map,ped。命名为BB.map,BB.ped
plink –vcf BB.vcf –recode –out BB -chr-set 39
用plink代码在34号染色体,1339431-1343416位置进行筛选
plink –file BB –chr 34 –from-bp 1339431 –to-bp 1343416 –make-bed –out output_BB –chr-set 39
再用plink把得到的bim,fam,bed转换成ped和map格式
plink –bfile output_BB –recode –out new_choice_BB –chr-set 39
我尝试再试一下3,表型关联分析:
plink –bfile output_BB –pheno phenotypes.txt –allow-no-sex –logistic –adjust –output_choice_ logistic_results –chr-set 39
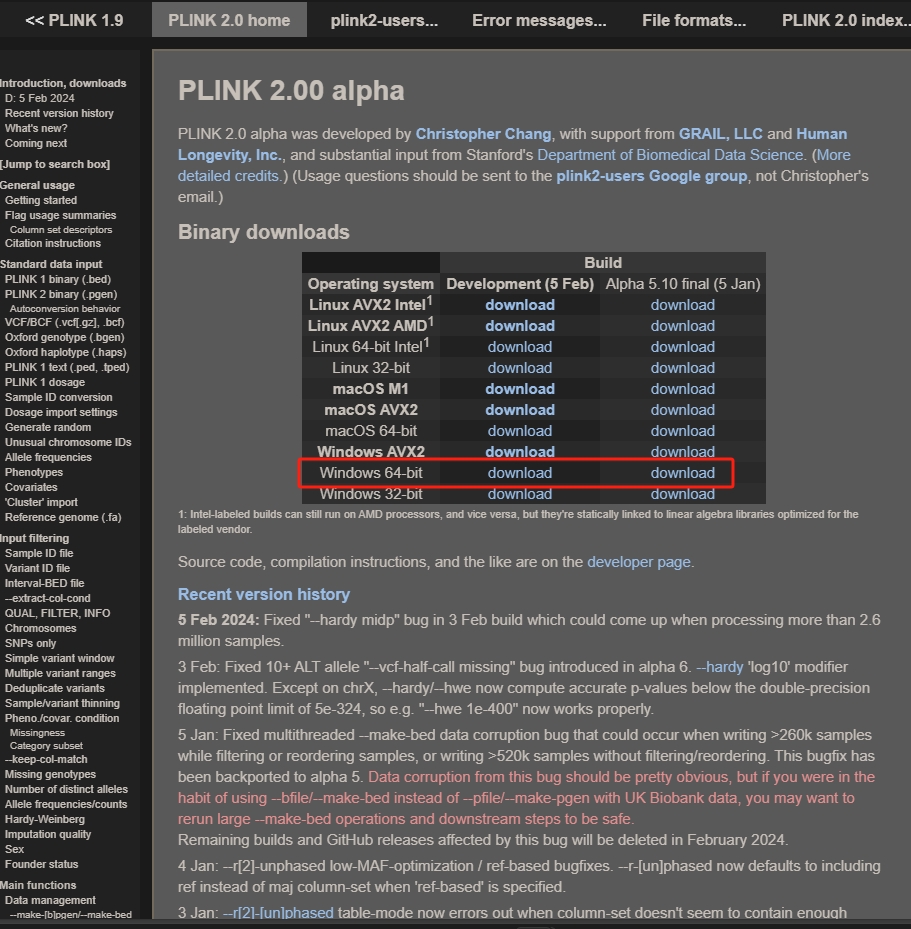
1,下载
https://www.cog-genomics.org/plink/2.0/
开始
首先,如果您的系统上未安装 plink 和/或 plink2,请下载并解压缩相应的二进制文件(v1.9、v2.0)。(或从 GitHub 克隆并重新编译。随着 alpha 和 beta 测试的继续,plink2 将变得越来越可用,但现在最好将其视为 v1.9 的补充而不是替代品。
2,先研究plink1
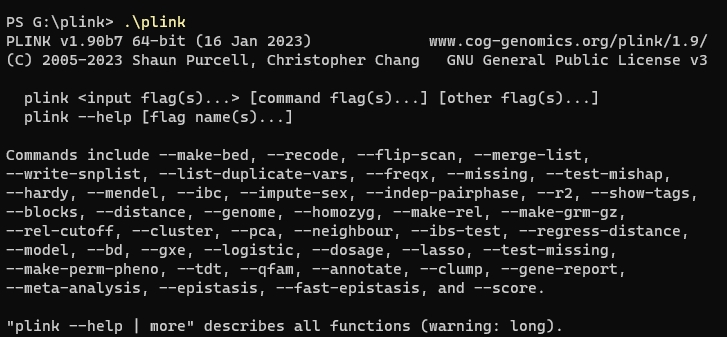
从你提供的输出信息来看,你已经成功运行了PLINK的命令行界面,并且PLINK给出了基本的使用方法以及一系列可用的命令。下面我会解释其中的一些关键命令和选项:
3,基本用法
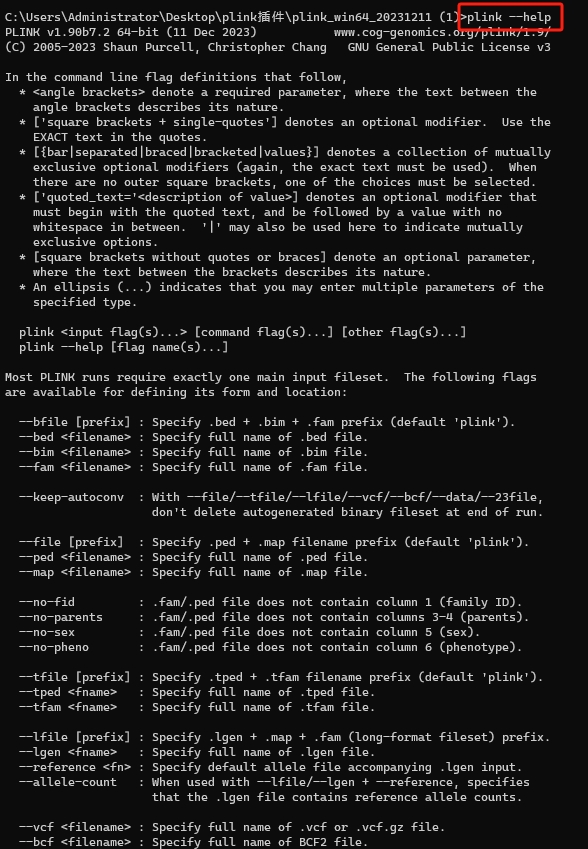
plink <input flag(s)...> [command flag(s)...] [other flag(s)...]: 这是PLINK的基本命令结构,其中<input flag(s)...>是指定输入文件的参数,[command flag(s)...]是指定要执行的分析或操作的参数,[other flag(s)...]是其他可选参数。plink --help [flag name(s)...]: 显示PLINK的帮助信息。如果指定了特定的标志名称,它会显示该标志的详细帮助信息。
常用命令
--make-bed: 将数据集转换为PLINK的二进制格式(.bed,.bim,.fam)。--recode: 将数据转换为其他格式,包括PLINK的文本格式(PED/MAP)或VCF。--freqx: 计算交叉等位基因频率。--missing: 统计个体和位点的缺失率。--hardy: 进行哈代-温伯格平衡检验。--mendel: 进行孟德尔错误分析。--ibc: 估算个体间的近亲系数。--impute-sex: 根据性染色体上的基因型数据估算性别。--pca: 进行主成分分析(PCA),常用于群体结构分析。--logistic: 进行逻辑斯蒂回归分析,常用于二分类表型的关联研究。--clump: 对GWAS结果进行整合,基于P值和物理距离来聚集关联信号。
这些命令覆盖了数据准备、基础统计分析、关联分析、群体遗传学分析等多个方面。PLINK的强大之处在于它提供了一整套工具,可以处理从数据准备到高级分析的整个流程。
要获取特定命令的详细帮助信息,可以使用--help标志后跟命令名,例如:
1 | plink --help --make-bed |
这将提供关于--make-bed命令的详细使用说明和选项。通过探索PLINK提供的各种命令和选项,你可以有效地执行各种遗传数据分析任务。
先用常规的这个命令吧,实现最简单的一个功能。
比如这个
用plink把得到的bim,fam,bed转换成ped和map格式,确认可以完成
plink –bfile output_BB –recode –out new_choice_BB –chr-set 39
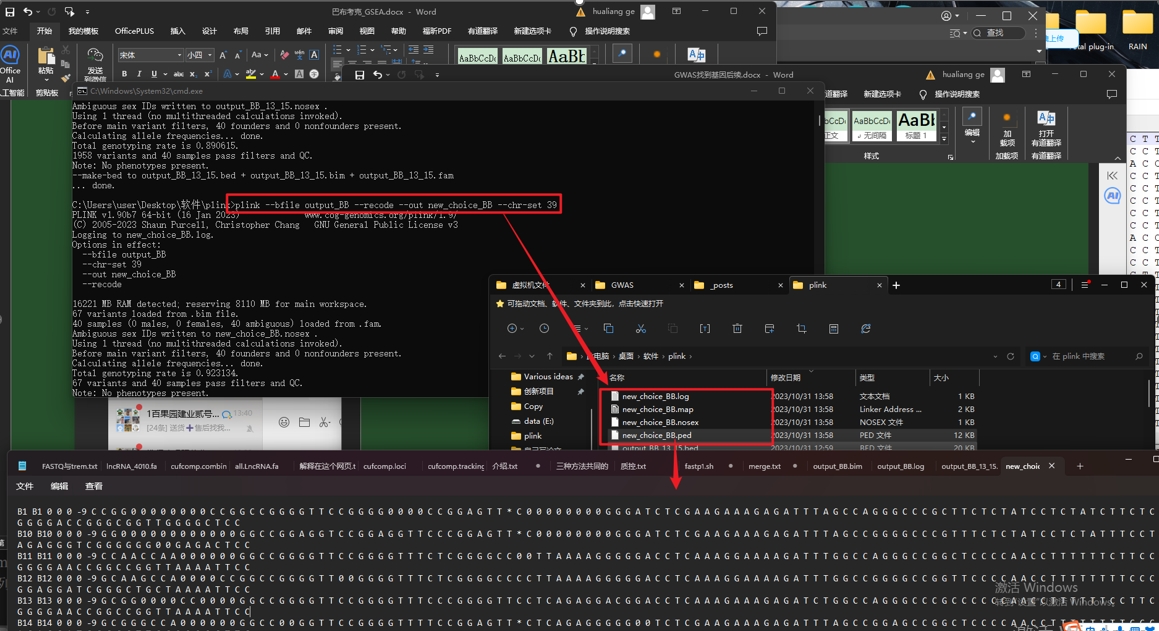
所以研究下插件
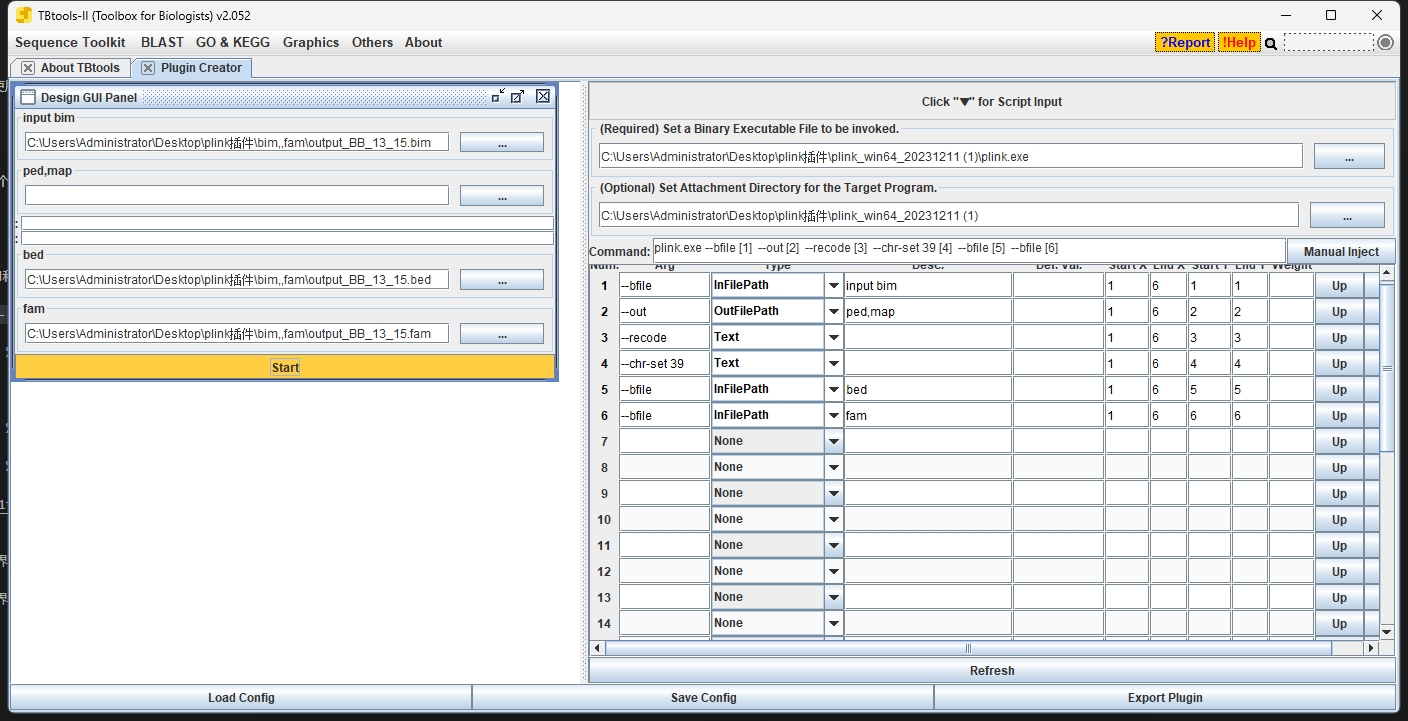
不行,报错,先保存C:\Users\Administrator\Desktop\plink插件\bim,,fam\1
先用最简单的吧
C:\Users\Administrator\Desktop\plink插件\plink_win64_20231211 (1)
1 | plink --file toy --recode vcf --out output_vcf |
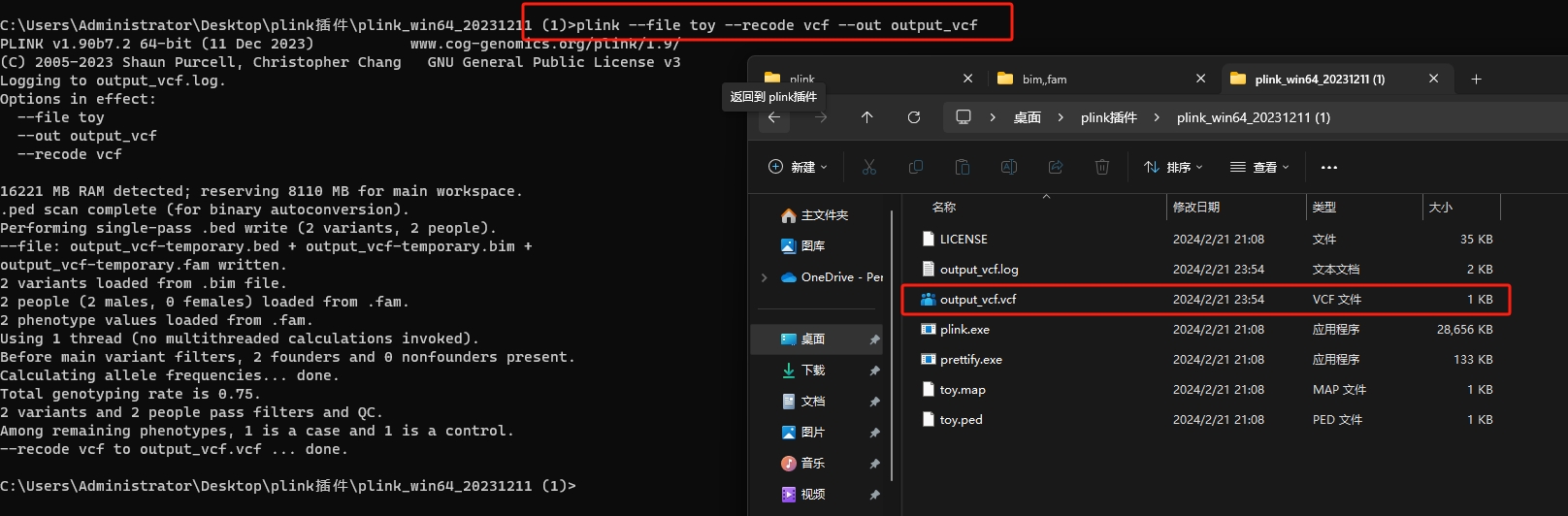
有个问题,好像不能直接输入多个文件?
插件1,先用vcf分吧,vcf变成ped,map
1 | plink --vcf output_vcf.vcf --recode --out toy_same |
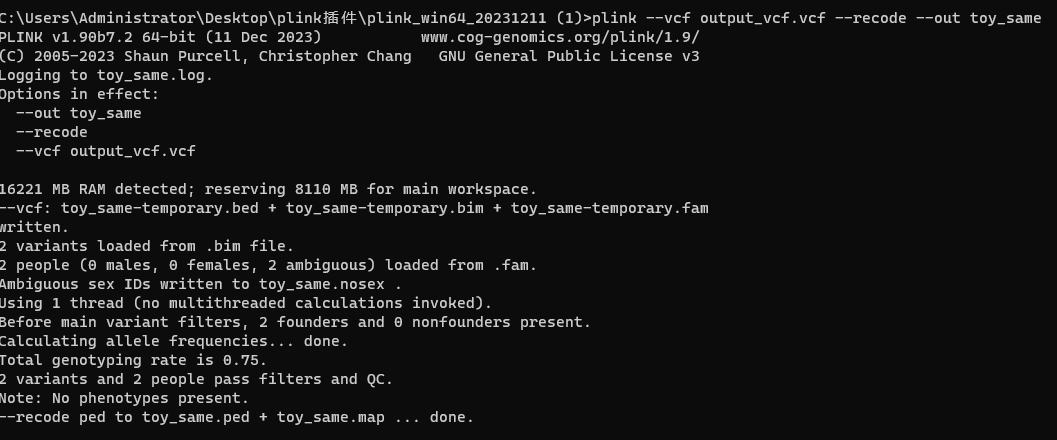
插件
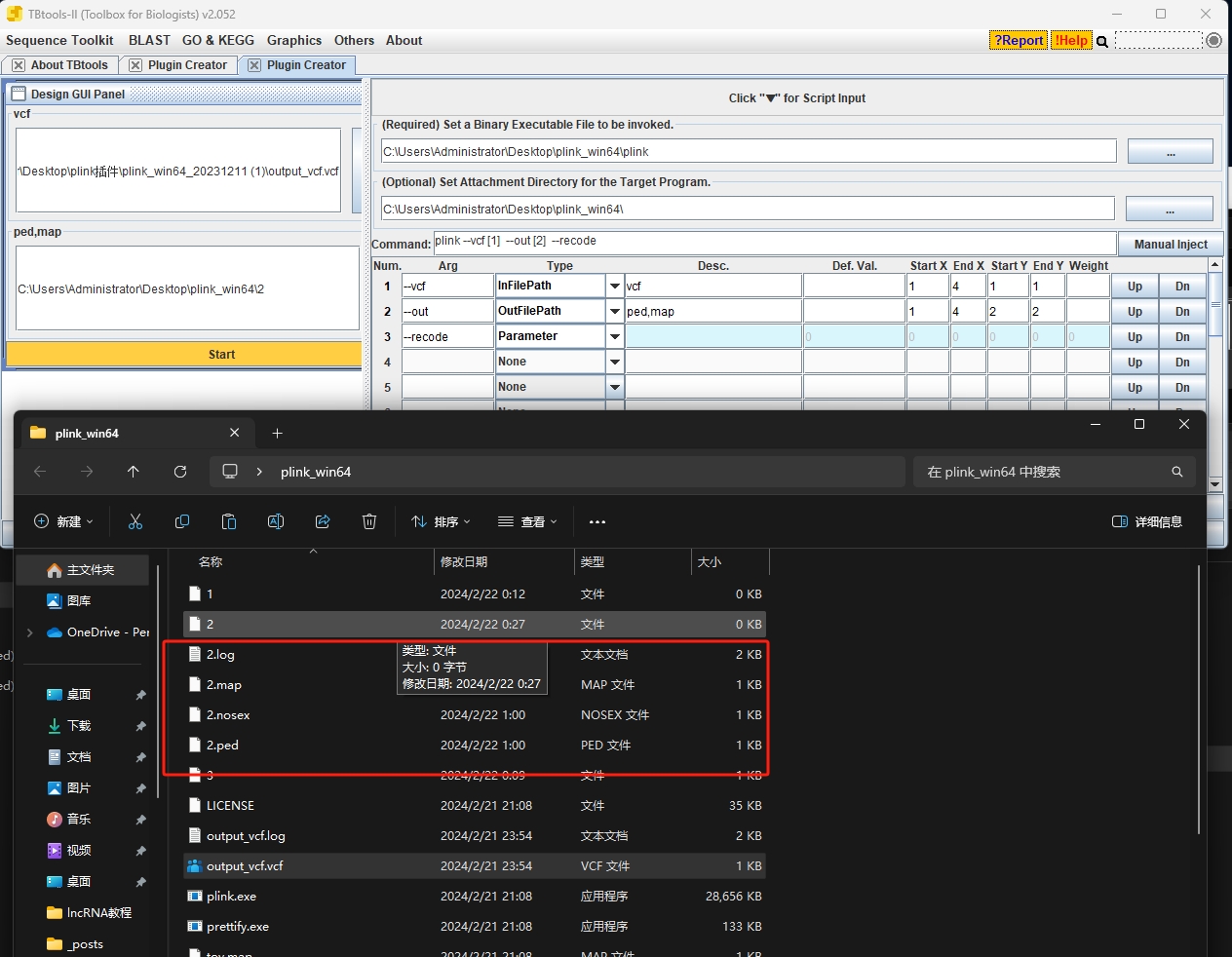
这个可以使用
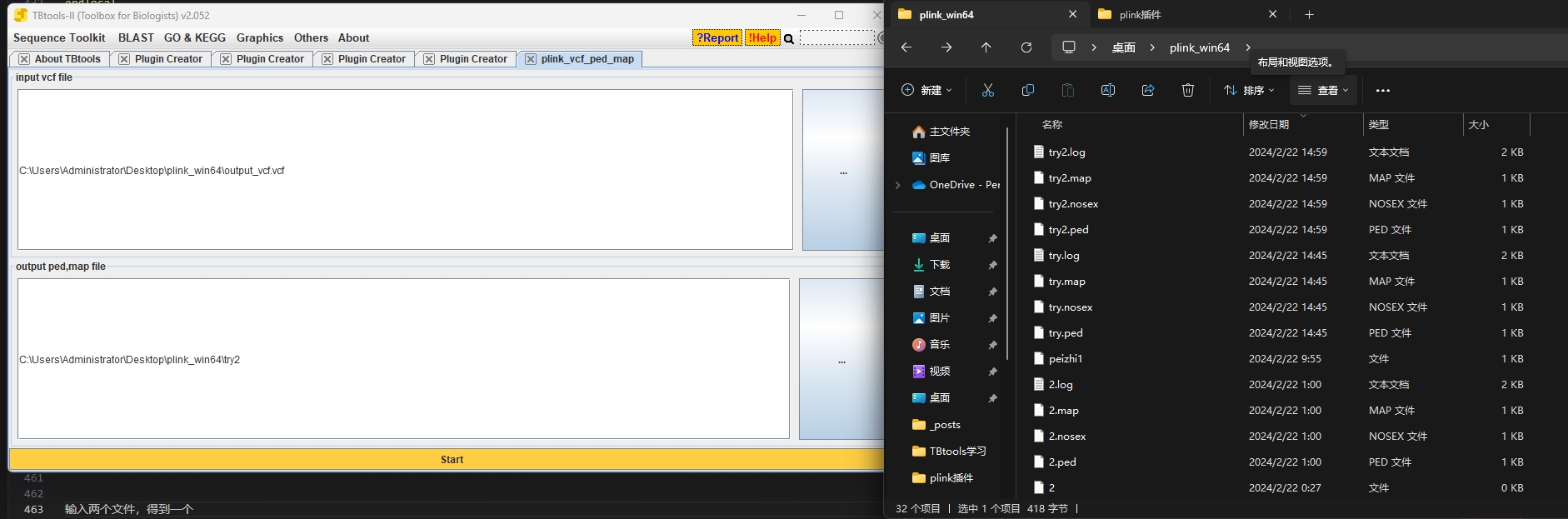
插件2,bim,fam,bed得到ped,map
那目前就是输入多个文件会报错
你的日志显示在尝试运行 PLINK 命令时出现了一个问题。PLINK 是一个用于执行大规模基因组关联分析的免费开源工具集。具体的错误是 PLINK 无法打开因 –file 标志参数格式错误指定的文件。让我们分析问题并找到解决方案:
问题分析
–file 标志在 PLINK 中用于指定输入文件的基本名(不含扩展名),PLINK 会寻找三个有相同基本名但扩展名分别为 .bed、.bim 和 .fam 的文件。错误信息表明 PLINK 试图打开一个名字很长、包含逗号和多个扩展名的文件,这并不符合 PLINK 对文件名格式的期待。
–file 标志的正确用法
正确使用 –file 标志的方式是提供输入文件的基本名,不包括任何扩展名。然后,PLINK 会自动寻找具有该基本名的 .bed、.bim 和 .fam 文件。例如,如果你的文件名为 example.bed、example.bim 和 example.fam,你应该在命令中使用 –file example。
解决方案
你需要修改命令,以正确指定输入文件的基本名,不包括扩展名或逗号。根据所提供的信息,命令应指向单一的基本文件名而非文件列表。如果 example 是 .bed、.bim 和 .fam 文件的预期基本名,修正后的命令部分应如下所示:
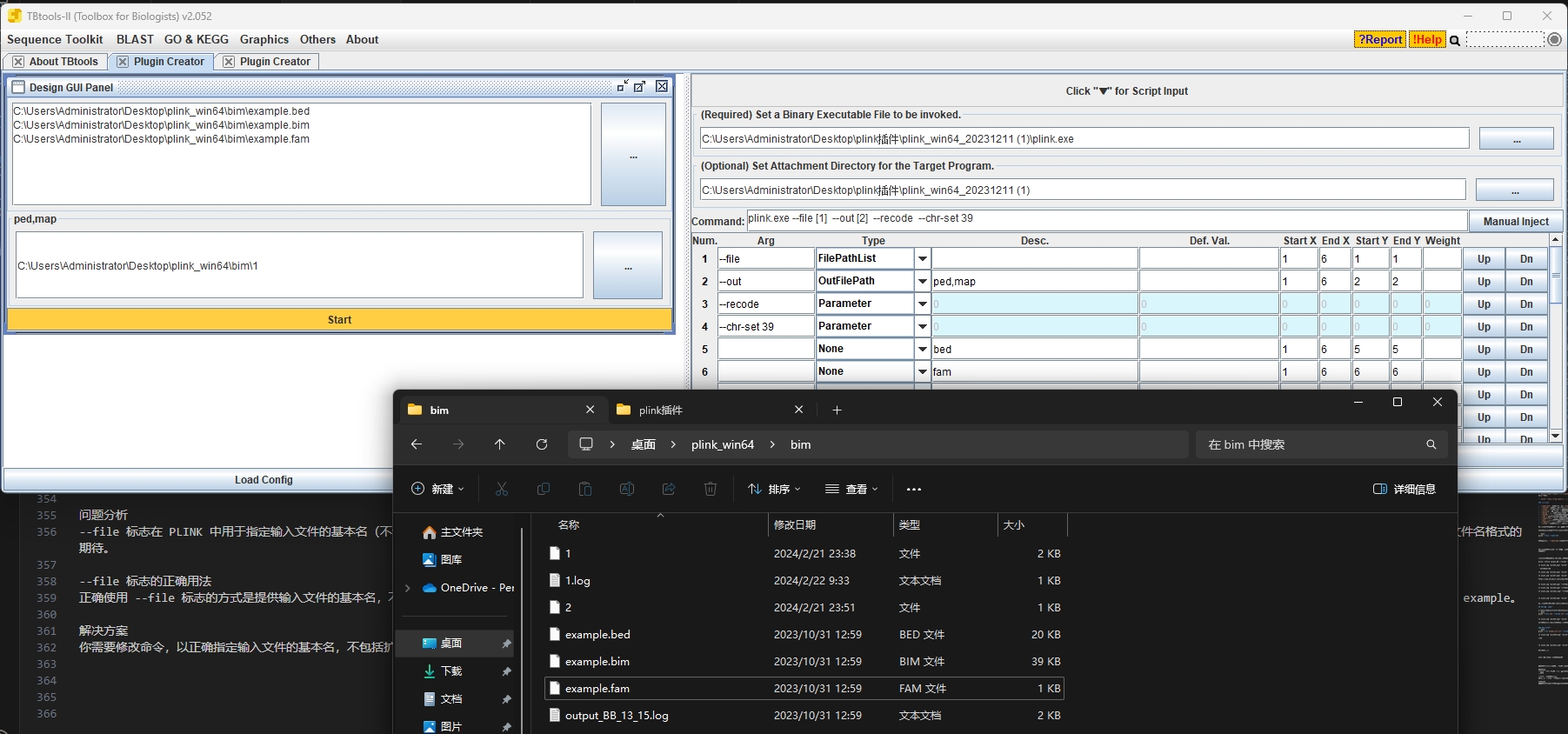
他运行的代码是这个
1 | plink.exe --file C:\Users\Administrator\Desktop\plink_win64\1\example.bed,C:\Users\Administrator\Desktop\plink_win64\1\example.bim,C:\Users\Administrator\Desktop\plink_win64\1\example.fam --out C:\Users\Administrator\Desktop\plink_win64\1\out_example --recode --chr-set 39 |
而我需要这个
1 | plink.exe --file C:\Users\Administrator\Desktop\plink_win64\1\example --out C:\Users\Administrator\Desktop\plink_win64\1\out_example --recode --chr-set 39 |
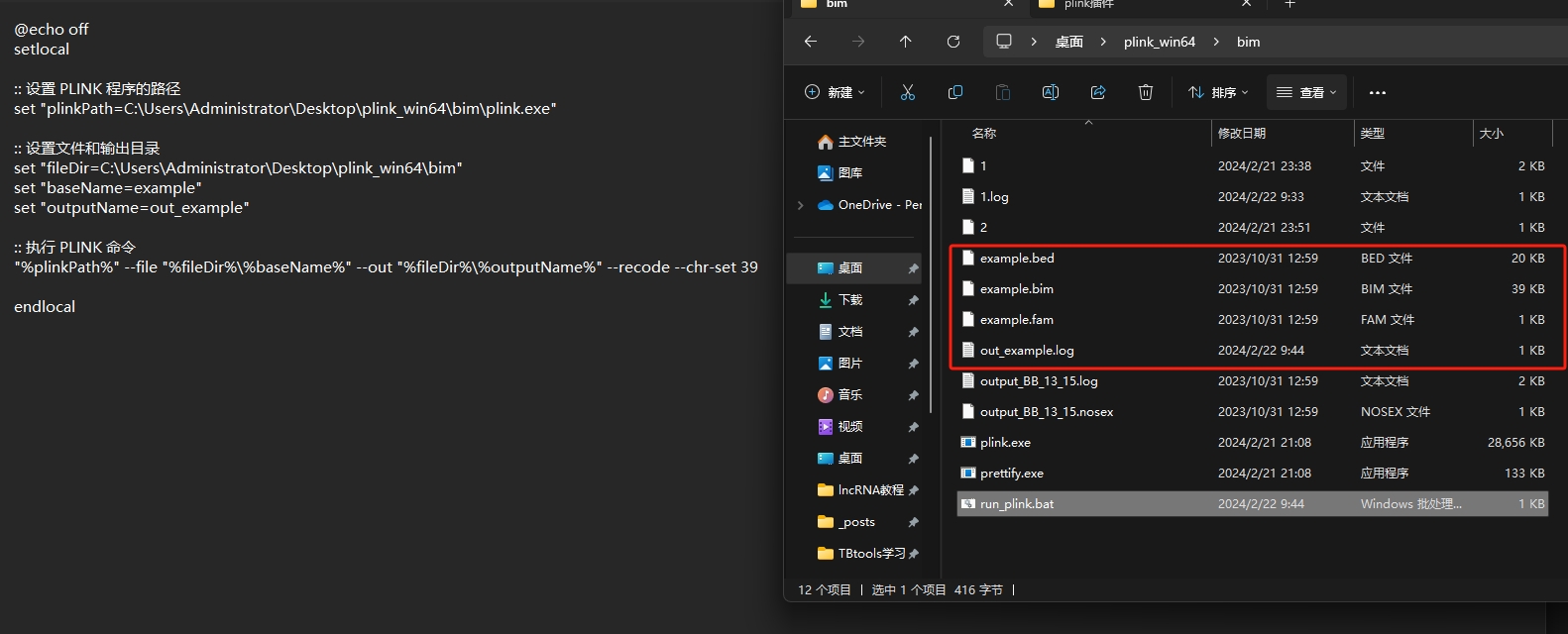
1 | @echo off |
但还要问题,
我的是一个插件,能够拖进去文件,我能拖进去C:\Users\Administrator\Desktop\plink_win64\1\example.bed,C:\Users\Administrator\Desktop\plink_win64\1\example.bim,C:\Users\Administrator\Desktop\plink_win64\1\example.fam
然后能能设置输出文件和其路径
C:\Users\Administrator\Desktop\plink_win64\1\1,我
1 |
|
@echo off命令用于关闭命令回显,这意味着在命令行执行时,命令本身不会被显示。setlocal命令开始一个局部环境的作用范围,使得脚本中设置的变量在脚本结束时不会影响到全局环境。
参数检查
1 | if "%~4"=="" ( |
- 这部分检查用户是否提供了足够的参数(至少4个)。如果没有,脚本将输出错误信息,显示正确的用法,然后退出。
提取文件目录和基本名
1 | for %%i in ("%~1") do ( |
- 这个循环使用批处理的参数扩展功能来从第一个参数(.bed文件的路径)提取文件的目录(
%%~dpi)和基本名(不包括扩展名,%%~ni)。
设置 PLINK 路径
1 | set "plinkPath=C:\Users\Administrator\Desktop\plink_win64\bim\plink.exe" |
- 这行命令设置了
plinkPath变量,指向PLINK可执行文件的路径。这个路径需要根据实际情况进行修改。
构建并执行 PLINK 命令
1 | "%plinkPath%" --bfile "%fileDir%%baseName%" --out "%~4" --recode --chr-set 39 |
- 这行命令实际上执行了PLINK程序。它使用
--bfile选项来指定输入文件(基于前面提取的目录和基本名),--out选项指定输出文件的前缀,--recode选项告诉PLINK需要重新编码输出数据,最后--chr-set 39指定了染色体集合的版本(这可能需要根据你的分析需求调整)。
脚本结尾
1 | endlocal |
endlocal命令结束局部变量的作用范围,这意味着在这之后设置的所有变量更改都不会影响到脚本外部的环境。
整个脚本的目的是为了简化PLINK命令的执行过程,使用户能够仅通过提供必要的文件路径和输出前缀就能自动化地运行PLINK进行数据分析。这对于需要处理多个数据集的用户来说尤其有用,因为它大大减少了重复劳动。
1 |
|
这将使脚本在当前文件夹内寻找 plink.exe 文件。
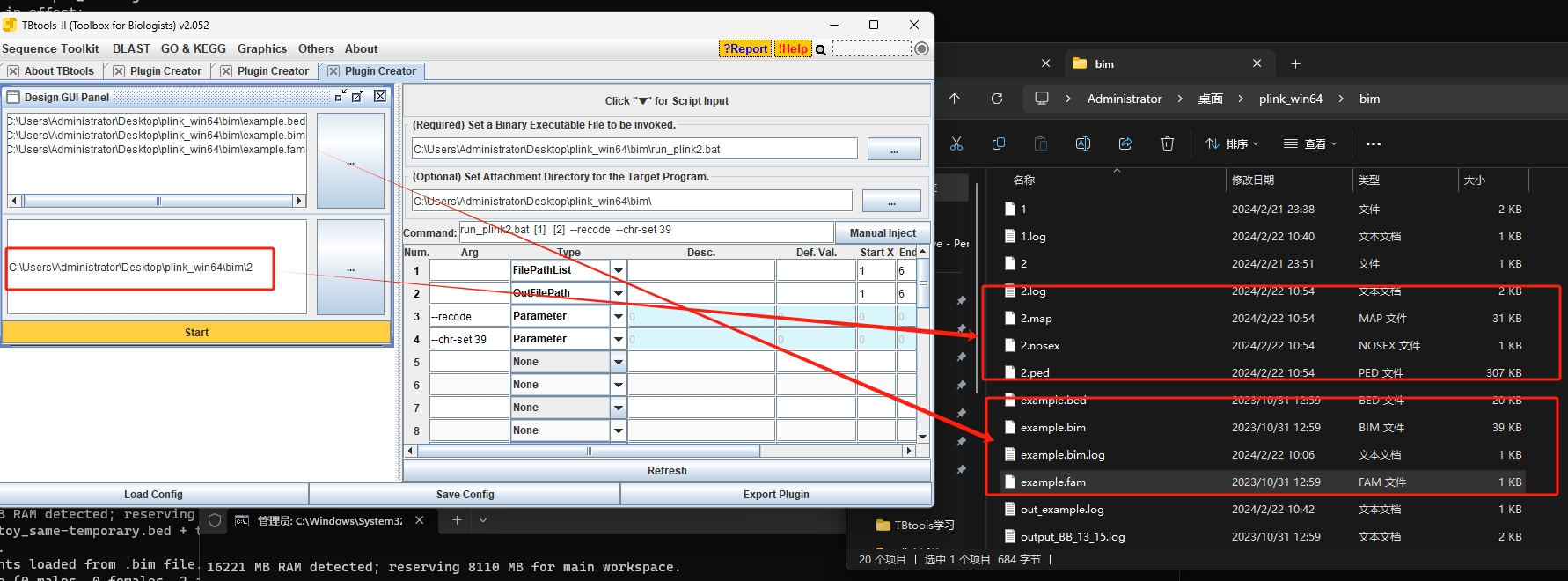
可以运行
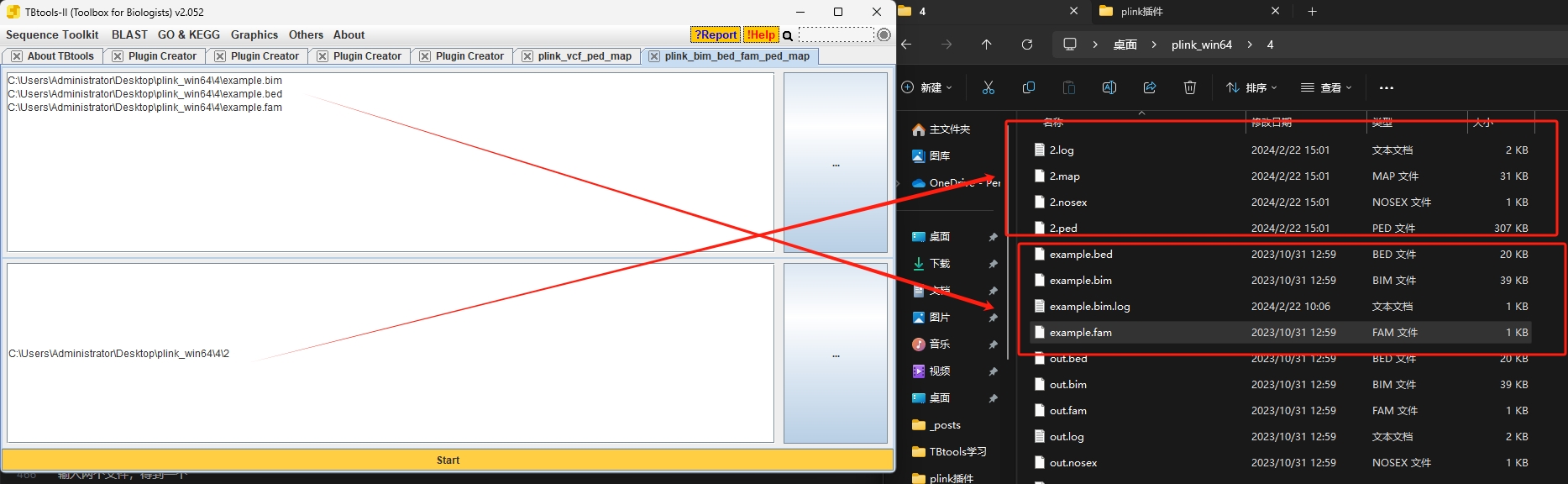
插件3,ped,map得到vcf
plink –file toy –recode vcf –out output_vcf –chr-set 39
输入两个文件,得到一个
1 |
|
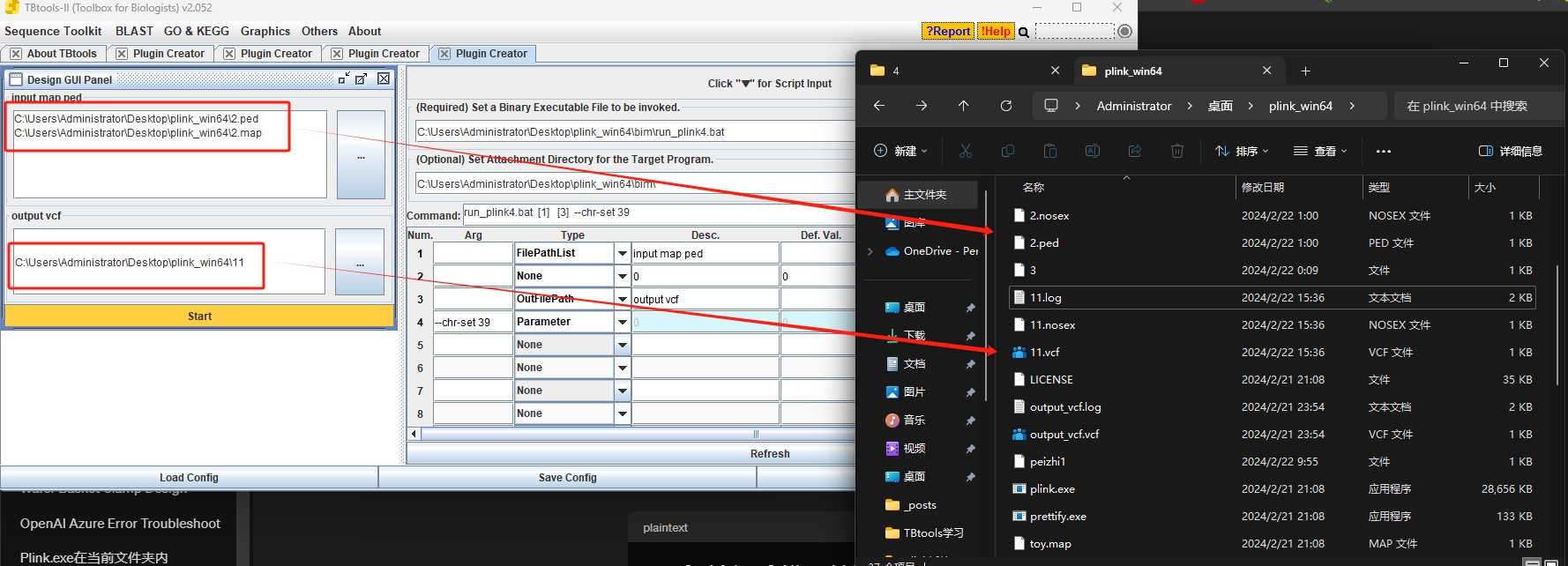
插件4,用plink代码在34号染色体,1339431-1343416位置进行筛选
plink –file BB –chr 34 –from-bp 1339431 –to-bp 1343416 –make-bed –out output_BB –chr-set 39
1 |
|
1 |
|
插件
1 | plink --bfile output_BB --pheno phenotypes.txt --allow-no-sex --logistic --adjust --output_choice_ logistic_results --chr-set 39 |
1 | @echo off |
这个命令将只分析第22号染色体上的SNP。
2. 使用性别作为协变量
如果您想在逻辑回归分析中考虑性别作为协变量,可以使用--covar参数指定一个包含性别信息的协变量文件:
1 | plink --bfile output_BB --pheno phenotypes.txt --covar covar.txt --allow-no-sex --logistic --adjust --covar-name sex --out logistic_results_with_sex --chr-set 39 |
这要求covar.txt文件中有一列名为sex,用于表示性别。
3. 分析特定的SNP群体
如果您只对一组特定的SNP感兴趣,可以使用--extract参数来指定一个包含这些SNP标识符的文件:
1 | plink --bfile output_BB --pheno phenotypes.txt --allow-no-sex --logistic --adjust --extract snp_list.txt --out logistic_results_specific_snps --chr-set 39 |
这要求snp_list.txt文件中包含您感兴趣的SNP的列表。
4. 调整分析模型
PLINK的--logistic命令支持多种模型调整选项,例如添加--dominant或--recessive来分别分析显性或隐性遗传效应:
1 | plink --bfile output_BB --pheno phenotypes.txt --allow-no-sex --logistic --adjust --dominant --out logistic_results_dominant --chr-set 39 |
或者
1 | plink --bfile output_BB --pheno phenotypes.txt --allow-no-sex --logistic --adjust --recessive --out logistic_results_recessive --chr-set 39 |
这些命令分别针对显性或隐性模型执行逻辑回归分析。
5. 控制族群结构和相关性
如果数据中包含族群结构或样本间存在相关性,可以使用--cluster或--pc参数来控制这些因素的影响:
1 | plink --bfile output_BB --pheno phenotypes.txt --allow-no-sex --logistic --adjust --cluster --out logistic_results_clustered --chr-set 39 |
或者,如果您已经从PCA分析中获得了主成分分数,可以使用它们作为协变量:
1 | plink --bfile output_BB --pheno phenotypes.txt --allow-no-sex --logistic --adjust --covar principal_components.txt --out logistic_results_with_pcs --chr-set 39 |
这些变体展示了PLINK命令行工具的灵活性和强大功能,允许研究人员根据具体的研究目的和数据特点定制分析策略。
1 |
|
在这个脚本中:
- 使用
echo命令显示提示信息,告诉用户需要输入什么。 - 使用
set /p命令接收用户的输入,每次接收一个参数。set /p param1=和set /p param2=分别用于接收两个不同的参数。 - 用户输入的参数随后被整合到 PLINK 命令中,作为命令的一部分执行。
这种方法让用户能够在批处理脚本执行过程中逐一输入多个参数,类似于逐个处理文件的方式。你可以根据需要添加更多的 set /p 语句来接收更多的输入。
空格问题
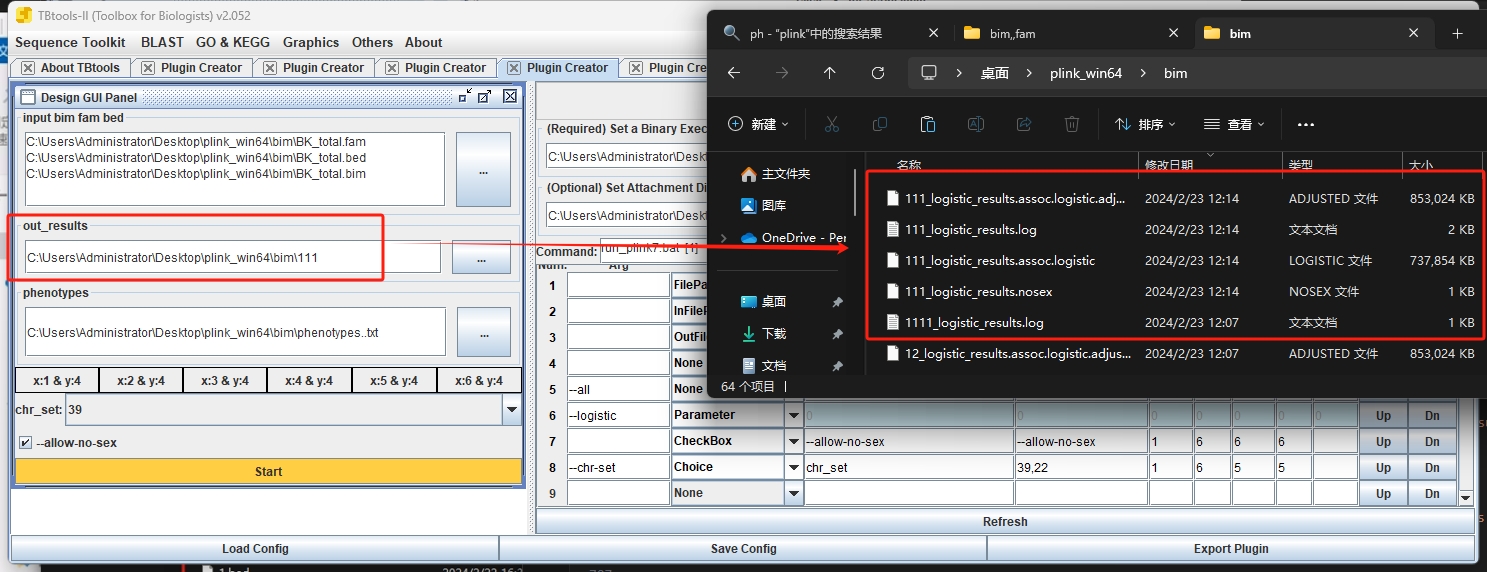
–allow-no-sex一输入进去前面会有两个空格



