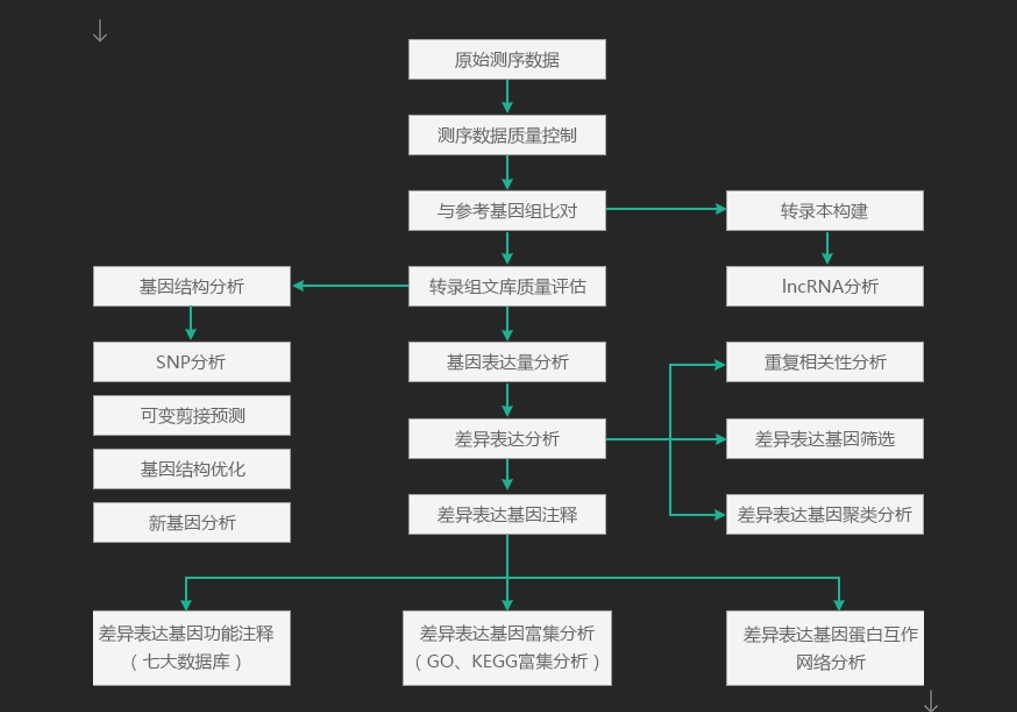
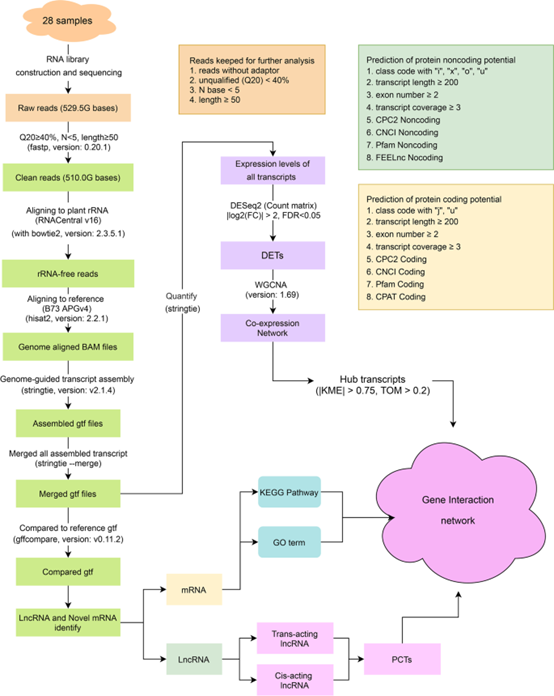
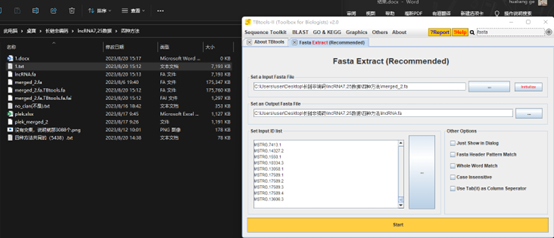
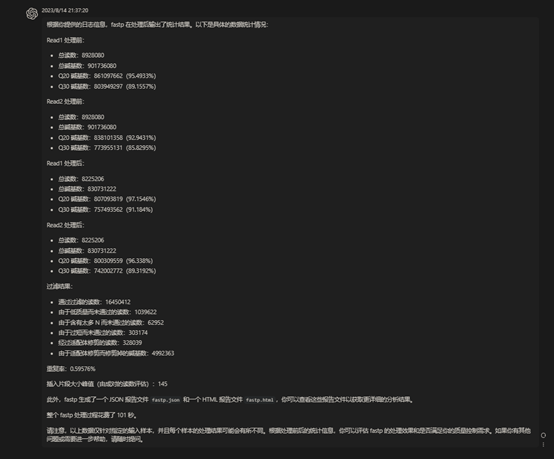
小知识点:(输入id和gtf。得到gtf文件)# 剩余的transcripts得到gtf
1 grep -w -Ff filter1_transcript_ID -w ~/lncRNA_project/06.gffcompare/merged.annotated.gtf > filter1_transcript.gtf
可能有用的网站 1,在鸡肌内前脂肪细胞分化过程中使用 RNA 测序分析长非编码 RNA 和 mRNA |公共图书馆一号 (plos.org)https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0172389
2,如何预测LncRNA靶基因(靶蛋白)? (sohu.com)lncRNA分析 - 简书 (jianshu.com)这个看着很厉害https://www.sohu.com/a/110662733_390793
https://www.jianshu.com/p/b6c40e9612c4
3,lncRNA分析完全指北 - 知乎 (zhihu.com)简单又详细的https://zhuanlan.zhihu.com/p/615591750
4,STEP8:鉴定全新的lncRNA - 简书 (jianshu.com)https://www.jianshu.com/p/5b104830751b
5,文献分享 | 长链非编码RNAs(lncRNAs)分析流程 | 转录组分析流程 - 知乎 (zhihu.com)https://www.zhihu.com/zvideo/1466176131120066560
6,GitHub - Alipe2021/NLncCirSmk:用于 lncRNA、环状 RNA 和新型 mRNA 鉴定的管道。https://github.com/Alipe2021/NLncCirSmk
7,小杜的生信筆記 - 知乎 (zhihu.com)可以询问他问题https://www.zhihu.com/people/xiaodu.com
8,转录组上游分析教程 | 课程介绍 - 知乎 (zhihu.com)付费教程,可能更全一点https://zhuanlan.zhihu.com/p/619830781
9,lncRNA鉴定方法整理 - 知乎 (zhihu.com)整理了很多方法,很有用https://zhuanlan.zhihu.com/p/612384333
10,如何快速从基因组中提取基因、转录本、蛋白、启动子、非编码序列? - 知乎 (zhihu.com)https://zhuanlan.zhihu.com/p/439168788
11,如何快速从基因组中提取基因、转录本、蛋白、启动子、非编码序列? - 知乎 (zhihu.com)https://zhuanlan.zhihu.com/p/439168788
12,长非编码RNA和mRNA的综合分析揭示了玉米幼苗根系对盐胁迫响应的调控网络 |BMC 基因组学 |全文 (biomedcentral.com)这个里面有个脚本https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-021-08286-7
13,RNA-seq:转录组数据分析处理(上)_super_qun的博客-CSDN博客https://blog.csdn.net/weixin_44452187/article/details/86646409?spm=1001.2101.3001.6650.2&utm_medium=distribute.pc_relevant.none-task-blog-2%7Edefault%7ECTRLIST%7ERate-2-86646409-blog-129909917.235%5Ev38%5Epc_relevant_sort_base1&depth_1-utm_source=distribute.pc_relevant.none-task-blog-2%7Edefault%7ECTRLIST%7ERate-2-86646409-blog-129909917.235%5Ev38%5Epc_relevant_sort_base1&utm_relevant_index=5
细致一点https://www.jianshu.com/p/15ef7df72391
15,4个发育时间点的总共12个鸡转录组测序样本的长非编码RNA的鉴定-腾讯云开发者社区-腾讯云 (tencent.com)https://cloud.tencent.com/developer/article/1842686
16,在鸡肌内前脂肪细胞分化过程中使用RNA测序分析长非编码RNA和mRNA - PubMed (nih.gov)https://pubmed.ncbi.nlm.nih.gov/28199418/
17,关于stringtie定量基因的时候,最后输出很多MSTRG样式的geneid - 简书 (jianshu.com)https://www.jianshu.com/p/1697f2d91eb5
18,「 bedtools 」提取上游+gene+下游序列 - 简书 (jianshu.com)https://www.jianshu.com/p/acb2bcb49882
https://www.jianshu.com/p/2b6aba1263d1
https://www.jianshu.com/p/2b6aba1263d1 https://www.jianshu.com/p/2b6aba1263d1
https://www.bioinfo-scrounger.com/archives/284/
20,转录组的组装Stingtie和Cufflinks | KeepNotes blog (bioinfo-scrounger.com)https://www.bioinfo-scrounger.com/archives/284/
21,RNA-seq入门实战(零):RNA-seq流程前的准备——Linux与R的环境创建 - 知乎 (zhihu.com)https://zhuanlan.zhihu.com/p/518132262
22,小L生信学习日记-3丨原始数据质量如何判断?-上 (qq.com)https://mp.weixin.qq.com/s?__biz=MzU3ODgxMDYyNw==&mid=2247483687&idx=1&sn=8689b12a0d30f90ae9ebe72182ee2899&chksm=fd6ee74bca196e5de8c17e762fb423c7e0cd24b8e8edb5fe4cf94ab061d166a10257447d3eb5&scene=21#wechat_redirect
23,小L生信学习日记-4丨原始数据质量如何判断?-下 - 知乎 (zhihu.com)https://zhuanlan.zhihu.com/p/57628300 https://mp.weixin.qq.com/s?search_click_id=2253394900344533860-1633341597788-628761&sub=&__biz=MzAxMDkxODM1Ng==&mid=2247503527&idx=4&sn=261be2c7ff2a7f80b2e6ab139028d780&chksm=9b4b8e1cac3c070a963f59fcc55095fe0ae37d1eecde3e67f6682779c91d0e907a8fd7eb8f84&scene=3&subscene=10000&clicktime=1633341597&enterid=1633341597&ascene=0&devicetype=android-30&version=28000f35&nettype=cmnet&abtest_cookie=AAACAA%3D%3D&lang=zh_CN&exportkey=AdXQxZkOzbrJK%2BUwprsoMEk%3D&pass_ticket=pvvLsQPcUT6hjrE3KSHbHTaZKL%2FVtNBVq%2BLbY9r0hucz%2FDdbU3NgO9ofB9mtC3fS&wx_header=1
25,基因组注释文件格式 –(一)BED文件格式 - 简书 (jianshu.com)https://www.jianshu.com/p/9208c3b89e44
26,一文搞懂NCBI Blast本地数据库(NT/NR等)构建 - 知乎 (zhihu.com)https://zhuanlan.zhihu.com/p/296579512
27,转录组测序(RNA-seq)的数据如何分析?_转录组数据怎么分析_砷在氟中的博客-CSDN博客https://www.jianshu.com/p/45fdb5cf930a
https://blog.csdn.net/weixin_55372631/article/details/129909917#:~:text=%E8%8B%A5%E6%A0%B7%E6%9C%AC%E6%95%B0%E7%9B%AE%E5%A4%A7%E4%BA%8E1,3%E4%B8%AA%E5%90%84%E5%85%B3%E9%94%AE%E5%9F%BA%E5%9B%A0%E3%80%82
28,在线工具】lncRNA编号XLOC_xxxxxx转换 - 简书 (jianshu.com)https://www.jianshu.com/p/44f8081e332d
29,stringtie merge与gene_id - 简书 (jianshu.com)https://www.jianshu.com/p/d828d45d8b6c
30,gffcompare的使用说明 - 简书 (jianshu.com)https://www.jianshu.com/p/08bb35f8e7c2
重要31.lncRNA分析https://www.jianshu.com/p/b6c40e9612c4
1 2 3 4 5 6 grep -Ff transcript_ID -w mergelist原来.gtf > lncRNA.gtf gffread protein_coding_gene.gtf -g /home/data1/Ghl/GHL/gtf/Gallus_gallus.GRCg6a.dna.toplevel.fa -w protein_coding_gene.fa gffread -w transcripts.fa -g Gallus_gallus.GRCg6a.dna.toplevel.fa merge_2.gtf
lncRNA组装流程的软件介绍之Stringtie (360doc.com)http://www.360doc.com/content/21/0714/12/76149697_986501836.shtml
非常详细介绍了转录本组装和定量,以及-e后不能筛选出新的
LncRNA的组装和鉴定(下游流程) (qq.com)https://mp.weixin.qq.com/s?__biz=MzAxMDkxODM1Ng==&mid=2247504567&idx=1&sn=3ddf2df1cd9554e2718de0abff04959e&chksm=9b4b920cac3c1b1a0ac7ae48b5de9802e7eeebf91a880fad9ffe13cd09132fb35a1405bd8d2f&cur_album_id=1322384987060142080&scene=189#wechat_redirect
jackhmmer search | HMMER (ebi.ac.uk)https://www.ebi.ac.uk/Tools/hmmer/search/jackhmmer
图:来源lncRNA测序分析优势及分析流程-百迈客生物 - 知乎 (zhihu.com)
没必要全部,选重要的
图:长非编码RNA和mRNA的综合分析揭示了玉米幼苗根系对盐胁迫响应的调控网络 |BMC 基因组学 |全文 (biomedcentral.com)
https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-021-08286-7
lncRNA检测工作流程,理论上的 从最开始的数据下载到最后筛选出lncRNA的完整工作流程涉及多个步骤。下面的指南提供了每一步的代码示例,但请注意,这些示例需要根据实际环境进行调整,包括文件路径、软件安装路径等。
步骤 1: 下载测序数据
下载SRA文件 。使用SRA Toolkit的prefetch工具下载SRA格式的测序数据文件。例如,下载样本SRR4046839:
转换SRA文件为FASTQ格式 。使用fastq-dump工具将SRA文件转换成FASTQ格式,便于后续分析。tbtools上有这个插件
1 fastq-dump --outdir /home/data1/Ghl/all --split-files SRR4046839
步骤 2: 质量控制
质量检查 。使用FastQC对FASTQ文件进行质量检查。
1 fastqc /home/data1/Ghl/all/SRR4046839_1.fastq /home/data1/Ghl/all/SRR4046839_2.fastq -o /home/data1/Ghl/fastqc_reports
质量修剪 。使用Trimmomatic对数据进行质量修剪和适配器序列的去除。
1 2 3 4 5 java -jar trimmomatic.jar PE -phred33 \ /home/data1/Ghl/all/SRR4046839_1.fastq /home/data1/Ghl/all/SRR4046839_2.fastq \ /home/data1/Ghl/trimmed/SRR4046839_1_paired.fastq /home/data1/Ghl/trimmed/SRR4046839_1_unpaired.fastq \ /home/data1/Ghl/trimmed/SRR4046839_2_paired.fastq /home/data1/Ghl/trimmed/SRR4046839_2_unpaired.fastq \ ILLUMINACLIP:TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
步骤 3: 序列映射
构建HISAT2索引 (如果尚未存在)。首先,为Gallus gallus参考基因组构建HISAT2索引。
1 hisat2-build Ghl/Gallus_gallus.GRCg6a.dna.toplevel.fa Gallus_gallus_GRCg6a_hisat2_index
使用HISAT2进行序列映射 。将预处理后的读取映射到参考基因组。应该也有tbtools插件
1 2 3 4 hisat2 -p 8 -x Gallus_gallus_GRCg6a_hisat2_index \ -1 /home/data1/Ghl/trimmed/SRR4046839_1_paired.fastq \ -2 /home/data1/Ghl/trimmed/SRR4046839_2_paired.fastq \ -S /home/data1/Ghl/hisat2_out/SRR4046839_aligned.sam
我的:
1 2 sudo hisat2 -p 16 --dta /home/dell/data/ghl/chicken/Gallus_index -1 /home/dell/data/ghl/SRR1_2/SRR6116749_1.fastq -2 /home/dell/data/ghl/SRR1_2/SRR6116749_2.fastq -S /home/dell/data/ghl/chicken/SRR6116749.sam
步骤 4: 转录本组装和定量
转换SAM为BAM格式 ,并对其进行排序。
1 samtools sort -o /home/data1/Ghl/hisat2_out/SRR4046839_aligned.sorted.bam /home/data1/Ghl/hisat2_out/SRR4046839_aligned.sam
我的:
1 2 3 sudo samtools view -@ 16 -bS /home/dell/data/ghl/chicken/SRR6116777.sam -o /home/data1/Ghl/bam/SRR6116777.bam samtools sort /home/data1/Ghl/bam/SRR6116777.bam -o /home/data1/Ghl/bam/SRR6116777.short.bam
使用StringTie进行转录本组装 。tbtools有插件
1 stringtie -p 8 -G Ghl/Gallus_gallus.GRCg6a.98.gtf -o /home/data1/Ghl/stringtie_out/SRR4046839.gtf /home/data1/Ghl/hisat2_out/SRR4046839_aligned.sorted.bam
我的:
1 2 3 4 5 6 7 8 stringtie SRR6116749.short.bam -o /home/data1/Ghl/TAB/SRR6116749.gtf -p 28 -G Gallus_gallus.GRCg6a.98.gtf -A /home/data1/Ghl/TAB/SRR6116749_gene_abund.tab -B -e(不是这个) sudo /usr/bin/stringtie -p 16 -G /home/dell/data/ghl/chicken/Gallus_gallus.GRCg6a.98.gtf -o /home/dell/data/ghl/chicken/Gallus00.gtf -l Gallus00 /home/dell/data/ghl/chicken/SRR9109597/Gallus00.short.bam sudo /usr/bin/stringtie -p 16 -G /home/data1/Ghl/lab4046839/Gallus_gallus.GRCg6a.98.gtf -o /home/data1/Ghl/GTF/SRR6116749.gtf -l Gallus00 /home/data1/Ghl/bam/SRR6116749.short.bam {% asset_img "stringtie.png" "stringTie" %}
步骤 5: lncRNA的筛选和注释,详细步骤在后面 命令 cuffcompare 是 Cufflinks 套件中的一个工具,用于比较多个转录组装文件(GTF/GFF格式)之间的差异,并且可以将转录本与参考注释进行比较。这个命令对于识别新的转录本、发现已知转录本的新变体,以及分类未知转录本非常有用。
让我们逐部分解析您提供的命令:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 cuffcompare -r Gallus_gallus.GRCg6a.98.gtf -o /home/data1/Ghl/lab4046839/cuffcompare mergelist.gtf mergelist.gtf是组装的文件,例如 stringtie --merge -p 8 \ -o stringtie_merge.gtf \ CK18.gtf T18.gtf CK69.gtf T69.gtf 运行 `cuffcompare` 命令后,通常会生成以下类型的文件: 1. **摘要统计文件 (`cuffcmp.stats`):** 包含比较结果的总体统计信息。 2. **参考注释文件 (`Gallus_gallus.GRCg6a.98.gtf`):** 你指定的参考注释文件。 3. **查询注释文件 (`mergelist.gtf`):** 你指定的查询注释文件。 4. **关系跟踪文件 (`cuffcmp.tracking`):** 详细描述了输入注释中转录本、基因等特征之间的关系,通常以表格形式呈现。 5. **参考映射文件 (`cmp.refmap`):** 显示输入注释中转录本与参考注释之间关系的映射文件。 6. **参考映射统计文件 (`refmap.stats`):** 总结输入注释与参考注释之间映射的统计信息。 7. **转录本映射文件 (`mergelist.tmap`):** 包含查询注释中转录本与参考注释之间的映射关系。 这些文件中的内容和格式会根据具体的 `cuffcompare` 命令参数和版本而有所不同。
上面代码解释
-r Gallus_gallus.GRCg6a.98.gtf: 这部分指定参考注释文件。-r 参数后面跟着的是参考基因组的注释文件(GTF格式),在这个例子中是 Gallus gallus(鸡)的参考注释,版本 GRCg6a.98。这个文件包含了鸡基因组的已知基因和转录本的位置、外显子边界等信息。
-o /home/data1/Ghl/lab4046839/cuffcompare: 这部分指定输出目录和输出文件的前缀。-o 参数后面跟着的是输出目录的路径,以及用于存放 cuffcompare 产生的所有输出文件的前缀。在这个例子中,所有的输出文件将被保存在 /home/data1/Ghl/lab4046839/cuffcompare 目录下,并且文件名将以 “cuffcompare” 为前缀。
mergelist.gtf: 这是输入文件,通常是由多个转录本组装结果文件(GTF格式)合并而成的文件,或者是单个转录本组装结果文件。cuffcompare 将会使用这个文件中的转录本信息,并将其与参考注释进行比较。
总结来说,这个命令的目的是将 mergelist.gtf 文件中的转录本与 Gallus gallus GRCg6a.98版本的参考注释进行比较。通过这种比较,您可以识别出新的转录本、未注释的基因,或者是与参考注释相比有差异的转录本。输出结果将包括一系列文件,例如统计信息、转录本分类汇总等,这些文件将提供关于比较结果的详细信息,有助于进一步的生物信息学分析和研究。
或者使用gffcompare
是的,gffcompare命令与cuffcompare命令的作用相似,它们都用于比较GTF/GFF文件中的转录本与参考注释之间的差异。gffcompare是cuffcompare的后继工具,提供了类似的功能,但通常认为在某些方面gffcompare更加灵活和高效。这两个工具都能帮助研究人员分析和理解转录本组装的结果,识别新的转录本,以及评估转录本组装的质量。
下面是gffcompare命令的详细解释:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 nohup gffcompare -R -r $gtf -o ./merged ../05.stringtie/02.merge_gtf/stringtie_merged.gtf > gffcompare.log 2>&1 &运行这个命令后,你会得到以下结果: 1. **REFMAP 文件 (`merged.refmap`)**:包含参考注释与预测转录本之间的映射关系。 2. **TRANSFRAG 文件 (`merged.annotated.gtf`)**:一个包含了所有输入注释文件中的所有转录本以及它们在参考基因组上的对应关系的 GTF 文件。 3. **Stats 文件 (`merged.stats`)**:包含了比较结果的统计信息。 4. **Log 文件 (`gffcompare.log`)**:包含了 `gffcompare` 命令的输出信息,包括程序运行时的任何警告或错误信息。 这些文件将保存在当前目录下,并且由于使用了 `nohup ` 命令,即使你关闭了终端,这些文件也会继续存在,直到该命令执行完毕。
nohup: 这是一个Unix命令,用于运行另一个命令,同时忽略所有挂起(hangup)信号。这意味着即使你关闭了终端或者断开了SSH连接,命令仍然在后台运行。
gffcompare: 这是执行比较的主命令,用于比较GTF/GFF文件。
-R: 这个选项告诉gffcompare在比较时考虑参考注释中的转录本重叠(包含在比较的转录本内的参考转录本)。
-r $gtf: -r选项后跟参考注释文件的路径。这里使用了变量$gtf来指定参考GTF文件的路径。$gtf应该在执行命令前定义,指向正确的参考注释文件。
-o ./merged: -o选项指定输出文件的前缀和输出目录。在这个例子中,所有的输出文件将被保存在当前目录下的merged前缀。
../05.stringtie/02.merge_gtf/stringtie_merged.gtf: 这是要比较的GTF文件,通常是使用StringTie或其他转录本组装工具生成的合并转录本文件。
> gffcompare.log 2>&1 &: 这部分将命令的标准输出(stdout)和标准错误(stderr)都重定向到gffcompare.log文件,并且在后台执行。这意味着你可以运行这个命令,然后继续在同一个终端中执行其他命令,而gffcompare的输出和错误都会被记录在日志文件中。
总的来说,这个gffcompare命令的作用是将stringtie_merged.gtf文件中的转录本与指定的参考注释进行比较,并且考虑转录本重叠情况,其输出结果将帮助识别新的或是未注释的转录本,以及提供转录本组装质量的评估。
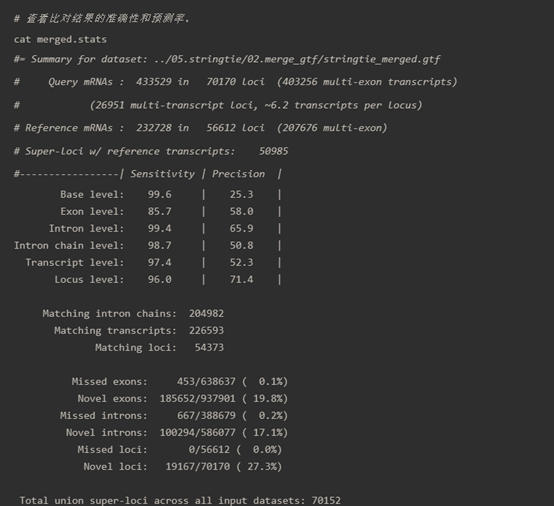
cat merged.stats
这个图像显示的是gffcompare工具生成的统计报告文件,通常命名为.stats,它提供了关于转录本组装质量和准确性的详细信息。报告分为几个部分,下面是对各部分内容的解释:
数据集摘要 :
Query mRNAs : 表示从输入数据集(如stringtie_merged.gtf)中识别出的mRNA转录本数量,以及这些转录本分布在的基因座位(loci)数量。Reference mRNAs : 表示在参考注释(如基因组注释文件)中的mRNA转录本数量和基因座位数量。
转录本级别的敏感度和精确度 :
Base level : 序列对比的基础级别,通常指核苷酸级别的准确性。Exon level : 外显子级别的对比,包括敏感度和精确度。敏感度指的是能够正确识别外显子的比例,而精确度指的是所有预测外显子中正确的比例。Intron level : 内含子级别的对比,同样包括敏感度和精确度。Intron chain level : 多个连续内含子的链的对比。Transcript level : 整个转录本级别的对比。Locus level : 基因座位级别的对比。
匹配和未匹配的统计 :
Matching intron chains : 正确匹配的内含子链的数量。Matching transcripts : 正确匹配的转录本数量。Matching loci : 正确匹配的基因座位数量。Missed exons : 未能检测到的外显子数量和总外显子数量的比例。Novel exons : 新发现的外显子数量和总外显子数量的比例。Missed introns : 未能检测到的内含子数量和总内含子数量的比例。Novel introns : 新发现的内含子数量和总内含子数量的比例。Missed loci : 未能检测到的基因座位数量和总基因座位数量的比例。Novel loci : 新发现的基因座位数量和总基因座位数量的比例。
总的合并基因座位数 :
这个报告对了解转录本组装的性能和准确性非常有用,可以帮助研究人员了解哪些区域被准确地重建,哪些区域可能需要进一步的验证或分析。敏感度(Sensitivity)是指正确识别的元素占所有应被识别元素的比例,而精确度(Precision)是指正确识别的元素占所有被识别元素的比例。通常,高敏感度和高精确度是期望达到的,但在实际应用中往往需要在二者之间寻求平衡。
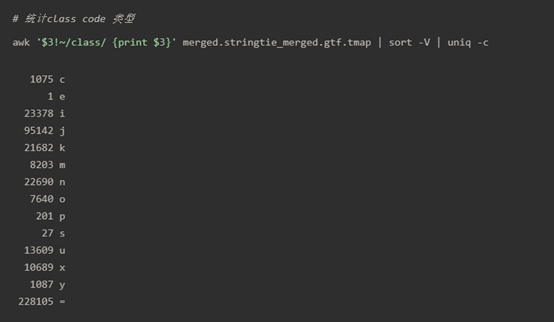
1 awk '$3!~/class/ {print $3}' merged.stringtie_merged.gtf.tmap | sort -V | uniq -c
接下来主要的操作对象是 merged.stringtie_merged.gtf.tmap 文件。
step1:保留指定class_code的transcripts 过滤,只保留class_code=”u”,”x”,”i”,”j”,”o”的 transcripts ,这个时候需要参考 stringtie官网提供的分类 :
图片是关于gffcompare或cuffcompare工具用于转录本分类的代码说明。这些代码帮助用户了解转录本相对于参考基因组注释的情况。每个代码代表了转录本与参考序列之间的特定关系。当gffcompare运行时,它会为每个转录本分配一个类别代码,表示其与参考基因组的对应关系。以下是您所询问的类别代码的解释:
u:未知的,基因间 。这个代码意味着转录本不能与参考基因组中的任何已知基因相匹配,通常位于已注释基因之间的区域。
x:异染色体上的外显子重叠 。指的是转录本的外显子与参考基因组上的外显子重叠,但是在异染色体上(如在X或Y染色体上的基因与常染色体上的基因重叠)。
i:内含子 。转录本完全位于一个已知基因的内含子区域内。
j:至少一个结合位点匹配的多外显子转录本 。这个代码表示转录本是多外显子的,并且至少有一个外显子的结合位点(即剪接位点)与参考基因组的外显子结合位点相匹配。
o:其他链上的外显子重叠 。转录本的外显子与参考基因组中的外显子在序列上重叠,但是位于反向链上。
这些类别代码可以帮助研究者识别潜在的新基因、非编码RNA或错误注释的基因。例如,分类为“u”的转录本可能代表未知的基因间区域的转录活动或假阳性信号;而分类为“i”的转录本可能指示内含子保留事件或潜在的新基因。
在gffcompare的输出中,每个转录本的类别代码将在其对应的行的属性字段中标注,这些信息通常被用来进一步筛选和验证实验数据。在后续的分析中,研究者可能会关注那些被标记为“u”或“x”的转录本,因为它们可能表示新的转录活动区域。
https://www.jianshu.com/p/2b6aba1263d1 重要
从组装开始,后续重要步骤
下载参考注释:Phytozome Sbicolor_454_v3.1.1.gene_exons.gff3.gz
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 gffread Sbicolor_454_v3.1.1.gene_exons.gff3.gz -T -o sbi311gx.gtf stringtie -p 16 -G ~/sbi311gx.gtf -o T18.gtf T18.bam stringtie -p 16 -G ~/sbi311gx.gtf -o CK18.gtf CK18.bam stringtie -p 16 -G ~/sbi311gx.gtf -o CK69.gtf CK69.bam stringtie -p 16 -G ~/sbi311gx.gtf -o T69.gtf T69.bam stringtie --merge -p 8 \ -o stringtie_merge.gtf \ CK18.gtf T18.gtf CK69.gtf T69.gtf stringtie -e -B -p 16 -G ~/4_stringtie/stringtie_merge.gtf -o T18.gtf ~/T18.bam Cuffcompare 将各样品的注释文件与参考基因组+参考注释比较,得到新转录本(lncRNA) 筛选条件: 1)FPKM>=0.5 2)coverage >1 3)Length > 200 4)分类为i j o u x 输入文件:cufcomp.CK18.gtf.tmap 等 结果文件:class.CK18等 (内容为注释,格式gtf) 提取序列 awk命令,比对转录本id 以class.CK18 从 CK18.gtf 中提取,文件名为CK18class.gtf gffread: 注释文件 、 参考基因组.fa 提取序列 结果文件:CK18class.fa (G:备份数据) cuffcompare -r ~/sbi311gx.gtf -s ~/Sbicolor_454_v3.0.1.fa -o cufcomp ../T18.gtf awk '{if($7>=0.5 && $10 > 1 && $11 >200) print $0}' cufcomp.T18.gtf.tmap > filter.T18 awk '{if ($3=="u" || $3=="x" || $3=="i" || $3=="j" || $3=="o"){print $0}}' filter.T18 > class.T18 $ wc -l class.T18 150458 class.T18 awk '{print $5}' class.T18 > filter1_transcript_ID $ wc -l filter1_transcript_ID 150458 filter1_transcript_ID awk 'NR==FNR{a[$5]=1} NR!=FNR{if(a[substr($12,2,length($12)-3)]==1) print $0}' class.T18 T18.gtf > T18class.gtf gffread T18class.gtf -g ~/Sbicolor_454_v3.0.1.fa -w T18class.fa
filter1_by_uxijo.tmap——filter1_transcript_ID—— filter1_transcript.gtf
筛选潜在的lncRNA转录本 。这可能需要基于转录本的特性(如长度、外显子数)以及编码潜
力(使用CPC2、CNCI等工具)进行筛选。
1 2 3 4 5 6 7 8 9 10 转录本编码能力预测,主要是4个软件,需要取交集: nohup cpat.py -x ../dat/Human_Hexamer.tsv \-d ../dat/Human_logitModel.RData \ -g ~/lncRNA_project/07.identification/step2/filter2_transcript_exon.fa \ -o ~/lncRNA_project/07.identification/step3/CPAT/cpat_result.txt > cpat.log 2>&1 & nohup Rscript ./LncFinder.R > LncFinder.log 2>&1 &
lnc_finder功能 - 研发 (rdocumentation.org)https://www.rdocumentation.org/packages/LncFinder/versions/1.1.5/topics/lnc_finder
lncRNA组装流程的软件介绍之lncFinder-腾讯云开发者社区-腾讯云 (tencent.com)
https://cloud.tencent.com/developer/article/1842771
官方https://cran.r-project.org/web/packages/LncFinder/index.html
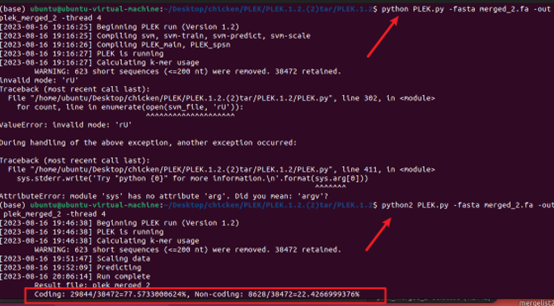
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 nohup python CNCI.py \-f ~/lncRNA_project/07.identification/step2/filter2_transcript_exon.fa \ -o ~/lncRNA_project/07.identification/step3/CNCI \ -m ve \ -p 4 > cnci.log 2>&1 & nohup python PLEK.py \-fasta ~/lncRNA_project/07.identification/step2/filter2_transcript_exon.fa \ -out ~/lncRNA_project/07.identification/step3/plek/plek \ -thread 4 > plek.log 2>&1 & 可能是这个原因吧 PLek下载 https://sourceforge.net/projects/plek/ python2 PLEK.py -fasta filter2_transcript_exon.fa -out plek -thread 4 改下 python PLEK.py -fasta merged_2.fa -out plek_merged_2 -thread 4
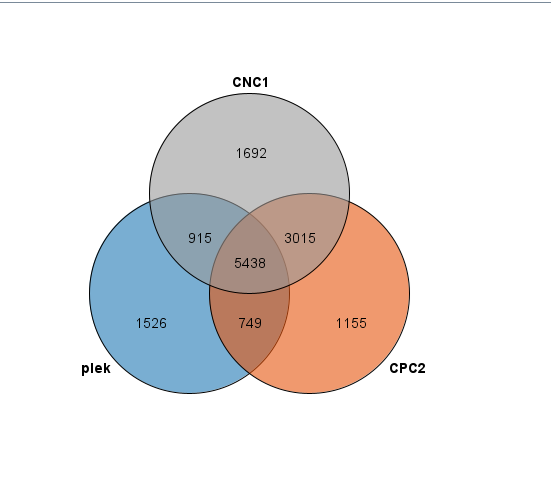
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 less CPC2_result.txt|grep 'noncoding' |awk '{print $1}' > CPC2_id.txt $ wc -l CPC2_id.txt 55359 CPC2_id.txt less -S cpat_result.txt|awk '($6<0.364){print $1}' > cpat_id.txt sed -i '1d' file $ wc -l cpat_id.txt 51956 cpat_id.txt less lncFinder_result.txt|grep -w 'NonCoding' > lncFinder_id.txt $ wc -l lncFinder_id.txt 52442 lncFinder_id.txt less -S plek | grep -w 'Non-coding' |awk '{print $3}' |sed 's/>//g' > plek_id.txt $ wc -l plek_id.txt 55996 plek_id.txt cat *txt |sort |uniq -c |awk '{if( $1==4){print}}' |wc {% asset_img "4个软件取交集.png" "4个软件的交集" %} CNCI 一共39060筛选出11059个nocoding CPC2 一个39154筛选出11356个nocoding PLEK 一个20935筛选出4097个nocoding
最终筛选出21872个codeing RNA和4932个 nocoding
最后得到的为lncRNA.fa
step4:比对到Pfam据库(我觉得filter3_by_noncoding_exon.fa可能是上面的交集的id转换来的)
比对到Pfam据库,过滤 (E-value < 1e-5)
pfem使用Transeq 将转录本序列翻译为6个可能的蛋白序列 transeq ../step3/intersection/filter3_by_noncoding_exon.fa filter4_protein.fa -frame=6
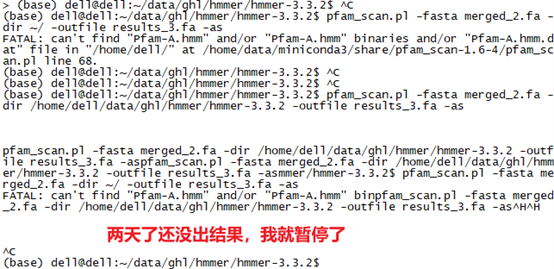
conda install pfam_scan
nohup pfam_scan.pl -fasta ./filter4_protein.fa -dir /home/data/lihe/database/Pfam/ -out Pfam_scan.out > Pfam_scan.log 2>&1 &
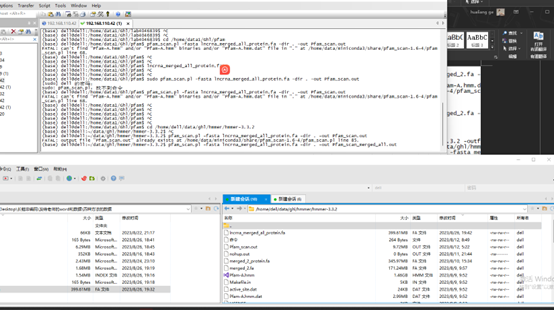
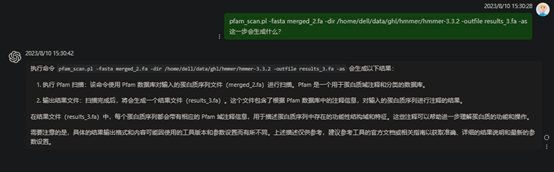
pfam_scan.pl -fasta merged_2.fa -dir /home/dell/data/ghl/hmmer/hmmer-3.3.2 -outfile results_3.fa -as
transeq ../step3/merged_2.fa merged_2_protein.fa -frame=6
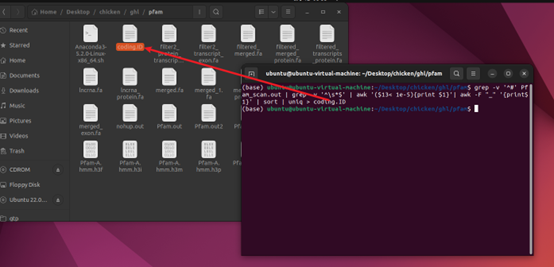
过滤 (E-value < 1e-5) grep -v ‘^#’ Pfam_scan.out | grep -v ‘^\s*$’ | awk ‘($13< 1e-5){print $1}’| awk -F “_” ‘{print$1}’ | sort | uniq > coding.ID
wc coding.ID
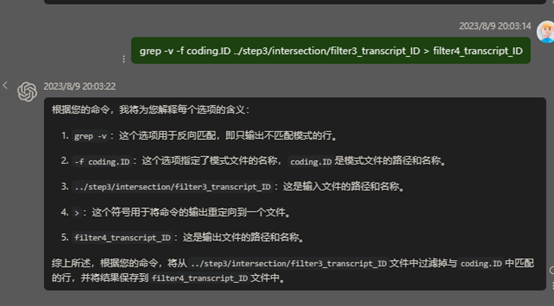
反向过滤 grep -v -f coding.ID ../step3/intersection/filter3_transcript_ID > filter4_transcript_ID
dos2unix filter4_transcript_ID.txt
grep -Ff transcript_ID -w Gallus_gallus.GRCg6a.98.gtf > lncRNA.gtf
注释和进一步的分析 。将筛选出的lncRNA候选与已知的lncRNA数据库进行比对,如NONCODE或ALDB,以确定新发现的lncRNA。
这个过程可能涉及到多个自定义脚本和具体的分析策略,具体取决于您的研究目标和可用资源。
注意:
上述步骤中的文件路径、索引名称和软件参数都需要根据您的实际环境进行调整。
某些步骤(如lncRNA的筛选和注释)需要您根据具体的研究需求来定制分析策略和使用相应的工具。
确保所有使用的工具都已正确安装,并且您有适当的权限执行这些命令。
第二种,lncRNA检测工作流程 测序数据分析 干净数据使用Tophat2 [19]程序按照以下参数映射到Gallus gallus参考基因组(gal6) 长非编码RNA (lncRNA) 的鉴定与注释在RNA-seq数据分析中是至关重要的一环,它分为两大部分:一个是处理通用RNA-seq数据,另一个是专门针对lncRNA的注释和分析。
0,前期准备: 1 2 3 4 5 6 下载方法 prefetch SRR4046839 fastq-dump --outdir /home/data1/Ghl/all --split-files SRR6116749.sra
通用RNA-seq数据处理流程: 1,质量控制:使用FastQC软件来生成初始序列数据的质量报告。 按照3.0 转录本比对组装HISAT2+StringTie+cuffcompare - 简书 (jianshu.com)进行质控https://www.jianshu.com/p/2b6aba1263d1
1 2 3 4 5 6 7 fastp --detect_adapter_for_pe -l 100 -q 19 -u 50 -i ~/CK18_1.fq.gz -o CK18c_1.fq.gz -I ~/CK18_2.fq.gz -O CK18c_2.fq.gz \ fastp --detect_adapter_for_pe -l 100 -q 19 -u 50 -i /home/dell/data/ghl/SRR1_2/SRR6116775_1.fastq -o /home/dell/data/ghl/SRR1_2/CK18c1_1.fastq \ -I /home/dell/data/ghl/SRR1_2/SRR6116775_2.fastq -O /home/dell/data/ghl/SRR1_2/CK18c1_2.fastq \ -f 5
步骤:
下面部分是我修改的代码
1 2 fastp --detect_adapter_for_pe -l 100 -q 19 -u 50 -i /home/dell/data/ghl/SRR1_2/SRR6116775_1.fastq -o /home/dell/data/ghl/SRR1_2/CK18c_1.fastq \ -I /home/dell/data/ghl/SRR1_2/SRR6116775_2.fastq -O /home/dell/data/ghl/SRR1_2/CK18c_2.fastq
数据过滤(这个要做吗)
#工具:选一种即可
1 2 3 4 5 6 7 8 9 conda install -c trimmomatic conda install fastp fastp --detect_adapter_for_pe -l 100 -q 19 -u 50 -i ~/CK18_1.fq.gz -o CK18c_1.fq.gz -I ~/CK18_2.fq.gz -O CK18c_2.fq.gz \ trimmomatic PE -phred33 CK18_1.fq.gz CK18_2.fq.gz CK18_1paired.fq.gz CK18_1unpaired.fq.gz CK18_2paired.fq.gz CK18_2unpaired.fq.gz ILLUMINACLIP:TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:19 MINLEN:50 fastqc -o ./ -t 4 ./ CK18c_11.fq.gz
2,序列预处理:借助Trimmomatic工具[20]对原始读取进行清洗,这包括移除来自测序适配器的污染物和修剪低质量的读取。常规的读取质量分数阈值是20。 3,序列映射:使用如Tophat2 [19]、Bowtie2 [21]或HISAT2 [22]等工具将预处理后的读取映射到参考基因组。 4,转录本组装:通常使用Cufflinks [23]或StringTie [24]来进行序列读取的组装。 cuffcompare -r ~/sbi311gx.gtf -s ~/Sbicolor_454_v3.0.1.fa -o cufcomp ../T18.gtf
awk ‘{if($7>=0.5 && $10 > 1 && $11 >200) print $0}’ cufcomp.T18.gtf.tmap > filter.T18
awk ‘{if ($3==”u” || $3==”x” || $3==”i” || $3==”j” || $3==”o”){print $0}}’ filter.T18 > class.T18
awk ‘NR==FNR{a[$5]=1} NR!=FNR{if(a[substr($12,2,length($12)-3)]==1) print $0}’ class.T18 T18.gtf > T18class.gtf
#从参考基因组中提取序列
gffread T18class.gtf -g ~/Sbicolor_454_v3.0.1.fa -w T18class.fa
最后的
到目前为止没有问题
(6116778.TSRR6116778.gtf.tmap– filter.6116778)
4,筛选tmap(filter.6116749– class.T6116749)
5,又得到gtf(TSRR6116749.gtf与class.T6116749– T6116749class.gtf)
下面这个代码的运行后
awk ‘{if ($3==”u” || $3==”x” || $3==”i” || $3==”j” || $3==”o”){print $0}}’ filter.6116778 > class.T6116778
又是每一个都保存
6,得到fa文件(T6116749class.gtf- T6116749class.fa)
7,
专门针对lncRNA的分析流程:
我的批量运行的代码,有的在服务器上,以后补充 第九步9_stringtie_batch.sh.txt 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 declare -a SRR_ids=("SRR6116749" "SRR6116750" "SRR6116751" "SRR6116752" "SRR6116753" "SRR6116754" "SRR6116761" "SRR6116762" "SRR6116763" "SRR6116764" "SRR6116765" "SRR6116766" "SRR6116773" "SRR6116774" "SRR6116775" "SRR6116776" "SRR6116777" "SRR6116778" ) MERGED_GTF="/path_to/mergelist.gtf" BAM_DIR="/home/data1/Ghl/GHL/short_bam" OUT_DIR="/home/data1/Ghl/GHL/_e_gtf" mkdir -p "${OUT_DIR} " for srr in "${SRR_ids[@]} " do echo "Processing ${srr} with StringTie..." stringtie -e -B -p 16 -G "${MERGED_GTF} " -o "${OUT_DIR} /e_${srr} .gtf" "${BAM_DIR} /${srr} .short.bam" done echo "StringTie processing done for all samples!"
第10步,10_cuffcompare.sh.txt 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 #!/bin/bash declare -a SRR_ids=("SRR6116749" "SRR6116750" "SRR6116751" "SRR6116752" "SRR6116753" "SRR6116754" "SRR6116761" "SRR6116762" "SRR6116763" "SRR6116764" "SRR6116765" "SRR6116766" "SRR6116773" "SRR6116774" "SRR6116775" "SRR6116776" "SRR6116777" "SRR6116778" ) REF_GTF="/home/data1/Ghl/GHL/Gallus_gallus.GRCg6a.98.gtf" REF_SEQ="/home/data1/Ghl/GHL/Gallus_gallus.GRCg6a.dna.toplevel.fa" INPUT_DIR="/home/data1/Ghl/GHL/_e_gtf" OUTPUT_DIR="/home/data1/Ghl/GHL/10_cuffcompare" mkdir -p "${OUTPUT_DIR} " for srr in "${SRR_ids[@]} " do SRR_OUT_DIR="${OUTPUT_DIR} /${srr} " mkdir -p "${SRR_OUT_DIR} " echo "Running cuffcompare for ${srr} ..." cuffcompare -r "${REF_GTF} " -s "${REF_SEQ} " -o "${SRR_OUT_DIR} /${srr} " "${INPUT_DIR} /${srr} /${srr} .gtf" done echo "Cuffcompare processing done for all samples!"
11 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 declare -a SRR_ids=("SRR6116749" "SRR6116750" "SRR6116751" "SRR6116752" "SRR6116753" "SRR6116754" "SRR6116761" "SRR6116762" "SRR6116763" "SRR6116764" "SRR6116765" "SRR6116766" "SRR6116773" "SRR6116774" "SRR6116775" "SRR6116776" "SRR6116777" "SRR6116778" ) BASE_GTF_DIR="/home/data1/Ghl/GHL/_e_gtf" BASE_OUT_DIR="/home/data1/Ghl/GHL/11_gffread" REF_SEQ="~/Sbicolor_454_v3.0.1.fa" mkdir -p "${BASE_OUT_DIR} " for srr in "${SRR_ids[@]} " do echo "Processing ${srr} ..." TMAP_FILE="${BASE_GTF_DIR} /${srr} /${srr} .gtf.tmap" GTF_FILE="${BASE_GTF_DIR} /${srr} /${srr} .gtf" PREFIX="${BASE_OUT_DIR} /${srr} " awk '{if($7>=0.5 && $10 > 1 && $11 >200) print $0}' "${TMAP_FILE} " > "${PREFIX} _filter.T18" awk '{if ($3=="u" || $3=="x" || $3=="i" || $3=="j" || $3=="o"){print $0}}' "${PREFIX} _filter.T18" > "${PREFIX} _class.T18" awk 'NR==FNR{a[$5]=1} NR!=FNR{if(a[substr($12,2,length($12)-3)]==1) print $0}' "${PREFIX} _class.T18" "${GTF_FILE} " > "${PREFIX} _T18class.gtf" gffread "${PREFIX} _T18class.gtf" -g "${REF_SEQ} " -w "${PREFIX} _T18class.fa" echo "Processing for ${srr} done!" done echo "All processing done!"
12_cpc2.sh(1).txt 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 INPUT_DIR="/home/data1/Ghl/GHL/11_gffread" OUTPUT_DIR="/home/data1/Ghl/GHL/CPC_CNCI_CPAT/cpc2" mkdir -p "${OUTPUT_DIR} " for input_file in ${INPUT_DIR} /*_Tclass.fa; do base_filename=$(basename "${input_file} " _Tclass.fa) output_file="${OUTPUT_DIR} /${base_filename} _cpc2_output.txt" python3 CPC2.py -i "${input_file} " -o "${output_file} " echo "CPC2 analysis done for ${base_filename} " done echo "All CPC2 processing done!"
把所有转录本注释分类为mRNA\lncRNA、新的鉴定的lncRNA https://www.jianshu.com/p/b6c40e9612c4
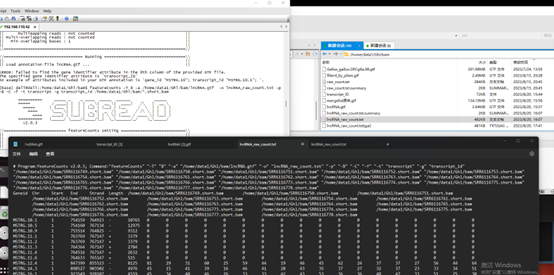
WGCNA count 图:featureCounts -T 8 -a /home/data1/Ghl/bam/lncRNA.gtf -o lncRNA_raw_count.txt -p -B -C -f -t transcript -g transcript_id /home/data1/Ghl/bam/*.short.bam
您提供的命令是使用 featureCounts 工具来计算基因表达量,特别是在这个上下文中,它被用来对长非编码RNA(lncRNA)进行计数。featureCounts 是 Subread 软件包的一部分,广泛用于对RNA-Seq数据进行定量分析。
下面是该命令中每个参数的解释:
featureCounts: 这是执行定量的主命令。
-T 8: 指定要使用的线程数,这里设置为8,意味着featureCounts会并行运行以加快处理速度。
-a /home/data1/Ghl/bam/lncRNA.gtf: -a 参数后跟注释文件的路径,这里指定了包含lncRNA注释信息的GTF文件。
-o lncRNA_raw_count.txt: -o 参数指定输出文件的名称,这里的输出文件将包含原始的计数数据。
-p: 这个参数告诉 featureCounts 输入的是配对端测序数据。
-B: 要求计数时只考虑每对reads中都成功比对的情况。
-C: 不计算跨越基因间区域的read对。
-f: 使用-f参数意味着计数是基于特征(例如外显子)而不是基于元注释的(如转录本或基因),这对于lncRNA这样的非编码RNA特别有用。
-t transcript: -t 参数指定计数时考虑的注释特征类型,这里设置为“transcript”,意味着计数将基于转录本进行。
-g transcript_id: -g 参数指定用于分组的特征属性,这里使用“transcript_id”表示计数将根据每个转录本的ID来汇总。
/home/data1/Ghl/bam/*.short.bam: 这是输入文件的路径,*.short.bam 表示该命令将处理指定路径下所有以.short.bam结尾的文件。
综上所述,这个 featureCounts 命令将对所有在指定路径下的BAM文件中的lncRNA进行计数,使用多线程来加速处理,并将结果输出到一个文本文件中。这个结果文件可以用于后续的差异表达分析或其他类型的生物信息学分析。
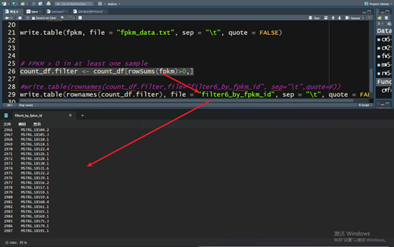
FPKEM 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 2,R代码 rm (list=ls ())raw_df <- read.table(file = "lncRNA_raw_count.txt" ,header = T,skip = 1,sep = "\t" ) count_df <- raw_df[ ,c(7:ncol(raw_df))] metadata <- raw_df[ ,1:6] rownames(count_df) <- as.character(raw_df[,1]) colnames(count_df) <- paste0("SRR10744" ,1:18) countToFpkm <- function (counts, effLen) { N <- colSums(counts) exp( log (counts) + log (1e9) - log (effLen) - log (N) ) } options(scipen = 200) fpkm = countToFpkm(count_df, metadata$Length ) write.table(fpkm, file = "fpkm_data.txt" , sep = "\t" , quote = FALSE) count_df.filter <- count_df[rowSums(fpkm)>0,] write.table(rownames(count_df.filter), file = "filter6_by_fpkm_id" , sep = "\t" , quote = FALSE)
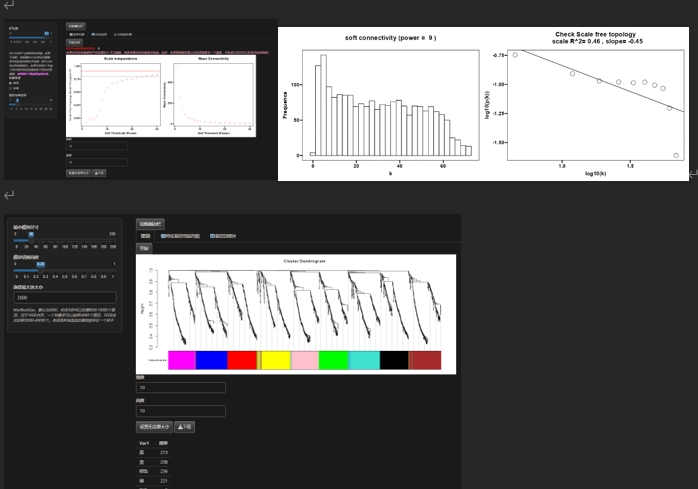
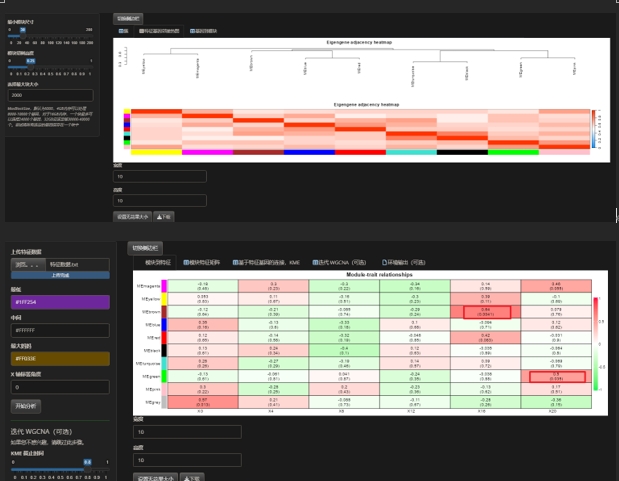
得到fpkm_data.txt,进行WGCNA 把txt,转为xlsx,在转txt后没报错了
一共fpkm_data.txt有5439行,但很多是零,大概删去0的,有2987个
WGCNA筛选2000个
lncRNA分析,第三种 https://www.jianshu.com/p/b6c40e9612c4
为了处理和压缩高通量测序数据,您可以按照以下步骤操作:
在您提到的场景中,下载数据(如SRR4046839)通常是使用NCBI的SRA Toolkit中的prefetch命令的第一步。在此之后,您需要通过多个步骤来处理和分析这些数据,直至达到上述步骤中提到的质控和数据清洗。下面是从下载SRR4046839到执行质控脚本的完整步骤:
1. 下载数据 使用prefetch命令从NCBI下载SRA文件:
这个命令会下载SRR4046839的SRA文件到本地。
2. 转换SRA文件到FASTQ格式 下载完SRA文件后,使用fastq-dump(SRA Toolkit的一部分)或更现代的工具如fasterq-dump来转换SRA文件为FASTQ格式。fasterq-dump比fastq-dump更快且更高效:
1 fasterq-dump SRR4046839 --split-files -O /path/to/output/directory
这个命令会生成两个FASTQ文件(如果是双端数据的话),分别对应每个读段的数据,例如SRR4046839_1.fastq和SRR4046839_2.fastq,并将这些文件保存到指定的输出目录中。
3. 准备质控分析 创建数据目录 创建一个存储清洗后数据的目录:
生成文件列表,等等就可以了 根据下载并转换的FASTQ文件位置,使用ls和paste命令生成包含读段文件路径的列表:
1 2 3 ls /path/to/output/directory/*.R1.fastq.gz > rawdata_fastq_1ls /path/to/output/directory/*.R2.fastq.gz > rawdata_fastq_2paste rawdata_fastq_1 rawdata_fastq_2 > rawdata_fastq
请将/path/to/output/directory/替换为实际存放SRR4046839_1.fastq和SRR4046839_2.fastq文件的目录路径。
4,合并FASTQ文件
合并R1读取文件 :使用cat命令将两个或多个R1读取的补测数据文件合并成一个文件。1 cat V1-2_L4_160A60.R1.fastq1 V1-2_L4_160A60.R1.fastq2 > V1-2_L4_160A60.R1.fastq
合并R2读取文件 :同样地,使用cat命令合并R2读取的补测数据文件。1 cat V1-2_L4_160A60.R2.fastq1 V1-2_L4_160A60.R2.fastq2 > V1-2_L4_160A60.R2.fastq
对其他样本重复以上步骤。
5,压缩合并后的文件
压缩新合并的文件 :使用gzip命令压缩合并后的文件以节省存储空间。1 2 gzip V1-2_L4_160A60.R1.fastq gzip V1-2_L4_160A60.R2.fastq
对所有合并后的文件重复此步骤。
6,重命名和压缩原始文件
重命名原始压缩文件 :使用mv命令简化文件名。1 mv V0-1_L4_157A57.R1.fastq.gz V0-1.R1.fastq.gz
对所有需要重命名的文件重复此步骤。
(可选)压缩重命名的文件 :如果重命名的文件未被压缩(适用于未压缩的文件),使用gzip进行压缩。
请注意,上述第5步仅适用于未压缩的文件。如果文件已经以.gz结尾,说明它们已经被压缩,无需再次压缩。
以上步骤将帮助您有效地管理和存储高通量测序数据。
7,rawdata质控,或者其他质控方法 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 mkdir cleandatals `pwd `/*.R1.fastq.gz > rawdata_fastq_1ls `pwd `/*.R2.fastq.gz > rawdata_fastq_2paste rawdata_fastq_1 rawdata_fastq_2> rawdata_fastq------------------------------------------------------------------------ vim qc.sh cat rawdata_fastq|while read id do arr=(${id} ) fq1=${arr[0]} fq2=${arr[1]} trim_galore -q 25 --phred33 --length 36 -e 0.1 --stringency 3 --paired -o cleandata/ $fq1 $fq2 & done chmod a+x qc.shnohup ./qc.sh
这段代码是用于在生物信息学分析中进行序列数据的预处理,特别是用于质量控制和修剪原始的FASTQ格式的测序数据。这些操作通常是高通量测序数据分析流程的初步步骤,用以确保数据的质量足够好,适合进行后续的分析,如比对、变异检测等。下面是代码的逐步解释:
创建目录和准备文件列表
mkdir cleandata:创建一个名为cleandata的目录,用于存放处理后的数据。
ls $(pwd)/*.R1.fastq.gz > rawdata_fastq_1:列出当前目录下所有以.R1.fastq.gz结尾的文件(代表配对测序的第一个文件),并将这些文件的完整路径保存到rawdata_fastq_1文件中。
ls $(pwd)/*.R2.fastq.gz > rawdata_fastq_2:同样,列出所有以.R2.fastq.gz结尾的文件(配对测序的第二个文件),并将路径保存到rawdata_fastq_2文件中。
paste rawdata_fastq_1 rawdata_fastq_2 > rawdata_fastq:将rawdata_fastq_1和rawdata_fastq_2文件中的内容合并(每行对应一个配对的样本),每对文件的路径由制表符分隔,结果保存到rawdata_fastq文件中。
编写和执行质量控制脚本
vim qc.sh:使用vim编辑器创建一个名为qc.sh的脚本文件。此步骤需要手动输入脚本内容,脚本的具体内容在下一部分解释。
脚本内容解释:
读取rawdata_fastq文件中的每一行,每行包含一对配对的FASTQ文件路径。
使用trim_galore命令对每对FASTQ文件进行质量控制和修剪。trim_galore是一个基于Cutadapt和FastQC的自动化质控工具,用于自动检测质量门槛、移除低质量的序列和adapter序列。
参数解释:
-q 25:设置质量分数门槛为25。--phred33:指定质量分数使用Phred+33编码。--length 36:移除修剪后长度小于36的读取。-e 0.1:允许的最大误差率为0.1。--stringency 3:在adapter修剪中,重叠长度需至少为3。--paired:指定输入文件为配对的。-o cleandata/:指定输出目录为cleandata。
&:在后台执行每个trim_galore命令,允许同时处理多个文件对。
chmod a+x qc.sh:给予qc.sh脚本执行权限。
nohup ./qc.sh &:在后台执行qc.sh脚本,并通过nohup命令确保即使关闭终端会话,脚本也会继续运行。
这个流程是高通量测序数据分析的常规质量控制步骤,旨在提高数据质量,为后续分析提供更准确的输入数据。
8,统计cleandatabase 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 mkdir cleandataBasels `pwd `/*.R1_val_1.fq.gz > cleandata_fastq_1ls `pwd `/*.R2_val_2.fq.gz > cleandata_fastq_2find *_1.fq.gz >sample sed -i "s/.R1_val_1.fq.gz/ /g" sample paste cleandata_fastq_1 cleandata_fastq_2 sample> cleandata_fastqvim fc.sh cat cleandata_fastq|while read id do arr=(${id} ) fq1=${arr[0]} fq2=${arr[1]} sample=${arr[2]} fastq-count $fq1 $fq2 >cleandataBase/$sample & done chmod a+x fc.shnohup ./fc.shcd cleandataBasecat * > cleandataBasesed -i 's/}/ /g' cleandataBase sed -i 's/:/\t/g' cleandataBase sed -i 's/,/\t/g' cleandataBase cat cleandataBase | awk '{print $2}' > cleandatabasescat cleandataBase | awk '{print $4}' > cleandatareadscat cleandatabasescat cleandatareads
这个过程涉及一系列的命令,用于处理和统计经过质量控制后的FASTQ文件中的数据。下面是每一步的解释:
准备工作和文件组织
创建目录 :
mkdir cleandataBase 创建一个名为cleandataBase的目录,用于存储最终的处理结果。
生成FASTQ文件列表 :
使用ls命令列出当前工作目录下所有经过质量控制处理的R1和R2文件,并分别将这些文件的路径保存到cleandata_fastq_1和cleandata_fastq_2文件中。
生成样本名称列表 :
find *_1.fq.gz >sample 查找所有以_1.fq.gz结尾的文件(通常代表R1的质控后文件),并将这些文件名输出到sample文件中。使用sed命令修改sample文件中的内容,去掉文件名中的.R1_val_1.fq.gz部分,仅留下样本名称。
合并文件列表和样本名称 :
paste cleandata_fastq_1 cleandata_fastq_2 sample > cleandata_fastq 将R1和R2的文件路径列表以及对应的样本名称合并到一个文件中,每行包含一对质控后的FASTQ文件和对应的样本名称。
执行统计分析
编写并执行统计脚本 (fc.sh):
脚本读取cleandata_fastq文件中的每一行,这一行包含了一对配对的FASTQ文件路径和样本名称。
对于文件中的每一对FASTQ文件,脚本使用fastq-count(一个假设的命令,用于示例说明)来统计其中的序列数量,并将统计结果输出到cleandataBase目录下,文件名为样本名称。
脚本以后台方式执行,允许同时处理多个文件。
给脚本执行权限并运行 :
chmod a+x fc.sh 使脚本可执行。nohup ./fc.sh & 在后台运行脚本,nohup确保即使终端关闭,脚本也会继续运行。
数据汇总和格式化
汇总统计结果 :
cd cleandataBase 切换到存储结果的目录。cat * > cleandataBase 将目录中所有文件的内容合并到一个名为cleandataBase的文件中。
格式化统计结果 :
使用sed命令调整cleandataBase文件的格式,例如去除不需要的字符,将特定符号替换为制表符,以便更容易地读取和分析数据。
使用awk命令从格式化后的文件中提取特定的列(如序列基数和读数),并将这些数据保存到cleandatabases和cleandatareads文件中,分别用于进一步分析。
这个过程从组织经过质量控制的FASTQ文件开始,通过统计分析,最终生成易于阅读和分析的数据汇总。这对于理解每个样本的数据质量和量化信息非常有用。
最终得到的数据
cleandatabases:包含每个样本的序列基数(即序列的唯一标识符数目),这有助于了解样本的多样性和复杂性。
9,建立索引,用上面的就行,下载fa和gtf文件 1 2 3 4 5 6 7 8 9 cd /public/jychu/reference/index/hisat/sheepwget http://ftp.ensembl.org/pub/release-104/fasta/anser_brachyrhynchus/dna/Anser_brachyrhynchus.ASM259213v1.dna.toplevel.fa.gz gzip -d Anser_brachyrhynchus.ASM259213v1.dna.toplevel.fa.gz mkdir index hisat2-build Anser_brachyrhynchus.ASM259213v1.dna.toplevel.fa index/index cd /public/jychu/reference/annotate/chickenwget http://ftp.ensembl.org/pub/release-104/gtf/anser_brachyrhynchus/Anser_brachyrhynchus.ASM259213v1.104.gtf.gz gzip -d Anser_brachyrhynchus.ASM259213v1.104.gtf.gz
10,hisat2比对,批量比对,上面也有 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 cd /public/jychu/lncRNA-chicken/cleandatamkdir bamls `pwd `/*_1.fq.gz > cleandata_fastq_1ls `pwd `/*_2.fq.gz > cleandata_fastq_2find *_1.fq.gz >sample sed -i "s/.R1_val_1.fq.gz/ /g" sample paste cleandata_fastq_1 cleandata_fastq_2 sample> cleandata_fastqvim compare.sh INDEX='/public/jychu/reference/index/hisat/chicken/index/index' cat cleandata_fastq|while read id do arr=(${id} ) fq1=${arr[0]} fq2=${arr[1]} sample=${arr[2]} hisat2 -p 8 -x $INDEX -1 $fq1 -2 $fq2 | samtools view -S -b - > bam/$sample .bam & done chmod a+x compare.shnohup ./compare.sh
11,排序,批量排序,上面有 1 2 3 4 5 6 7 8 cd bamls *.bam|while read id do samtools sort -@ 4 $id -o ${id%.*} .sorted rm -f ${id%.*} .bamdone
12,统计比对率 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 mkdir flagstatls *.bam > flagstat_bamfind *.bam>bam_sample sed -i "s/.sorted.bam/ /g" bam_sample paste flagstat_bam bam_sample >flagstatbam_listvim flagstatbam.sh cat flagstatbam_list |while read id do arr=(${id} ) bam=${arr[0]} sample=${arr[1]} samtools flagstat $bam > flagstat/$sample .flagstat.txt & done chmod a+x flagstatbam.shnohup ./flagstatbam.shcd flagstatcat * > flagstatgrep "0 mapped" flagstat > flagstats cat flagstats | awk '{print $5}' > flagsed -i "s/(/ /g" flag sed -i "s/%/ /g" flag cat flag
13,stringTie组装,批量组装 1 2 3 4 5 6 7 8 9 10 11 12 mkdir gtfsfor i in `ls *.sorted`;do new=${i%.*} .sorted.bammv "$i " "$new " done ls *.sorted.bam|while read id do nohup stringtie $id -p 4 -G /public/jychu/reference/annotate/chicken/Anser_brachyrhynchus.ASM259213v1.104.gtf -o gtfs/${id} .gtf -l ${id%.*} &done
14,合并转录本 1 2 3 4 5 cd gtfs mkdir mergedls *.gtf > merge.liststringtie --merge -G /public/jychu/reference/annotate/chicken/Anser_brachyrhynchus.ASM259213v1.104.gtf merge.list -o merged/merged.gtf
15,定量,获得转录本表达矩阵 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 cd /public/jychu/lncRNA-chicken/cleandata/bammkdir ballgownls *sorted.bam|while read id do stringtie -e $id -p 4 -G gtfs/merged/merged.gtf -o ballgown/${id%%.*} .bg.gtf -b ballgown/${id} _out_dirdone cd ballgownmkdir qualityls *.bg.gtf|while read id ; do cat $id | awk '$3=="transcript"' |grep -Eo 'transcript_id \"\w+|transcript_id \"\w+\.\w+\.\w+' |cut -d\" -f 2 > quality/${id%%.*} .t; done ls *.bg.gtf|while read id do less $id |awk '$3=="transcript"' |grep -Eo 'FPKM \"\w+\.\w+' |awk -F\" '{print$2}' > quality/${id%%.*} .value done cd qualityls *.t|while read id ; do paste ${id%%.*} .t ${id%%.*} .value > ${id%%.*} .FPKM ; done ls *.FPKM|while read id ;do less $id |sort -k 1 > ${id%.*} .sorted.FPKM;done echo "transcript" > FPKMless `ls *.sorted.FPKM|shuf -n1`|cut -f 1 >> FPKM ls *.sorted.FPKM|while read id ;do echo ${id%%.*} > ${id%%.*} .tmp;less $id |cut -f 2 >> ${id%%.*} .tmp;done paste FPKM *.tmp > all.FPKMls *.value *.t *.FPKM *.tmp FPKM|grep -v all.FPKM |xargs rm
16,ballgrown差异分析 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 cd /public/jychu/lncRNA-chicken/cleandata/bam/ballgownconda activate dna R if (!requireNamespace("BiocManager" , quietly = TRUE)) install.packages("BiocManager" ) BiocManager::install("ballgown" ) library(ballgown) bg = ballgown(samples = c("V0-1.sorted.bam_out_dir" , "V0-3.sorted.bam_out_dir" ,"V0-4.sorted.bam_out_dir" ,"V1-2.sorted.bam_out_dir" ,"V1-3.sorted.bam_out_dir" ,"V1-4.sorted.bam_out_dir" ),meas='all' )transcript_fpkm = texpr(bg , 'FPKM' ) head (transcript_fpkm)pData(bg ) <- data.frame(id =sampleNames(bg ),group=rep(c(1,0), each=3)) stat_results = stattest(bg ,feature='transcript' ,meas='FPKM' ,covariate='group' ) write.csv(stat_results, "ballgrown_transcript_results.csv" ,row.names=FALSE) ------------------------------------------------------------------ cd /public/jychu/lncRNA-chicken/cleandata/bam/ballgown/V0-1.sorted.bam_out_dircat t_data.ctab|cut -f2-8>test ------------------------------------------------------------------ 在你提供的命令和上下文中,`V0-1.sorted.bam_out_dir` 是一个目录,用于存储特定样本的`ballgown`分析数据。具体来说,这个目录通常包含`ballgown`需要读取的文件,用于进行转录组数据的表达量分析和差异表达分析。而这些文件通常是由`StringTie`生成的,用于描述样本中转录本的表达量信息。 在`V0-1.sorted.bam_out_dir`这样的目录中,你可能会找到以下类型的文件: 1. **e_data.ctab**:包含每个外显子的表达量信息,如外显子ID、长度、覆盖度等。 2. **i_data.ctab**:包含每个内含子的信息。 3. **t_data.ctab**:包含每个转录本的表达量信息,如转录本ID、长度、FPKM(Fragment Per Kilobase Million)值等。 4. **e2t.ctab** 和 **i2t.ctab**:分别是外显子到转录本和内含子到转录本的映射文件。 以`t_data.ctab`为例,它的内容可能如下:
t_id gene_id length eff_length est_counts abundance
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 - `t_id`:转录本ID - `gene_id`:基因ID - `length`:转录本长度 - `eff_length`:考虑到测序读长后的有效长度 - `est_counts`:估计的计数,基于对该转录本的覆盖度 - `abundance`:表达量估计,比如FPKM值 你最后的命令`cat t_data.ctab | cut -f2-8 > test` 是将`t_data.ctab`文件中的第2到第8列的内容提取出来,并将这部分内容重定向到一个名为`test`的新文件中。这意味着你会得到一个包含转录本ID、基因ID、长度、有效长度、估计计数和丰度等信息的文件,但是以一种更简化的格式。 这些步骤和文件是转录组分析流程中的一部分,用于评估和比较不同样本中的基因和转录本的表达水平。 这段代码主要用于进行转录组数据的差异表达分析,使用的是`ballgown`包在R语言环境下。它从一系列的`.bam`文件中读取数据,然后进行统计分析,最终生成差异表达的结果。这里简单解释一下输入和输出文件的内容: ### 输入文件: 1. **.bam文件**:这些是经过排序的二进制版本的序列对齐/比对文件(例如,`V0-1.sorted.bam`),通常由比对工具(如`TopHat`、`HISAT2`等)生成。`.bam`文件包含了序列数据(如RNA序列)与参考基因组的对齐信息。 2. **t_data.ctab**:这是`ballgown`需要的文件之一,包含了转录本的表达量和其他统计数据。它位于每个样本的`ballgown`输出目录中(如`V0-1.sorted.bam_out_dir`)。`ctab`文件包括转录本ID、基因ID、长度、覆盖度、FPKM等信息。 ### 输出文件: 1. **ballgrown_transcript_results.csv**:这个文件包含了转录本水平的差异表达分析结果。对于每个转录本,它可能包含列如转录本ID、基因名、统计值(如p值)、FPKM值等,用于指示在不同条件或组之间表达量的显著差异。 2. **test**(通过`cut`命令从`t_data.ctab`生成):这个文件是`t_data.ctab`的简化版本,只保留了部分列(第2到第8列),通常包括转录本ID、位置信息、编码区长度、外显子数量等。这个文件更便于用户进行快速查看或特定分析。 ### 例子: 假设一个简化的`t_data.ctab`文件如下:
t_id gene_id length exon_count FPKM
1 2 3 经过上述代码处理后,得到的`test`文件将会是:
gene_id length exon_count FPKM
1 2 3 而`ballgrown_transcript_results.csv`将包含差异表达分析的结果,比如:
transcript_id gene_name p_value q_value FPKM_group1 FPKM_group2
1 2 3 4 5 6 这个文件显示了每个转录本在不同组之间的表达量差异,包括统计显著性和表达量变化。
17,初步筛选lncRNA#,上面也有 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 cd /public/jychu/lncRNA-chicken/cleandata/bam/gtfs/mergedmkdir Identificationcd Identificationgffcompare -R -M -r /public/jychu/reference/annotate/chicken/Anser_brachyrhynchus.ASM259213v1.104.gtf -s /public/jychu/reference/index/hisat/chicken/Anser_brachyrhynchus.ASM259213v1.dna.toplevel.fa ../merged.gtf cd ~/soft/wget https://github.com/Dukunbioinfo/in-house-gtftools/archive/master.zip unzip master.zip cd in-house-gtftools-master/g++ *.cpp -o inhouseGTFtools ~/soft/in-house-gtftools-master/inhouseGTFtools STscreen -classCode u -i gffcmp.annotated.gtf > u.gtf ~/soft/in-house-gtftools-master/inhouseGTFtools STscreen -classCode i -i gffcmp.annotated.gtf > i.gtf ~/soft/in-house-gtftools-master/inhouseGTFtools STscreen -classCode x -i gffcmp.annotated.gtf > x.gtf cat i.gtf u.gtf x.gtf > iux.gtfconda install gffread -y gffread -w iux.fa -g /public/jychu/reference/index/hisat/chicken/Anser_brachyrhynchus.ASM259213v1.dna.toplevel.fa iux.gtf samtools faidx iux.fa less iux.fa.fai |awk '$2>200{print$1}' > iuxL200.MSTRG cat iuxL200.MSTRG | tr "." "\t" > iuxL200.tmpcat iuxL200.tmp | while read id ;do arr=(${id} ) MSTRG=${arr[0]} N1=${arr[1]} N2=${arr[2]} grep -w "$MSTRG \.$N1 \.$N2 " ../../../ballgown/quality/all.FPKM >> iux200FPKM.tmp done cat iux200FPKM.tmp | awk '$2>0.1 || $3>0.1 || $4>0.1 || $5>0.1 || $6>0.1 || $7>0.1 {print$1}' > iux200LFPKM0.1.name cat iux200LFPKM0.1.name | tr "." "\t" > iux200LFPKM0.1.tmpcat iux200LFPKM0.1.tmp | while read id ;do arr=(${id} ) MSTRG=${arr[0]} N1=${arr[1]} N2=${arr[2]} grep -w "$MSTRG \.$N1 \.$N2 " gffcmp.annotated.gtf >> iux200LFPKM0.1.gtf done gffread -w iux200LFPKM0.1.fa -g /public/jychu/reference/index/hisat/chicken/Anser_brachyrhynchus.ASM259213v1.dna.toplevel.fa iux200LFPKM0.1.gtf rm -f *.tmp执行这一系列的命令后,你将完成一整套基于转录组数据的过滤和分析流程,特别是针对长非编码RNA(lncRNA)的鉴定和分析。这个过程包括几个关键步骤:比对、分类、过滤和序列提取。具体来说,你会得到: 1. **比对和分类(gffcompare)**:使用`gffcompare`比对你的转录本到参考基因组,考虑了移除单外显子转录本的参数(`-M`),并且只关注与参考基因组重叠的转录本(`-R`)。`gffcompare`会生成几个文件,包括`gffcmp.annotated.gtf`,其中包含了转录本的分类代码(class codes),这些代码描述了转录本相对于参考基因组的位置关系。 2. **过滤(class code filtration)**:通过自定义工具`inhouseGTFtools`对`gffcmp.annotated.gtf`进行过滤,提取特定分类代码的转录本,如基因间(`u`)、内含子(`i`)和反义(`x`)转录本,然后将这些分类的转录本合并成一个新的GTF文件`iux.gtf`。 3. **序列提取和长度过滤**:使用`gffread`根据`iux.gtf`从参考基因组提取相应的转录本序列到`iux.fa`,并利用`samtools faidx`和`awk`命令筛选出长度大于200bp的转录本,结果保存在`iuxL200.MSTRG`文件中。 4. **基于FPKM值的过滤**:通过脚本过滤出至少在一个样本中FPKM值大于0.1的转录本,并将这些转录本的ID保存在`iux200LFPKM0.1.name`文件中。 5. **最终提取和清理**:使用这个过滤后的转录本ID列表从`gffcmp.annotated.gtf`中提取相应的转录本信息,保存在`iux200LFPKM0.1.gtf`。然后,利用`gffread`再次从参考基因组中提取这些过滤后的转录本的序列,保存在`iux200LFPKM0.1.fa`。 6. **清理临时文件**:最后,删除所有中间生成的临时文件。 执行这些命令后,你将获得一组经过长度和表达量过滤后,可能包含潜在长非编码RNA的转录本序列文件(`iux200LFPKM0.1.fa`),以及相应的GTF文件(`iux200LFPKM0.1.gtf`),这为进一步的功能分析和实验验证提供了基础。
18,鉴定lncRNA#,CPC2,CPAT(详细) 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 131 132 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148 149 150 151 152 153 154 155 156 157 158 159 160 161 162 163 164 165 166 167 168 169 170 171 172 173 174 175 176 177 178 179 180 181 182 183 184 185 186 187 188 189 190 191 192 193 194 195 196 1.CPC2 先下载软件压缩包到桌面,地址(http://cpc2.gao-lab.org/data/CPC2-beta.tar.gz) tar xzvf CPC2-beta.tar.gz cd CPC2-beta/ ls pwd export CPC_HOME="/public/jychu/soft/CPC2-beta" cd libs/libsvm tar xzvf libsvm-3.18.tar.gz cd libsvm-3.18 ls make clean && make 1.1 使用 conda activate python2 cd /public/jychu/lncRNA-chicken/cleandata/bam/gtfs/merged/Identificationmkdir lncRNApython /public/jychu/soft/CPC2-beta/bin/CPC2.py -i iux200LFPKM0.1.fa -o lncRNA/chicken. cpc2 chicken.cpc2.txt grep "noncoding" chicken.cpc2.txt |cut -f1 >chicken.cpc2.lnc 2.CPAT 使用方法http://rna-cpat.sourceforge.net/ pip install CPAT 1.CAPT预测 mkdir -p /public/jychu/reference/index/hisat/chickenwget http://ftp.ensembl.org/pub/release-103/fasta/anser_brachyrhynchus/cdna/Anser_brachyrhynchus.ASM259213v1.cdna.all.fa.gz wget http://ftp.ensembl.org/pub/release-103/fasta/anser_brachyrhynchus/ncrna/Anser_brachyrhynchus.ASM259213v1.ncrna.fa.gz conda install seqkit grep "protein" Anser_brachyrhynchus.ASM259213v1.cdna.all.fa | sed "s/>//g" | tr " " "\t" |cut -f1 > cdna.name cat Anser_brachyrhynchus.ASM259213v1.cdna.all.fa | seqkit grep -f cdna.name > cdna.name.fa grep "lncRNA" Anser_brachyrhynchus.ASM259213v1.ncrna.fa| sed "s/>//g" | tr " " "\t" |cut -f1 > lncRNA.name cat Anser_brachyrhynchus.ASM259213v1.ncrna.fa | seqkit grep -f lncRNA.name > lncRNA.name.fa mkdir -p /public/jychu/soft/CPAT/chickenmake_hexamer_tab.py -c cdna.name.fa -n lncRNA.name.fa >/public/jychu/soft/CPAT/chicken/chicken_Hexamer.tsv make_logitModel.py -x ~/soft/CPAT/chicken/chicken_Hexamer.tsv -c cdna.name.fa -n lncRNA.name.fa -o /public/jychu/soft/CPAT/chicken/chicken cpat.py -x /public/jychu/soft/CPAT/chicken/chicken_Hexamer.tsv -d /public/jychu/soft/CPAT/chicken/chicken.logit.RData -g iux200LFPKM0.1.fa -o lncRNA/chicken.CAPT awk '$2>=200' chicken.CAPT.ORF_prob.tsv |cut -f1|tr "_" "\t" |cut -f1|sort |uniq > capt.alltrans awk '$10>=0.3 && $2>=200' chicken.CAPT.ORF_prob.tsv|cut -f1|tr "_" "\t" |cut -f1|sort |uniq > chicken.CAPT.unlncRNA cat chicken.unlncRNA capt.alltrans|sort |uniq -u > chicken.capt.lnc 这些步骤涉及到使用CPAT (Coding Potential Assessment Tool) 来评估转录本的编码潜力,特别是为了区分编码蛋白的mRNA和非编码的长链非编码RNA(lncRNA)。这是一个重要的过程,因为lncRNAs在基因调控、疾病发展等方面扮演着关键角色,而它们不编码蛋白质。 1. **安装seqkit**: 首先,安装`seqkit`,这是一个用于处理FASTA/FASTQ文件的高效工具。 2. **提取cdna序列名称**: 使用`grep`、`sed`、`tr `和`cut `命令从`.cdna.all.fa`文件中提取包含"protein" 的序列名称,这些是编码蛋白质的mRNA序列。 3. **提取cdna序列**: 通过`seqkit grep`命令,使用上一步提取的名称从相同的FASTA文件中提取对应的序列。 4. **提取lncRNA序列名称**: 类似地,从`.ncrna.fa`文件中提取包含"lncRNA" 的序列名称。 5. **提取lncRNA序列**: 使用`seqkit grep`命令,根据提取的lncRNA名称从FASTA文件中提取对应的序列。 6. **生成CPAT所需的hexamer表和逻辑回归模型**: `make_hexamer_tab.py`和`make_logitModel.py`脚本分别用于生成hexamer频率表和逻辑回归模型,这两者都是CPAT评估转录本编码潜力所需的。输入文件包括编码蛋白的mRNA序列(cdna.name.fa)和非编码的lncRNA序列(lncRNA.name.fa)。 7. **使用CPAT评估转录本的编码潜力**: `cpat.py`脚本根据之前生成的模型和hexamer表来评估`iux200LFPKM0.1.fa`文件中转录本的编码潜力,并输出结果。 8. **筛选转录本**: 最后,使用`awk`和`cut `命令根据ORF长度和编码潜力的阈值筛选转录本。这包括提取ORF长度大于200的转录本,以及编码潜力大于0.3的转录本(可能被认为是mRNA),然后识别出那些编码潜力小于0.3且ORF长度大于200的转录本(被认为是潜在的lncRNA)。 通过这些步骤,研究人员可以有效地从大量转录本数据中识别出可能具有重要生物学功能的lncRNAs,这对于理解复杂的基因表达调控网络至关重要。 3.EMBOSS transeq 把核酸序列转换成氨基酸序列 网址:https://www.ebi.ac.uk/Tools/st/emboss_transeq/ 在线版本,缺点是序列文件最大为1M,最多序列数为500 把iux200LFPKM0.1.fa 下载到桌面 cd /public/jychu/lncRNA-chicken/cleandata/bam/gtfs/merged/Identificationseqkit seq iux200LFPKM0.1.fa -w 0 > chicken.iux200LFPKM0.1.fa wc -l chicken.iux200LFPKM0.1.fa8034 chicken.iux200LFPKM0.1.fa cd /mnt/c/Users/TE/Desktop/lncRNA/绍梅师姐数据sed -n '1,250p' chicken.iux200LFPKM0.1.fa >chicken-1.iux200LFPKM0.1.fa sed -n '251,450p' chicken.iux200LFPKM0.1.fa >chicken-2.iux200LFPKM0.1.fa sed -n '451,650p' chicken.iux200LFPKM0.1.fa >chicken-3.iux200LFPKM0.1.fa sed -n '651,858p' chicken.iux200LFPKM0.1.fa >chicken-4.iux200LFPKM0.1.fa sed -n '859,890p' chicken.iux200LFPKM0.1.fa >chicken-5.iux200LFPKM0.1.fa sed -n '891,1160p' chicken.iux200LFPKM0.1.fa >chicken-6.iux200LFPKM0.1.fa sed -n '1161,1420p' chicken.iux200LFPKM0.1.fa >chicken-7.iux200LFPKM0.1.fa sed -n '1421,1700p' chicken.iux200LFPKM0.1.fa >chicken-8.iux200LFPKM0.1.fa sed -n '1701,1810p' chicken.iux200LFPKM0.1.fa >chicken-9.iux200LFPKM0.1.fa sed -n '1811,2070p' chicken.iux200LFPKM0.1.fa >chicken-10.iux200LFPKM0.1.fa sed -n '2071,2290p' chicken.iux200LFPKM0.1.fa >chicken-11.iux200LFPKM0.1.fa sed -n '2291,2476p' chicken.iux200LFPKM0.1.fa >chicken-12.iux200LFPKM0.1.fa sed -n '2477,2680p' chicken.iux200LFPKM0.1.fa >chicken-13.iux200LFPKM0.1.fa sed -n '2681,3000p' chicken.iux200LFPKM0.1.fa >chicken-14.iux200LFPKM0.1.fa sed -n '3001,3300p' chicken.iux200LFPKM0.1.fa >chicken-15.iux200LFPKM0.1.fa sed -n '3301,3562p' chicken.iux200LFPKM0.1.fa >chicken-16.iux200LFPKM0.1.fa sed -n '3563,3800p' chicken.iux200LFPKM0.1.fa >chicken-17.iux200LFPKM0.1.fa sed -n '3801,4100p' chicken.iux200LFPKM0.1.fa >chicken-18.iux200LFPKM0.1.fa sed -n '4101,4400p' chicken.iux200LFPKM0.1.fa >chicken-19.iux200LFPKM0.1.fa sed -n '4401,4840p' chicken.iux200LFPKM0.1.fa >chicken-20.iux200LFPKM0.1.fa sed -n '4841,5200p' chicken.iux200LFPKM0.1.fa >chicken-21.iux200LFPKM0.1.fa sed -n '5201,5550p' chicken.iux200LFPKM0.1.fa >chicken-22.iux200LFPKM0.1.fa sed -n '5551,5776p' chicken.iux200LFPKM0.1.fa >chicken-23.iux200LFPKM0.1.fa sed -n '5777,6100p' chicken.iux200LFPKM0.1.fa >chicken-24.iux200LFPKM0.1.fa sed -n '6101,6500p' chicken.iux200LFPKM0.1.fa >chicken-25.iux200LFPKM0.1.fa sed -n '6501,6870p' chicken.iux200LFPKM0.1.fa >chicken-26.iux200LFPKM0.1.fa sed -n '6871,7500p' chicken.iux200LFPKM0.1.fa >chicken-27.iux200LFPKM0.1.fa sed -n '7501,8034p' chicken.iux200LFPKM0.1.fa >chicken-28.iux200LFPKM0.1.fa 每一个结果保存为txt格式 cat *.txt>chicken.iux.ped seqkit seq chicken.iux.ped -w 0 >chicken.iux.ped.fa grep -rn "MSTRG.13560.1" chicken.iux.ped.fa 42493:>MSTRG.13560.1_1 42495:>MSTRG.13560.1_2 42497:>MSTRG.13560.1_3 42499:>MSTRG.13560.1_4 42501:>MSTRG.13560.1_5 42503:>MSTRG.13560.1_6 sed -i "42493,42504d" chicken.iux.ped.fa step1 通过hmmmerspress 来把下载的pfam数据建库 使用conda安装Pfam_scan ,hmmpress包含在里面 conda install pfam_scan 下载pfam数据 cd databasemkdir pfamcd pfamwget ftp://ftp.ebi.ac.uk:21/pub/databases/Pfam/current_release/Pfam-A.hmm.gz wget ftp://ftp.ebi.ac.uk:21/pub/databases/Pfam/current_release/Pfam-A.hmm.dat.gz gunzip *.gz hmmpress Pfam-A.hmm step2 用HMMER和pfam数据库比对,过滤比对上的序列(Evalue<1e-5) cd /public/jychu/lncRNA-chicken/cleandata/bam/gtfs/merged/Identification/lncRNAnohup pfam_scan.pl -fasta chicken.iux.ped.fa -dir /public/jychu/database/pfam -out chicken.pfam_scan.out &grep -v "#" chicken.pfam_scan.out | grep -v "^\s*$" |awk '{if($13<1e-5){print$1}}' | awk -F "_" '{print$1}' |sort |uniq >pfam.coding.ID cat pfam.coding.ID ../iux200LFPKM0.1.name|sort |uniq -u |grep -v "MSTRG.13560.1" >chicken.pfam.lnc step3 利用diamond将蛋白序列和nr-animal,uniRef100数据库比对,过滤比对上的序列(1e-5) 1.和nr-animal比对 ~/soft/diamond/diamond blastp -d ~/database/nr/nr.animal -q chicken.iux.ped.fa --sensitive --evalue 1e-5 -o chicken.nr-animal_matches_fmt6.txt cat chicken.nr-animal_matches_fmt6.txt| awk '{if($3>80){print$1}}' |awk -F "_" '{print$1}' |sort |uniq >nr.coding.ID cat nr.coding.ID ../iux200LFPKM0.1.name|sort |uniq -u |grep -v "MSTRG.13560.1" >chicken.nr.lnc2.和uniRef100比对 cd ~/databasemkdir uniRefcd uniRefwget https://ftp.uniprot.org/pub/databases/uniprot/uniref/uniref100/uniref100.fasta.gz gunzip uniref100.fasta.gz ~/soft/diamond/diamond makedb --in uniref100.fasta --db uniref100 ~/soft/diamond/diamond blastp -d ~/database/uniRef/uniref100 -q chicken.iux.ped.fa --sensitive --evalue 1e-5 -o chicken.uniref100_matches_fmt6.txt cat chicken.uniref100_matches_fmt6.txt| awk '{if($3>80){print$1}}' |awk -F "_" '{print$1}' |sort |uniq >uniref100.coding.IDcat uniref100.coding.ID ../iux200LFPKM0.1.name|sort |uniq -u |grep -v "MSTRG.13560.1" >chicken.uniref100.lncmkdir lnccp *.lnc lnc/ cat *.lnc|sort |uniq -c|tr " " "\t" |cut -f7,8|awk '{if($1==5){print$2}}' >chicken.lncRNA.lnc 这个流程是用于鉴定和验证lncRNA(长非编码RNA)序列的一系列步骤,主要通过不同的生物信息学工具和数据库对给定的转录本序列进行编码潜力分析和同源性搜索。下面是这个流程的具体解释: - 首先,利用`seqkit`将`iux200LFPKM0.1.fa`文件中的序列格式化,每个序列一行,便于处理。 - 因为EMBOSS transeq的在线版本限制了文件大小和序列数目,所以需要将大文件分割成多个小文件,每个包含一定数量的序列。 - 这些小文件准备上传到EMBOSS transeq工具在线转换核酸序列到氨基酸序列,由于大小和数量限制,这是必须的步骤。 - 将所有分割文件的转换结果合并,准备进一步的分析。 - 使用`Pfam_scan.pl`脚本,结合Pfam数据库,对合并后的序列进行同源性搜索,筛选出可能具有编码潜力的序列(基于E-value的阈值)。 - 进一步使用DIAMOND工具对序列进行同源性搜索,与nr-animal和uniRef100数据库比对,筛选出具有高度同源性的序列,即可能具有编码潜力的序列。 - 通过上述步骤筛选出的编码潜力序列,与原始转录本列表对比,排除这些可能编码蛋白的转录本,剩下的被认为是lncRNA。 - 最终,通过各种方法的交叉验证,得到最终被认定为lncRNA的转录本列表。 这个过程涉及到多个生物信息学工具和数据库,每一步都是为了确保所鉴定的lncRNA序列具有较高的可信度,排除那些可能具有编码潜力或与已知蛋白质高度同源的序列。通过这种方法,可以有效地识别出具有研究和临床价值的lncRNA序列。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 setwd("C:/Users/TE/Desktop/lncRNA/绍梅师姐数据/LNC" ) V1 = read.table("chicken.capt.lnc" ,header=F, sep="\t" ) capt=V1$V1 V2 = read.table("chicken.cpc2.lnc" ,header=F, sep="\t" ) cpc2=V2$V1 V3 = read.table("chicken.nr.lnc" ,header=F, sep="\t" ) nr=V3$V1 V4 = read.table("chicken.pfam.lnc" ,header=F, sep="\t" ) pfam=V4$V1 V5 = read.table("chicken.uniref100.lnc" ,header=F, sep="\t" ) uniref=V5$V1 library(VennDiagram) venn.diagram(list("CAPT" = capt,"CPC2" =cpc2,"NR" = nr,"Pfam" =pfam,"UniRef100" = uniref),height=6000,width=6000,resolution=840, filename="VennPlot.tiff" ,col="white" ,fill = c("#7fffd4" , "#ff69b4" ,"#ffa07a" ,"#7cfc00" ,"#ff8c00" ), alpha = 0.55,fontfamily = "serif" ,fontface = "bold" , cat.col = "black" ,cat.cex=1.6,cat.dist = c(0.18,0.192,0.22,0.22,0.196), cat.pos = c(0,330,250,115,25),cat.fontfamily = "serif" ) 这个R脚本是用来生成一个Venn图,目的是为了可视化不同方法预测出的长非编码RNA(lncRNA)之间的重叠情况。Venn图是展示集合间交集与并集的一种图形化方法。在这个脚本中,使用了五个不同的数据集,每个数据集代表一种预测lncRNA的方法或来源: 1. **CAPT**:通过CPAT预测的lncRNA。 2. **CPC2**:通过CPC2(Coding Potential Calculator 2)预测的lncRNA。 3. **NR**:通过与NR(非冗余蛋白数据库)比对,预测的非编码RNA。 4. **Pfam**:通过与Pfam数据库比对,预测的非编码RNA。 5. **UniRef100**:通过与UniRef100数据库比对,预测的非编码RNA。 脚本步骤如下: - 首先,设置工作目录到存储预测结果文件的文件夹。 - 使用`read.table`函数读取每个预测结果文件,并将结果存储在变量中。 - 载入`VennDiagram`包,用于生成Venn图。 - 使用`venn.diagram`函数生成Venn图,输入是一个列表,列表中包含了上述五个变量。每个变量代表一组lncRNA的集合。 - 在`venn.diagram`函数中,指定了各种参数,如图形的尺寸、分辨率、文件名、颜色、透明度、字体等,以定制Venn图的外观。 - `filename="VennPlot.tiff" `参数指定输出文件的名称和格式。 - `fill`参数定义了五个集合的颜色。 - `alpha`参数设置颜色的透明度,使得重叠区域的颜色混合可以清晰可见。 - `cat.col`、`cat.cex`、`cat.dist`、`cat.pos`和`cat.fontfamily`等参数用于定制分类标签的颜色、大小、距离、位置和字体。 通过这个Venn图,研究人员可以直观地看到不同方法预测的lncRNA集合之间的相互关系,包括它们之间的独有和共有预测结果,这有助于评估预测方法的一致性和差异性,以及确定潜在的lncRNA候选。
mRNA鉴定的思路:完全匹配到粉脚鹅已有的参考基因组上mRNA的转录本定义为已知的mRNA
20,把所有转录本注释分类为mRNA\lncRNA、新的鉴定的lncRNA等 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 已知的 cd /public/jychu/reference/annotate/chickencat Anser_brachyrhynchus.ASM259213v1.104.gtf |awk '{if($3=="transcript"){print$0}}' |tr ";" "\t" |sort -k11|cut -f1,4,5,7,9,11,13-15|sed "s/gene_id //g" |sed "s/transcript_id //g" |sed "s/\"//g" |tr " " ":" |sed "s/:ENSABRT/ENSABRT/g" |sed "s/:gene/gene/g" |awk -F "\t" '{if($7=="gene_source:ensembl"){$7=$6}}{print$0}' |tr " " "\t" |awk -F "\t" '{if($8=="gene_source:ensembl"){$8=$9}}{print$0}' |tr " " "\t" |cut -f1-8|sed -e "s/gene_name://g;s/gene_biotype://g" >chicken.transcript.type下载到桌面,用excel打开 新的lncRNA cd /mnt/c/Users/TE/Desktop/lncRNA/绍梅师姐数据/LNCcat *.lnc |sort |uniq -c|tr " " "\t" |cut -f7-8 |awk '{if($1==5){print$0}}' |cut -f2>novel.lnccat novel.lnc | while read id ;do arr=(${id} ) MSTRG=${arr[0]} N1=${arr[1]} N2=${arr[2]} grep -w "$MSTRG \.$N1 \.$N2 " ../ballgown/alltranscript.txt >> novel.lnc.expression done 这段脚本是用于从之前通过多种方法预测得到的长非编码RNA(lncRNA)候选集中,进一步筛选出同时被所有方法预测为lncRNA的转录本,并提取这些转录本的表达数据。 1. **合并并筛选lncRNA**: 首先,使用`cat *.lnc | sort | uniq -c`命令合并所有的`.lnc`文件(这些文件包含了不同预测方法识别的lncRNA候选),然后通过管道(`|`)传递给`tr " " "\t" `将空格转换为制表符,再通过`cut `和`awk`命令来筛选出在所有五个预测方法(CAPT、CPC2、NR、Pfam、UniRef100)中都被识别为lncRNA的转录本。这些转录本的ID被保存在`novel.lnc`文件中。 2. **提取表达数据**: 接下来,脚本通过读取`novel.lnc`文件中的每个转录本ID,使用`grep`命令在一个包含所有转录本表达信息的文件(`../ballgown/alltranscript.txt`)中搜索这些ID对应的表达数据。找到的表达数据被追加到`novel.lnc.expression`文件中。(筛选找到的lncRNA又没有已知的) 这个过程允许研究人员聚焦于那些高度可能是真正的lncRNA的转录本,并进一步分析这些转录本的表达模式,这对于理解它们在生物过程中的作用是非常有价值的。 当然可以。让我们通过一个简化的例子来说明这个过程。 假设你有多个`.lnc`文件,每个文件都包含一系列转录本ID,这些ID由不同的方法预测为lncRNA。文件内容可能如下: **capt.lnc**
MSTRG.1.1
MSTRG.1.1
MSTRG.1.1
MSTRG.1.1
MSTRG.1.1
1 2 3 4 5 6 7 8 ### 中间过程 当执行`cat *.lnc | sort | uniq -c | ...`这一系列命令时,目的是找出在所有文件中都出现的转录本ID。在这个例子中,`MSTRG.1.1`是唯一出现在所有五个文件中的ID。 因此,`novel.lnc`文件将包含: **novel.lnc**
MSTRG.1.1
1 2 3 4 5 6 ### 输出文件示例 假设`../ballgown/alltranscript.txt`文件包含了转录本及其表达数据,如下所示: **alltranscript.txt**
MSTRG.1.1 100 200
1 2 3 4 执行第二部分的脚本后,`novel.lnc.expression`文件将包含`MSTRG.1.1`的表达数据,因为它是被所有方法共同预测的lncRNA。 **novel.lnc.expression**
MSTRG.1.1 100 200
1 2 3 4 5 6 7 8 9 在这个简化的例子中,我们看到如何从多个预测方法中筛选出共同的lncRNA转录本,并从一个包含表达数据的文件中提取这些转录本的信息。
21,取新的转录本对应的xloc编号,重要,我也能做到,不过利用自己的那个插件 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 cd /public/jychu/lncRNA-chicken/cleandata/bam/gtfs/merged/Identificationcat iux.gtf|awk '{if($3=="transcript"){print$0}}' >test.tmpvi novel.lnc Vim 的命令模式中输入 :%s/^M$//g 消除EXCEL模式 cat novel.lnc|tr "." "\t" >novel.lnc.tmpvi test.sh cat novel.lnc.tmp | while read id ;do arr=(${id} ) MSTRG=${arr[0]} N1=${arr[1]} N2=${arr[2]} grep -w "$MSTRG \.$N1 \.$N2 " test.tmp >> novel.lnc.tmp.tmp done chmod a+x test.sh./test.sh cat novel.lnc.tmp.tmp|cut -f9|tr ";" "\t" |cut -f1,3- |sed "s/\"//g" |sed -e "s/transcript_id //g;s/xloc //g" |awk '{$NF=NULL;print}' |awk '{$NF=NULL;print}' |sed "s/class_code//g" |awk '{if($2=="gene_name"){print$1"\t"$4"\t"$7"\t"$2":"$3";"$5":"$6}else{print$1"\t"$2"\t"$3}}' >novel.lnc.test下载到桌面上 rm -f *.tmp这个脚本的目的是处理特定的转录本数据,提取特定转录本的相关信息,并生成一个格式化的输出文件。下面是各个步骤的解释: 1. **提取转录本信息**: - `cat iux.gtf | awk '{if($3=="transcript"){print$0}}' > test.tmp`:从`iux.gtf`文件中提取所有表示转录本的行(第三个字段为`transcript`),并将这些行保存到`test.tmp`。 2. **处理`novel.lnc`文件**: - 使用`vi novel.lnc`打开`novel.lnc`文件,并在Vim的命令模式中输入`:%s/^M$//g`来删除由Excel导入时可能引入的特殊字符(`^M`)。 - `cat novel.lnc | tr "." "\t" > novel.lnc.tmp`:将`novel.lnc`文件中的点(`.`)转换为制表符(`\t`),并保存到临时文件`novel.lnc.tmp`中。 3. **编写和执行`test.sh`脚本**: - 在`test.sh`脚本中,通过`cat novel.lnc.tmp | while read id ; do ... done `循环读取`novel.lnc.tmp`中的每行,这些行包含了需要进一步搜索的转录本ID。 - 对于每个ID,使用`grep -w`从`test.tmp`中查找匹配的行,并将结果追加到`novel.lnc.tmp.tmp`。 - `chmod a+x test.sh`:给`test.sh`脚本文件添加执行权限。 - `./test.sh`:执行`test.sh`脚本。 4. **格式化输出文件**: - `cat novel.lnc.tmp.tmp | cut -f9 | tr ";" "\t" | cut -f1,3- | sed "s/\"//g" | sed -e "s/transcript_id //g;s/xloc //g" | awk '{$NF=NULL;print}' | awk '{$NF=NULL;print}' | sed "s/class_code //g" | awk '{...}' > novel.lnc.test`:这一系列命令对匹配的转录本信息进行了多步处理,包括字段提取、替换、格式化等,最终生成的格式化输出保存在`novel.lnc.test`。 5. **清理临时文件**: - `rm -f *.tmp`:删除所有临时文件。 **输入输出文件例子**: - **输入** (`novel.lnc`原始格式):
MSTRG.1.11 - **中间文件** (`novel.lnc.tmp`格式化后):
1 - **输出** (`novel.lnc.test`):
1 2 3 4 5 6 7 8 9 这个流程从`iux.gtf`文件中筛选特定的转录本,处理`novel.lnc`中的转录本ID,执行文本处理和格式化,最终生成一个包含所需转录本信息的文件。
22,差异表达的lncRNA顺式靶基因预测 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 先把差异lncRNA筛选出来 在坐标前后加减100kb 然后把所有的mRNA整理出来 -wa -wb 输出overlap的区域所在-a和-b中的原内容: bedtools intersect -a lnc.bed.txt -b mRNA.bed.txt -wb |cut -f4,8|sort |uniq >cis.lnc 这条命令使用`bedtools intersect`来找出`lnc.bed.txt`(长非编码RNA, lncRNA)和`mRNA.bed.txt`(信使RNA, mRNA)文件中空间上相交(overlap)的区域。`-wb`选项意味着除了输出`-a`文件中的内容,还会输出与之相交的`-b`文件中的内容。接着,使用`cut `命令提取第4和第8列,这通常代表着特定的标识符或名称(具体取决于BED文件的格式)。然后,`sort `和`uniq `命令用来排序并去除重复的行,最后的结果被重定向到`cis.lnc`文件中。 - **lnc.bed.txt (输入文件a)**:
chr1 1000 2000 lncRNA11 2 3 这个文件列出了lncRNA的位置和名称,其中每行包含染色体名、起始位置、结束位置和lncRNA的名称。 - **mRNA.bed.txt (输入文件b)**:
1 2 3 4 5 类似地,这个文件列出了mRNA的位置和名称。 ### 输出文件内容示例 - **cis.lnc (输出文件)**:
1 2 3 4 5 6 这个文件包含了空间上与mRNA相交的lncRNA的名称。在这个例子中,`lncRNA1`和`mRNA1`在染色体1上的位置有重叠,`lncRNA2`和`mRNA2`也是如此,这意味着它们可能在功能上有联系,比如在同一个调控网络中。 这个过程对于研究lncRNA和mRNA之间的空间和功能关系非常重要,特别是在探索基因表达调控和基因间相互作用的研究中。通过识别空间上相交的lncRNA和mRNA,研究者可以进一步分析它们是否参与相同的生物过程或疾病。
23,差异表达的lncRNA的反式靶基因准备文件如下格式 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 R data<-read.table(file="lnc-mRNA.fpkm" ,row.names=1,header=F) corr<- round(cor(t(data)),3) head (corr[,1:16])id = row.names(corr)n = dim(corr)[1] re = data.frame(ID1= rep(id ,n),ID2=rep(id ,each = n),r = round(as.vector(corr),3)) nrow(re) res=subset(re,re$r >0.95 | re$r < -0.95) res1<-res[grep(pattern="lncRNA" ,res[,1]),] res2<-res1[grep(pattern="protein_coding" ,res1[,2]),] write.table(res2,file="trans.lnc" ,sep="\t" ) 这段代码主要用于处理基因表达数据,计算转录本间的相关性,并筛选出与长非编码RNA(lncRNA)表达强相关的蛋白编码基因(protein_coding)。让我们一步步解析代码: 1. **读取数据**: - `data <- read.table(file="lnc-mRNA.fpkm" , row.names=1, header=F)`:从文件`lnc-mRNA.fpkm`中读取数据,假设第一列为行名(转录本ID),没有列名(header=F)。 2. **计算相关性**: - `corr <- round(cor(t(data)), 3)`:计算转置后的数据矩阵(`t(data)`)的相关性矩阵,并将结果四舍五入到小数点后三位。转置是因为`cor()`函数默认计算列与列之间的相关性,而这里的数据每一行代表一个转录本的表达,每一列代表一个样本。 3. **查看前16列的相关性**: - `head (corr[, 1:16])`:查看相关性矩阵的前16列,这可以提供一些关于转录本间相关性的初步信息。 4. **创建一个包含所有可能的转录本对及其相关性的数据框**: - 使用`rep(id , n)`和`rep(id , each = n)`生成所有可能的转录本对组合。 - `r = round(as.vector(corr), 3)`:将相关性矩阵转换成一个向量,并四舍五入到小数点后三位。 - `re = data.frame(ID1= ..., ID2= ..., r = ...)`:创建一个新的数据框,包含每一对转录本的ID(ID1和ID2)及它们之间的相关性(r)。 5. **筛选高度相关的转录本对**: - `res = subset(re, re$r > 0.95 | re$r < -0.95)`:从创建的数据框中筛选出相关性大于0.95或小于-0.95的转录本对,即寻找非常强的正相关或负相关。 6. **进一步筛选涉及lncRNA和蛋白编码基因的对**: - `res1 <- res[grep(pattern="lncRNA" , res[, 1]),]`:在筛选出的高度相关转录本对中,进一步筛选出第一列(ID1)包含"lncRNA" 的行。 - `res2 <- res1[grep(pattern="protein_coding" , res1[, 2]),]`:在上一步的基础上,进一步筛选出第二列(ID2)包含"protein_coding" 的行,即保留那些lncRNA与蛋白编码基因之间高度相关的对。 7. **输出结果**: - `write.table(res2, file="trans.lnc" , sep="\t" )`:将最终筛选出的转录本对写入到文件`trans.lnc`中,使用制表符作为字段分隔符。 这个过程通过筛选相关性高的转录本对,特别是关注于lncRNA与蛋白编码基因之间的相关性,有助于识别可能在调控路径或生物过程中发挥作用的关键lncRNA。 这段代码旨在分析和筛选出具有高度相关性的长非编码RNA(lncRNA)和蛋白质编码基因(protein_coding)之间的表达量数据。下面是对这个流程的简要解释,以及输入和输出文件的示例。 1. **读取数据**:`data <- read.table(file="lnc-mRNA.fpkm" , row.names=1, header=F)` 从名为 "lnc-mRNA.fpkm" 的文件中读取表达量数据,其中行名为基因ID,没有列名。 2. **计算相关性矩阵**:`corr <- round(cor(t(data)), 3)` 计算转录本表达数据的相关性矩阵,并将结果四舍五入到小数点后三位。这里使用 `t(data)` 是因为 `cor()` 函数需要在列之间计算相关性,而输入数据中的每一列代表一个样本,每一行代表一个基因。 3. **格式化输出**:创建一个新的数据框 `re`,包含每对基因ID的相关性得分。 4. **筛选高度相关的转录本对**:从 `re` 中筛选出相关性大于0.95或小于-0.95的条目,这意味着只保留极度正相关或负相关的转录本对。 5. **进一步筛选**:首先筛选出第一列(ID1)包含 "lncRNA" 的行,然后在这个子集中进一步筛选出第二列(ID2)包含 "protein_coding" 的行,即寻找与蛋白质编码基因高度相关的lncRNA。 6. **保存结果**:将筛选后的结果保存到名为 "trans.lnc" 的文件中。
Sample1 Sample2 Sample3
lncRNA1 2.5 3.2 2.8
1 2 3 4 5 这个文件包含了每个转录本在不同样本中的表达量(FPKM值)。 ### 输出文件示例 (`trans.lnc`)
“ID1” “ID2” “r”
1 2 3 4 5 6 7 8 这个文件列出了那些与蛋白质编码基因高度相关(相关性大于0.95或小于-0.95)的lncRNA,以及它们之间的相关性系数。 请注意,实际的输入和输出文件内容会根据实际数据而有所不同,这里只提供了一个简化的示例。
24,找有表达量的lncRNA做顺式反式靶基因预测 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 cd /mnt/c/Users/TE/Desktop/lncRNA/绍梅师姐数据/ballgownbedtools intersect -a alllnc.bed -b mRNA.bed.txt -wa -wb |cut -f4,5,9,10>cis.alllnc cd /mnt/c/Users/TE/Desktop/lncRNA/绍梅师姐数据/ballgownawk '{print $1":"$2":"$3"\t"$4"\t"$5"\t"$6"\t"$7"\t"$8"\t"$9}' alllnc-mRNA.fpkm.txt >alllnc-mRNA.fpkm R data<-read.table(file="alllnc-mRNA.fpkm" ,row.names=1,header=F) corr<- round(cor(t(data)),3) head (corr[,1:16])id = row.names(corr)n = dim(corr)[1] re = data.frame(ID1= rep(id ,n),ID2=rep(id ,each = n),r = round(as.vector(corr),3)) nrow(re) res=subset(re,re$r >0.95 | re$r < -0.95) res1<-res[grep(pattern="lncRNA" ,res[,1]),] res2<-res1[grep(pattern="protein_coding" ,res1[,2]),] write.table(res2,file="trans.alllnc" ,sep="\t" ) 这个过程涉及几个步骤,旨在找出lncRNA和mRNA之间的相关性,并筛选出高度相关的对。让我们逐步解析: ```bash bedtools intersect -a alllnc.bed -b mRNA.bed.txt -wa -wb | cut -f4,5,9,10 > cis.alllnc
bedtools intersect用于找出alllnc.bed(lncRNA的位置信息)和mRNA.bed.txt(mRNA的位置信息)中空间上重叠的区域。-wa -wb选项表示同时输出-a和-b文件中重叠区域的详细信息。cut -f4,5,9,10提取重要的字段,假设是lncRNA和mRNA的标识符及其位置信息。输出保存到cis.alllnc文件中,这个文件包含空间上相互作用的lncRNA和mRNA对。
第二步:准备相关性分析的数据 1 awk '{print $1":"$2":"$3"\t"$4"\t"$5"\t"$6"\t"$7"\t"$8"\t"$9}' alllnc-mRNA.fpkm.txt > alllnc-mRNA.fpkm
这一步使用awk处理alllnc-mRNA.fpkm.txt文件(包含lncRNA和mRNA的表达量数据),格式化输出以便于R语言读取。
第三步:在R中计算相关性并筛选高度相关的lncRNA-mRNA对
读取数据 :data <- read.table(file="alllnc-mRNA.fpkm", row.names=1, header=F)读取处理过的表达量数据。计算相关性 :corr <- round(cor(t(data)), 3)计算转置后的数据矩阵的相关性,round用于保留三位小数。构建相关性数据框 :构建一个包含所有可能对的数据框re,其中包含两个ID和它们之间的相关系数。筛选高度相关的对 :res中筛选出相关系数大于0.95或小于-0.95的对,然后进一步筛选出第一列包含lncRNA和第二列包含protein_coding的行,这意味着找出了与mRNA高度相关的lncRNA。保存结果 :将这些高度相关的lncRNA-mRNA对保存到trans.alllnc文件中。
输入文件示例
alllnc.bed 和mRNA.bed.txt 文件包含BED格式的lncRNA和mRNA的位置信息。alllnc-mRNA.fpkm.txt 包含lncRNA和mRNA的表达量数据。
输出文件示例
cis.alllnc 文件列出了空间上相互作用的lncRNA和mRNA对的标识符及其位置信息。trans.alllnc 文件包含了基于表达量数据计算得出的高度相关的lncRNA-mRNA对。
这个分析流程能够帮助研究者识别可能在基因表达调控中扮演重要角色的lncRNA和mRNA对,为进一步的生物学验证提供依据。
让我给你举个例子来说明这些输入和输出文件可能的内容。
输入文件示例
alllnc.bed (lncRNA的位置信息,BED格式):
1 2 chr1 1000 2000 lncRNA1 chr1 5000 6000 lncRNA2
mRNA.bed.txt (mRNA的位置信息,BED格式):
1 2 chr1 1500 2500 mRNA1 chr1 5500 6500 mRNA2
alllnc-mRNA.fpkm.txt (lncRNA和mRNA的表达量数据,假设格式为ID和FPKM值):
1 2 3 4 lncRNA1 10 lncRNA2 20 mRNA1 30 mRNA2 40
中间处理文件示例
alllnc-mRNA.fpkm (处理后的表达量数据文件,假设格式转换为列格式): 1 2 3 4 5 ID FPKM lncRNA1 10 lncRNA2 20 mRNA1 30 mRNA2 40
输出文件示例
cis.alllnc (空间上相互作用的lncRNA和mRNA对及其位置信息):
1 2 lncRNA1 1000 mRNA1 1500 lncRNA2 5000 mRNA2 5500
这个文件说明lncRNA1与mRNA1、lncRNA2与mRNA2在基因组上有空间上的接近或重叠,可能暗示着它们之间的功能关联或调控作用。
trans.alllnc (基于表达量数据计算得出的高度相关的lncRNA-mRNA对):
1 2 lncRNA1 mRNA1 0.95 lncRNA2 mRNA2 -0.96
这个文件列出了基于表达量数据(如FPKM值)计算得出的相关性很高(正相关或负相关)的lncRNA和mRNA对。在这个示例中,lncRNA1与mRNA1的相关性为0.95(正相关),而lncRNA2与mRNA2的相关性为-0.96(负相关),说明这些lncRNA可能通过某种方式影响或与这些mRNA的表达调控相关。
这些示例简单展示了从位置信息和表达量数据出发,通过空间关系(cis作用)和表达量相关性(trans作用)分析,来探索lncRNA与mRNA之间的潜在功能联系的过程。
两种FPKM的区别 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 131 132 133 134 135 136 137 138 139 140 141 142 下面两个FPKM值的获得方法,有什么区别或相同吗? 第一种: cd /public/jychu/lncRNA-chicken/cleandata/bammkdir ballgownls *sorted.bam|while read id do stringtie -e $id -p 4 -G gtfs/merged/merged.gtf -o ballgown/${id%%.*} .bg.gtf -b ballgown/${id} _out_dirdone cd ballgownmkdir qualityls *.bg.gtf|while read id ; do cat $id | awk '$3=="transcript"' |grep -Eo 'transcript_id \"\w+|transcript_id \"\w+\.\w+\.\w+' |cut -d\" -f 2 > quality/${id%%.*} .t; done ls *.bg.gtf|while read id do less $id |awk '$3=="transcript"' |grep -Eo 'FPKM \"\w+\.\w+' |awk -F\" '{print$2}' > quality/${id%%.*} .value done cd qualityls *.t|while read id ; do paste ${id%%.*} .t ${id%%.*} .value > ${id%%.*} .FPKM ; done ls *.FPKM|while read id ;do less $id |sort -k 1 > ${id%.*} .sorted.FPKM;done echo "transcript" > FPKMless `ls *.sorted.FPKM|shuf -n1`|cut -f 1 >> FPKM ls *.sorted.FPKM|while read id ;do echo ${id%%.*} > ${id%%.*} .tmp;less $id |cut -f 2 >> ${id%%.*} .tmp;done paste FPKM *.tmp > all.FPKMls *.value *.t *.FPKM *.tmp FPKM|grep -v all.FPKM |xargs rm 第二种 featureCounts -T 8 -a /home/data1/Ghl/bam/lncRNA.gtf -o lncRNA_raw_count.txt -p -B -C -f -t transcript -g transcript_id /home/data1/Ghl/bam/*.short.bam 生成了lncRNA_raw_count.txt 2,R代码 rm (list=ls ())raw_df <- read.table(file = "lncRNA_raw_count.txt" ,header = T,skip = 1,sep = "\t" ) count_df <- raw_df[ ,c(7:ncol(raw_df))] metadata <- raw_df[ ,1:6] rownames(count_df) <- as.character(raw_df[,1]) colnames(count_df) <- paste0("SRR10744" ,1:18) countToFpkm <- function (counts, effLen) { N <- colSums(counts) exp( log (counts) + log (1e9) - log (effLen) - log (N) ) } options(scipen = 200) fpkm = countToFpkm(count_df, metadata$Length ) write.table(fpkm, file = "fpkm_data.txt" , sep = "\t" , quote = FALSE) count_df.filter <- count_df[rowSums(fpkm)>0,] write.table(rownames(count_df.filter), file = "filter6_by_fpkm_id" , sep = "\t" , quote = FALSE) 这两种方法用于获得FPKM(Fragment Per Kilobase of transcript per Million mapped reads)值,这是一种用来量化基因表达的方法。尽管两者的最终目的相同,即获得转录本的FPKM值,但它们在数据处理流程、工具使用和具体实现细节上有所不同。 1. **数据准备与处理**: - 使用StringTie进行转录本组装和表达量估计,以`-e`参数对所有样本中都存在的转录本进行定量,输出为`.bg.gtf`文件。 - 创建一个ballgown目录,用于存放每个样本的转录本定量结果。 2. **提取转录本ID和FPKM值**: - 通过处理`.bg.gtf`文件,分别提取每个样本的转录本ID和对应的FPKM值。 3. **数据整合**: - 将转录本ID和FPKM值合并,生成每个样本的FPKM文件,然后对这些文件进行排序、合并,最终形成一个包含所有样本FPKM值的矩阵。 这个方法直接从转录本组装和定量的结果中提取FPKM值,依赖于StringTie的输出格式。 1. **计数数据生成**: - 使用featureCounts对BAM文件进行基因计数,得到原始的计数数据(`lncRNA_raw_count.txt`)。 2. **FPKM值计算**: - 在R中,首先读入featureCounts的输出,然后定义一个函数`countToFpkm`,将原始计数转换为FPKM值。 - 这个方法考虑了转录本的有效长度和总映射读数,手动计算FPKM值。 3. **数据过滤和输出**: - 对FPKM矩阵进行过滤,仅保留在至少一个样本中FPKM值大于0的转录本。 - 输出过滤后的转录本ID和对应的FPKM矩阵。 - **相同点**:两种方法最终都提供了转录本的FPKM值,用于量化基因表达水平。 - **不同点**: - **工具和流程**:第一种方法依赖于StringTie和Ballgown进行转录本的组装和表达量估计,而第二种方法使用featureCounts进行计数,然后在R中计算FPKM值。 - **数据处理**:第一种方法直接从转录本定量结果中提取FPKM值,第二种方法则从计数数据出发,使用自定义函数在R中计算FPKM值。 - **灵活性**:第二种方法在FPKM计算过程中提供了更多的自定义选项,例如可以调整有效长度等参数,而第一种方法的计算细节更多地依赖于StringTie的内部算法。 - **输出格式**:第一种方法的输出需要额外的步骤来整合和格式化数据,第二种方法则直接在R中进行数据处理和输出,可能对于熟悉R的用户来说更为方便。 总的来说,选择哪种方法取决于你的具体需求、数据类型以及个人对工具的熟悉程度。 两种方法都是用来计算FPKM(Fragments Per Kilobase of transcript per Million mapped reads)值,这是一种用来量化基因表达的方法。尽管目的相同,但它们在处理过程、工具使用和细节实现上有所不同。 - **过程**:这种方法首先使用StringTie进行转录本的组装和定量,然后通过Ballgown进行表达矩阵的构建。这个过程更多地依赖于基因组注释文件(如GTF文件)来指导转录本的重建和定量。 - **工具**:StringTie用于转录本的重建和定量,Ballgown用于表达数据的分析和可视化。 - **特点**:这种方法可以提供更细致的转录本水平的分析,能够识别和量化新的或已知的转录本。 - **过程**:这种方法直接使用featureCounts对比对后的BAM文件进行读数计数,然后通过R脚本计算FPKM值。这个过程侧重于已知转录本的定量。 - **工具**:featureCounts用于读数计数,R脚本用于后续的FPKM计算和数据处理。 - **特点**:这种方法相对简单直接,侧重于对已知基因或转录本的表达量进行定量分析。通过R脚本可以灵活地进行数据的进一步处理和分析。 - **目标**:两种方法的最终目的都是计算FPKM值,以量化基因或转录本的表达水平。 - **依赖数据**:两者都需要基因组注释信息和比对后的序列数据(BAM格式)。 - **工具和流程**:第一种方法依赖于StringTie和Ballgown这两个专门的生物信息学工具,而第二种方法主要依赖于featureCounts和R脚本进行数据处理和FPKM的计算。 - **数据处理深度**:第一种方法可能提供更深入的转录本级别的分析,包括新转录本的发现和注释,而第二种方法更多关注于对已知转录本的量化分析。 - **两种方法都是正确的**,选择哪一种取决于研究的具体需求、可用的工具、以及个人对工具的熟悉程度。对于需要深入转录本级别分析的研究,第一种方法可能更合适;而对于关注已知基因或转录本表达量的分析,第二种方法可能更直接和高效。
额外:stringtie -e与非-e gffread Sbicolor_454_v3.1.1.gene_exons.gff3.gz -T -o sbi311gx.gtf
#组装转录本:
#四样本合并,得到非冗余转录本集
#以stringtie_merge.gtf 为参考注释重新组装
Cuffcompare
筛选条件:
过滤,只保留exon>1并且长度>200bp的transcripts 1)FPKM>=0.5 2)coverage >1 3)Length > 200 4)分类为i j o u x
上面的代码是筛选lncRNA的,为什么stringtie -p 16 -G ~/sbi311gx.gtf -o CK18.gtf CK18.bam
没有-e,而下面的有-e,stringtie -e -B -p 16 -G ~/4_stringtie/stringtie_merge.gtf -o T18.gtf ~/T18.bam
这段代码描述的是使用StringTie和其他工具对RNA-Seq数据进行转录本组装、合并和筛选lncRNA的流程。解释-e参数在不同步骤中的使用与不使用的原因,我们需要了解StringTie的参数含义和这些步骤的目的。
StringTie -e参数 -e参数在StringTie中用于告诉程序仅使用提供的参考注释文件(GTF/GFF)中的已知转录本来估计表达量,而不尝试发现任何新的转录本。这通常用于更准确地量化已知转录本的表达,尤其是在后续分析步骤中需要精确的表达量估计时。
不同步骤中-e参数的使用
初步组装转录本(没有使用-e) :
在初步组装转录本的步骤中(如stringtie -p 16 -G ~/sbi311gx.gtf -o T18.gtf T18.bam),目的是从每个样本的RNA-Seq数据中尽可能地发现并组装转录本,包括新的和未注释的转录本。因此,不使用-e参数,以允许新转录本的发现。
合并转录本集(没有使用-e) :
在合并不同样本的转录本以生成非冗余转录本集的步骤中,同样不使用-e参数,因为目标是整合所有样本中发现的转录本,包括新的和已知的。
重新组装并量化(使用-e) :
在使用合并后的转录本集(stringtie_merge.gtf)作为参考来重新组装和量化每个样本的转录本的步骤中,使用了-e参数。这一步骤的目的是基于已经确定的转录本集(包括新发现的和已知的转录本)精确估计每个转录本的表达量。使用-e参数,因为此时关注的是对已识别转录本的精确量化,而不是发现新转录本。
总结
在初期的转录本组装和合并步骤中,目标是尽可能地发现和组装转录本,包括新的和未注释的,因此不使用-e参数。
在基于合并后的转录本集重新组装和量化表达量的步骤中,使用-e参数,因为此时的目标是精确量化已经识别的转录本集中的转录本,而不再是发现新的转录本。
featureCounts与stringtie featureCounts -T 8 -a /home/data1/Ghl/bam/lncRNA.gtf -o lncRNA_raw_count.txt -p -B -C -f -t transcript -g transcript_id /home/data1/Ghl/bam/*.short.bam
这个用stringtie可以吗?而且也可以生成的FPKM
featureCounts是一个专门用于计数RNA-Seq数据中的reads,映射到基因或转录本上的工具,它是Subread软件包的一部分。它主要用于定量阶段,用来统计对应于特定基因或转录本的reads数目。这个命令使用了多个参数来定制计数的行为,如-T指定线程数,-a指定注释文件,-o指定输出文件名,-p、-B、-C、-f等参数用于指定如何处理reads和如何匹配到注释等。
StringTie,另一方面,主要用于组装和量化基于RNA-Seq数据的转录本。尽管它与featureCounts在RNA-Seq数据分析中的作用不同,StringTie确实可以用于生成转录本的表达量估计,包括FPKM(每千基因组片段每百万基对映射reads数)或TPM(每百万转录本片段每千碱基映射reads数)。
若想用StringTie来代替featureCounts的功能,并获取FPKM值,你可以按照以下步骤进行:
使用StringTie进行转录本的组装和量化 :
首先,使用StringTie来组装每个样本的转录本,并生成每个转录本的表达量估计,包括FPKM。这一步是使用StringTie的基本功能,不需要featureCounts参与。
命令示例 :
1 stringtie -p 8 -G /home/data1/Ghl/bam/lncRNA.gtf -o output.gtf -A gene_abundance.txt -e -B /home/data1/Ghl/bam/sample.short.bam
-p 8:指定使用的线程数。-G /home/data1/Ghl/bam/lncRNA.gtf:指定注释文件。-o output.gtf:指定输出的GTF文件,其中包含组装的转录本。-A gene_abundance.txt:输出基因丰度估计文件,其中包括FPKM值。-e:仅估计提供的注释文件中的已知转录本的表达量。-B:生成可以被Ballgown读取的文件,用于后续的表达量分析。
通过这种方式,你可以使用StringTie来进行转录本的量化,并获取每个转录本的FPKM值,类似于featureCounts的功能,但是是通过不同的方法实现的。需要注意的是,StringTie提供的是转录本级别的表达量估计,而featureCounts提供的是基于注释的reads计数,两者提供的信息虽然相关,但是侧重点和使用的方法不同。
如果有多个bam文件,prepDE.py 如果你有很多.bam文件需要用StringTie进行转录本的组装和量化,你可以采用批处理的方式来处理它们。这通常涉及编写一个简单的脚本,该脚本遍历所有的.bam文件,并为每个文件运行StringTie命令。这种方法可以自动化处理大量样本,节省时间和减少重复劳动。
下面是一个基于Unix/Linux shell的简单脚本示例,演示如何自动化这个过程:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 #!/bin/bash GTF_PATH="/home/data1/Ghl/bam/lncRNA.gtf" OUTPUT_DIR="/path/to/output/directory" mkdir -p "${OUTPUT_DIR} " for BAM_FILE in /home/data1/Ghl/bam/*.bam; do BASE_NAME=$(basename "${BAM_FILE} " .bam) stringtie -p 8 -G "${GTF_PATH} " -o "${OUTPUT_DIR} /${BASE_NAME} .gtf" -A "${OUTPUT_DIR} /${BASE_NAME} _gene_abundance.txt" -e -B "${BAM_FILE} " done
这个脚本做了以下几件事:
定义了GTF文件的路径和输出目录的路径。
创建了输出目录(如果它还不存在的话)。
遍历指定目录下的所有.bam文件。
对每个.bam文件运行StringTie,组装转录本并量化表达量,输出结果到指定的输出目录。
在运行这个脚本之前,请确保你已经安装了StringTie,并且替换脚本中的GTF_PATH和OUTPUT_DIR为你自己的路径。你可能还需要根据你的系统环境和具体需求调整脚本中的其他参数。
要运行这个脚本,你需要将其保存到一个文件中,例如run_stringtie.sh,然后在终端中给予执行权限并执行它:
1 2 chmod +x run_stringtie.sh./run_stringtie.sh
这将自动处理指定目录下的所有.bam文件,并为每个文件生成相应的转录本组装和表达量估计文件。
如果你的目标是将多个.bam文件(每个代表一个样本)的表达量(如FPKM值)合并到一个大的文件中,你可以采取以下步骤来实现:
步骤1: 为每个.bam文件运行StringTie 首先,你需要为每个.bam文件运行StringTie来生成各自的表达量文件。这一步可以使用之前提到的批处理脚本来自动化完成。
步骤2: 使用StringTie的prepDE.py脚本合并表达量 StringTie附带了一个Python脚本prepDE.py,专门用于从多个样本的输出中提取并合并表达量数据(如FPKM或TPM),生成一个大的CSV或TSV文件,其中包含所有样本的表达量信息。
你可以按照以下步骤使用prepDE.py:
确保所有StringTie输出文件都在一个目录中 :确保你之前运行StringTie时指定的输出文件(包括每个样本的gene_abundance.txt文件)都在同一个目录下。
运行prepDE.py脚本 :
1 python prepDE.py -i <input_directory> -g <gene_count_matrix.csv> -t <transcript_count_matrix.csv>
<input_directory>是包含所有StringTie输出文件的目录。<gene_count_matrix.csv>是你希望生成的基因级别表达量矩阵的文件名。<transcript_count_matrix.csv>是你希望生成的转录本级别表达量矩阵的文件名。
这个脚本会扫描指定目录下的所有StringTie输出文件,并从中提取表达量数据,最终生成包含所有样本的表达量矩阵。这些矩阵可以是基于基因或转录本的,并且可以直接用于后续的表达量分析,如差异表达分析等。
注意
确保prepDE.py脚本的路径正确,或者你在其所在的目录下运行命令。
如果你没有prepDE.py脚本,可以从StringTie的GitHub仓库或安装目录中找到。
这种方法可以有效地处理大量样本,并将它们的表达量数据合并为单个文件,便于后续分析。
要将多个.bam文件(每个代表一个样本)的FPKM值合并到一个大文件中,你可以遵循以下步骤:
步骤1: 组装和量化每个样本的转录本
对每个.bam文件运行StringTie以组装转录本并计算FPKM值。这可以通过一个批处理脚本自动化完成,脚本需要遍历所有.bam文件并为每个文件执行StringTie命令。
示例命令:
1 stringtie -p 8 -G <path_to_annotation.gtf> -o <output_path>/<sample_name>.gtf -A <output_path>/<sample_name>_gene_abundance.txt -e -B <path_to_bam>/<sample_name>.bam
其中<path_to_annotation.gtf>是参考注释文件的路径,<output_path>是输出文件存放的目录,<sample_name>是样本名称,<path_to_bam>是.bam文件所在的目录。
步骤2: 合并所有样本的FPKM值
使用StringTie提供的prepDE.py脚本来从步骤1生成的所有样本的表达量文件中提取和合并FPKM值。
运行prepDE.py脚本的命令:
1 python prepDE.py -i <input_directory> -g <gene_count_matrix.csv> -t <transcript_count_matrix.csv>
<input_directory>是包含所有StringTie输出文件的目录。<gene_count_matrix.csv>是你希望生成的基因级别表达量矩阵的文件名(包含FPKM值)。<transcript_count_matrix.csv>是你希望生成的转录本级别表达量矩阵的文件名(如果需要)。
这个脚本将扫描指定目录下的所有StringTie输出文件,提取表达量数据,并生成包含所有样本的表达量矩阵文件。
注意
确保在执行这些步骤之前,你已经安装了StringTie和Python。
根据你的系统和具体需求,可能需要调整命令中的参数。
确保prepDE.py脚本的路径正确,或者你在其所在的目录下运行命令。
通过这些步骤,你将能够得到一个包含所有样本FPKM值的大文件,适用于进一步的表达量分析,如差异表达分析等。