安装教程
国内镜像在评论区
docker hub
docker 学习教程
https://docker.easydoc.net/doc/81170005/cCewZWoN/AWOEX9XM
https://docs.portainer.io/start/install/server/docker/wsl
https://docs.docker.com/desktop/install/windows-install/
一个例子,安装生信软件
想使用stringtie
在linux中stringtie
1. 基本用法
StringTie的基本命令格式如下:
stringtie [options]* <input.bam>
这里,<input.bam>是一个排序过的BAM文件,它包含了RNA-Seq数据的对齐信息。[options]是可选的命令行参数,用于控制StringTie的运行方式和输出。
2. 重建转录本和估计表达量
为了重建转录本并估计它们的表达量,可以使用以下命令:
stringtie -p 8 -G <reference.gtf> -o <output.gtf> <input.bam>
-p 8:指定使用8个线程进行运算,以加快处理速度。
-G <reference.gtf>:指定参考基因组的注释文件。这个文件包含了已知基因和转录本的信息,有助于提高转录本重建的准确性。
-o <output.gtf>:指定输出文件的名称,其中包含重建的转录本。
<input.bam>:输入的BAM文件,包含了RNA-Seq数据的对齐信息。
3. 查看帮助信息
如果您需要查看StringTie的详细命令行选项,可以运行:
stringtie –help
4 ,示例
假设有一个文件reads.bam(已排序的BAM文件),并且您有一个参考注释文件genome.gtf,以下是一个运行StringTie的示例命令:
stringtie -p 4 -G genome.gtf -o assembled_transcripts.gtf reads.bam
这个命令会使用4个核心,基于参考注释genome.gtf来处理reads.bam文件,并将重建的转录本保存到assembled_transcripts.gtf文件中。
注意
确保您的BAM文件已经根据参考基因组排序和索引。
根据您的服务器或个人计算机的性能,您可以调整使用的线程数(-p参数)来优化运行时间。
StringTie的输出(GTF文件)可以用于后续的基因表达量分析,例如使用Ballgown、Cufflinks的Cuffdiff或其他工具进行差异表达分析。
确保查阅StringTie的官方文档或发行说明,以获取最新的功能更新和详细的使用指南。
5,我当时运行的
stringtie -e -B -p 16 -G mergelist.gtf -o /home/data1/Ghl/lab4046839/TSSR/TSRR6116749.gtf /home/data1/Ghl/bam/SRR6116749.short.bam
需要gtf文件,排序后的bam文件,我随便找一个。
G:\Gallus_gallus.GRCg6a.106.gtf\Gallus_gallus.GRCg6a.106.gtf
G:\lab4046839\Gallus39.short.bam
stringtie -e -B -p 16 -G C:\Users\Administrator\Desktop\docker学习\stringtie\Gallus_gallus.GRCg6a.106.gtf -o C:\Users\Administrator\Desktop\docker学习\stringtie\1.gtf C:\Users\Administrator\Desktop\docker学习\stringtie\Gallus39.short.bam
在docker上运行
1,安装
在这个库里搜索stringtie
docker hub
https://hub.docker.com/
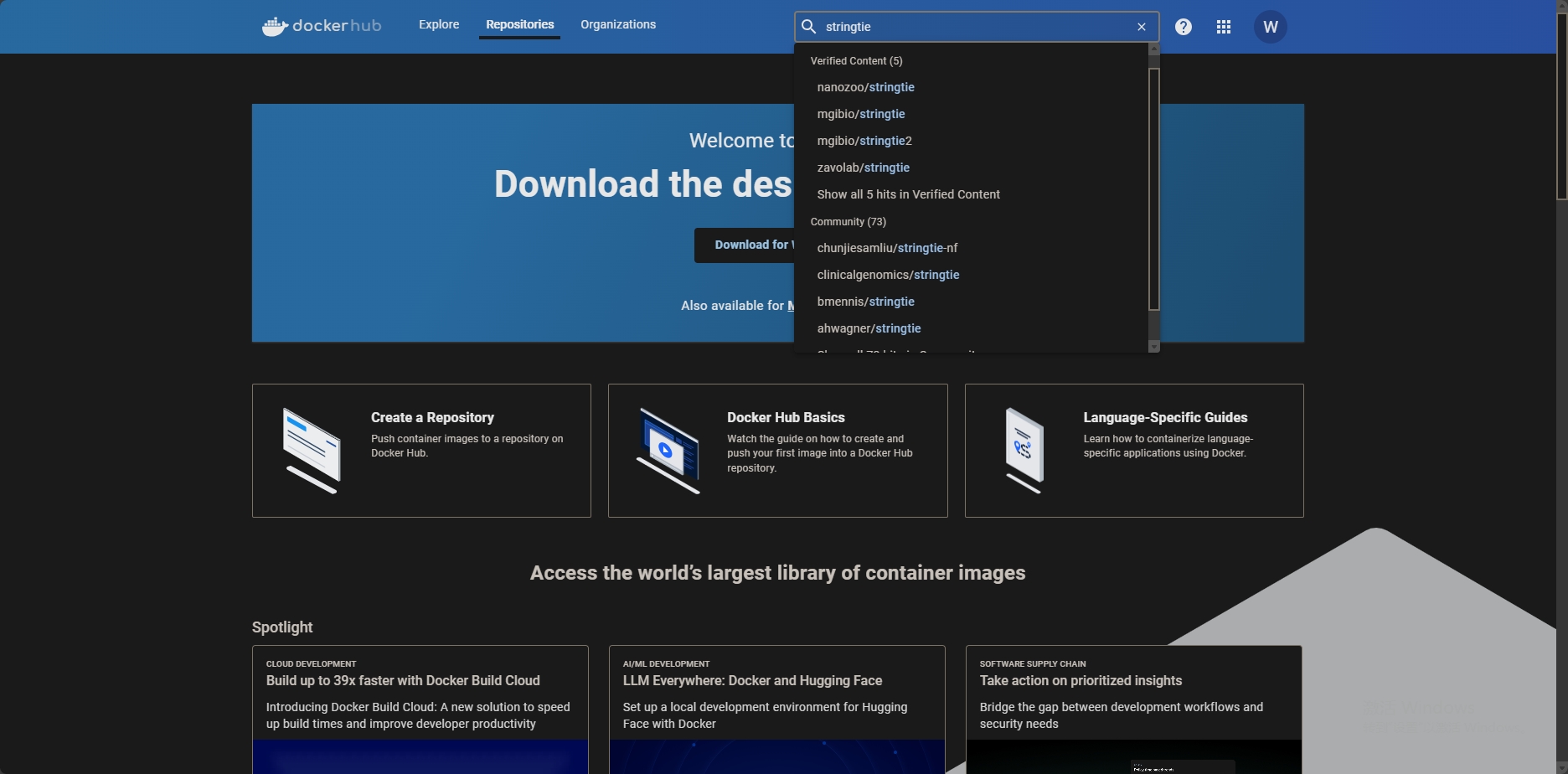
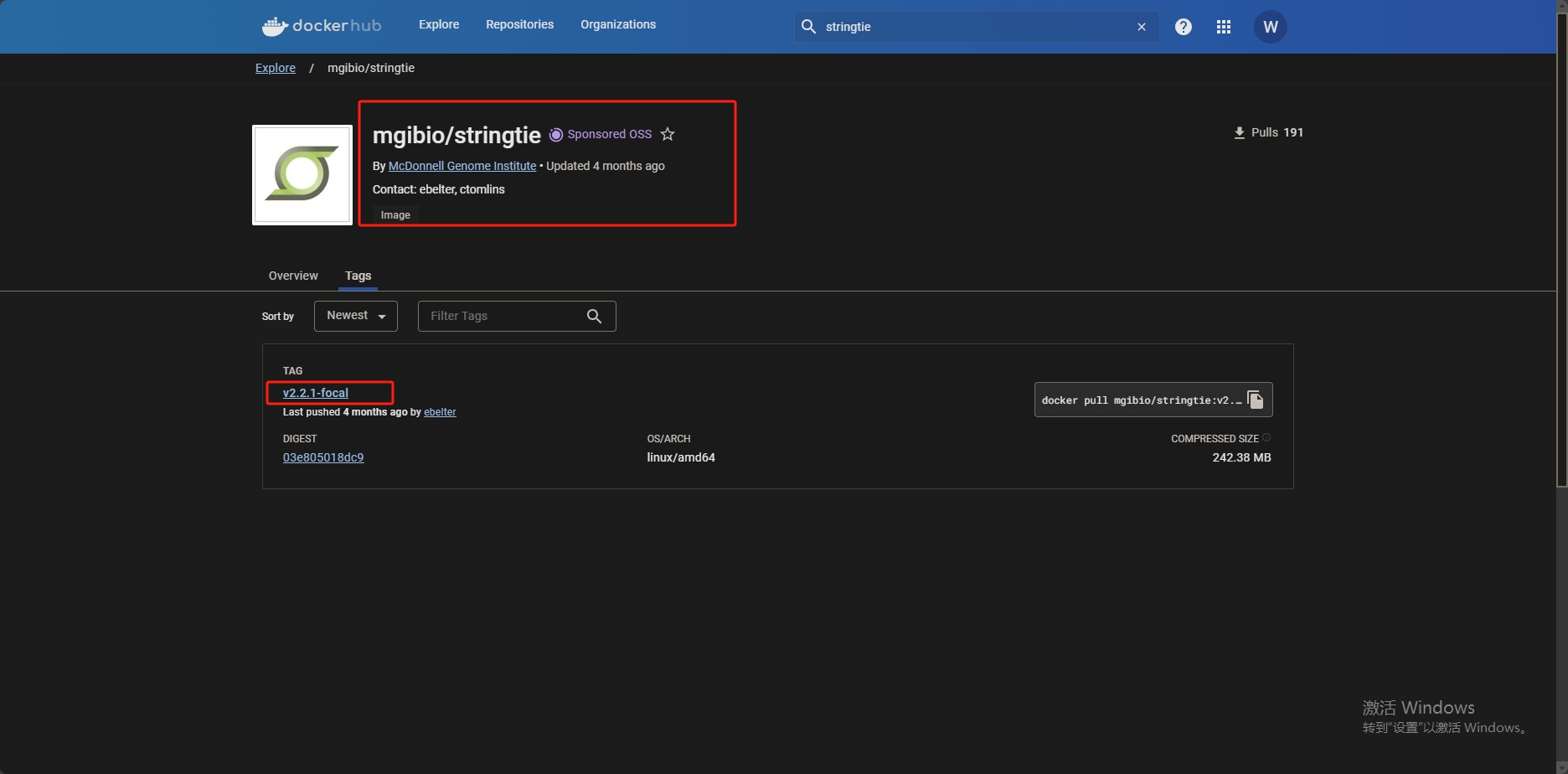
mgibio/stringtie确实是Docker Hub上的一个容器镜像,属于McDonnell Genome Institute。这个镜像是为了运行StringTie软件而设置的,StringTie是一个高效的RNA-seq读取组装和定量的工具,主要用于基因和转录本的重建和定量。
如何使用mgibio/stringtie镜像?拉取镜像
安装Docker:首先确保你的系统上安装了Docker。如果未安装,你需要先安装Docker。
拉取镜像:使用以下命令从Docker Hub拉取mgibio/stringtie的特定版本镜像,例如上图的v2.2.1-focal版本。
docker pull mgibio/stringtie:v2.2.1-focal
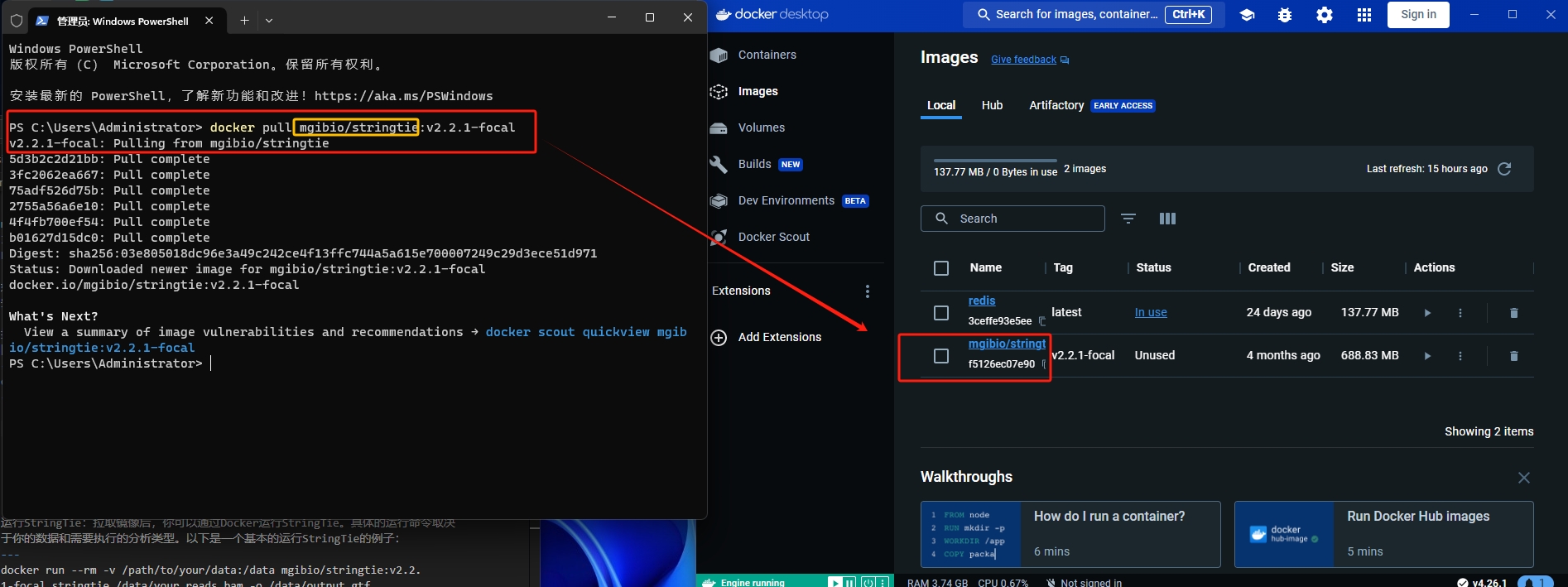
您已成功从Docker Hub上拉取了mgibio/stringtie镜像的版本v2.2.1-focal。StringTie是一个高效的RNA-Seq读取组装和表达量估计的工具,通常用于基因组注释和表达量的定量分析。
接下来,您可以通过以下步骤使用StringTie:
1,运行StringTie容器:
使用Docker运行StringTie的命令格式如下,其中[options]是您根据需要传递给StringTie的选项,例如输入文件、输出文件等:
docker run –rm -v $(pwd):/data mgibio/stringtie:v2.2.1-focal [options]
–rm:运行结束后自动删除容器。
-v $(pwd):/data:将当前目录挂载到容器的/data目录。这样做是为了让容器能够访问宿主机的文件系统中的文件,并且任何输出文件也将被写入到宿主机的当前目录。根据您的工作目录,您可能需要调整$(pwd)部分。
2,指定输入文件和运行参数:
StringTie需要RNA-Seq对齐文件(如BAM或SAM格式)作为输入。假设您的对齐文件位于当前目录,您可以这样运行StringTie:
docker run –rm -v $(pwd):/data mgibio/stringtie:v2.2.1-focal stringtie /data/your_alignment_file.bam -o /data/output.gtf
这里,your_alignment_file.bam是您的输入文件名,而output.gtf是您希望生成的输出文件名。
运行StringTie:拉取镜像后,你可以通过Docker运行StringTie。具体的运行命令取决于你的数据和需要执行的分析类型。以下是一个基本的运行StringTie的例子:
3,查看帮助和选项:
如果您需要查看StringTie支持的所有命令和选项,可以运行以下命令来获取帮助信息:
docker run –rm mgibio/stringtie:v2.2.1-focal stringtie –help
通过这些步骤,您可以开始使用StringTie对RNA-Seq数据进行处理和分析。根据您具体的分析需求,StringTie支持多种高级功能,包括但不限于基因和转录本的重建、表达量估计和差异表达分析。您可能需要根据StringTie的文档来调整命令行选项,以适应您的特定研究目的
docker run –rm -v /path/to/your/data:/data mgibio/stringtie:v2.2.1-focal stringtie /data/your_reads.bam -o /data/output.gtf
这个命令:
–rm:运行完后自动删除容器。
-v /path/to/your/data:/data:挂载卷,将你本地的数据目录(/path/to/your/data)映射到容器内的/data目录,以便StringTie可以访问你的BAM文件和输出结果。
mgibio/stringtie:v2.2.1-focal:指定要运行的Docker镜像。
stringtie /data/your_reads.bam -o /data/output.gtf:StringTie的命令行,使用/data/your_reads.bam作为输入文件,输出结果到/data/output.gtf。
请替换/path/to/your/data, your_reads.bam, 和output.gtf为你的实际文件路径和文件名。
注意事项
数据卷挂载:确保正确映射你的数据目录到容器内部,以便StringTie可以访问到输入文件,并能够将输出文件保存到你的主机上。
参数调整:根据你的具体需求,可能需要调整StringTie的命令行参数。查阅StringTie的官方文档以了解所有可用的命令和参数。
使用Docker运行StringTie提供了一个简单且一致的方式来部署和运行软件,无需担心依赖问题。
实际运行
docker run –rm -v “C:/Users/Administrator/Desktop/docker学习/stringtie:/data” mgibio/stringtie:v2.2.1-focal stringtie -e -B -p 16 -G /data/Gallus_gallus.GRCg6a.98.gtf -o /data/1.gtf /data/Gallus40.short.bam
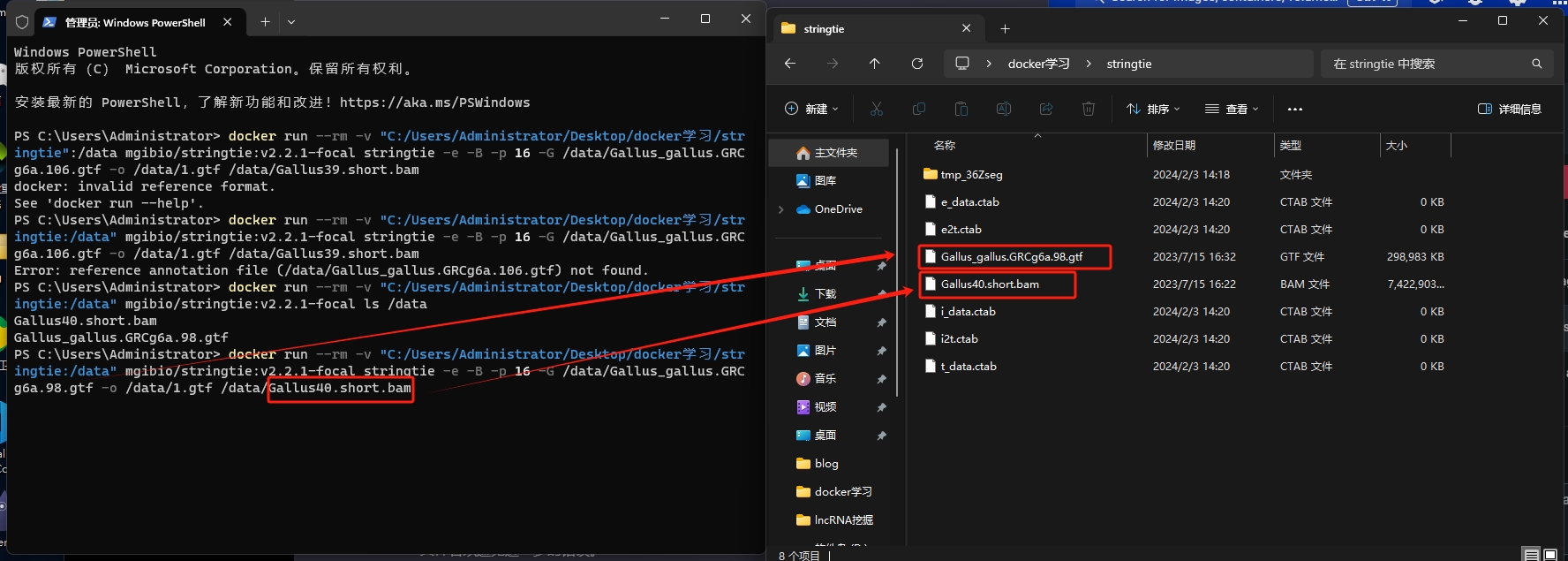
另外:docker run –rm -v “C:/Users/Administrator/Desktop/docker学习/stringtie:/data” mgibio/stringtie:v2.2.1-focal ls /data
这个命令将会列出挂载到容器/data目录下的所有文件,以确保挂载成功并且文件确实存在。
最后,是能运行的,这样看,确实很方便。搜索,安装,运行就可以了。



